CAM/gradCAM과 다르게 LIME/SHAP는 모델과 독립적으로 존재하는 모델이다. 모델만 있으면 LIME/SHAP을 추가할 수 있다. LIME/SHAP은 local한 요소를 강조한다.

정답인 경우 정답이라고 인식하게 된 부위를 붉은색으로 표시한다. 틀리게 나온 경우 어디를 보고 잘못 말한 건지 표시해준다. 이때 잘못 말한 경우를 보고 모델 튜닝도 가능하다. 예컨대 턱 부위를 보고 계속 잘못 판단한다면 그 부위를 특별히 조정할 수 있다.
(1) code
0. MLP MODEL
! pip install lime구글 코랩, 아나콘다에서 기본 지원이 안 된다.
import numpy as np
import matplotlib.pyplot as plt
from skimage.color import gray2rgb, rgb2gray
from skimage.util import montage
from sklearn.datasets import fetch_olivetti_faces
faces = fetch_olivetti_faces()scikit-image 패키지를 사용해서 올리베티 얼굴 데이터를 로드하고 확인한다.
X_vec = np.stack([gray2rgb(iimg) for iimg in faces.data.reshape((-1, 64, 64))],0)라임은 이미지를 rgb채널로 해석하기 때문에 흑백사진의 경우 다음처럼 흑백사진(1채널)을 컬러(3채널)로 변경해야 한다. 이때 분석하는 이미지의 크기가 모두 같아야 하므로 64x64로 resize해주어야 한다.
index = 93
plt.imshow(X_vec[index], cmap='gray')
plt.title('{} index face'.format(index))
plt.axis('off')이미지 한 장을 그리는 코드이다.
def predict_proba(image):
return session.run(model_predict, feed_dict={preprocessed_image: image})tensorflow를 이용한 분류 모델을 LIME에서 사용할 수 있게 클래스 별로 확률을 구해야 한다. 클래스 별 확률은 keras에는 있지만, sklearn에는 없다.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X_vec, y_vec, train_size=0.70)train 데이터와 test 데이터를 7:3 비율로 나누어준다.
from sklearn.pipeline import Pipeline
from sklearn.neural_network import MLPClassifier
class PipeStep(object):
""" Wrapper for turning functions into pipeline transforms (no-fitting) """
def __init__(self, step_func):
self._step_func=step_func
def fit(self,*args):
return self
def transform(self,X):
return self._step_func(X)
makegray_step = PipeStep(lambda img_list: [rgb2gray(img) for img in img_list])
flatten_step = PipeStep(lambda img_list: [img.ravel() for img in img_list])
simple_pipeline = Pipeline([
('Make Gray', makegray_step),
('Flatten Image', flatten_step),
('Normalize', Normalizer()),
('MLP', MLPClassifier(
activation='relu',
hidden_layer_sizes=(400, 40),
random_state=1))
])MLP는 CNN처럼 위치 정보가 유지되지 않으므로 이미지 데이터를 처리하기 위해 위 함수처럼 이미지를 전처리하는 파이프라인을 생성한다.
이때 Normalizer()함수로 정규화를 하며 모델 성능을 높인다.
simple_pipeline.fit(X_train, y_train)학습 데이터를 MLP가 있는 파이프라인에 붓는 코드
def test_model(X_test, y_test):
pipe_pred_test = simple_pipeline.predict(X_test)
pipe_pred_prop = simple_pipeline.predict_proba(X_test)
from sklearn.metrics import classification_report
print(classification_report(y_true=y_test, y_pred = pipe_pred_test))
test_model(X_test, y_test)classification_report를 사용해서 모델 성능을 테스트하는 코드,
predict_proba으로 클래스 별 확률도 표시한다.
1. LIME
from lime import lime_image
from lime.wrappers.scikit_image import SegmentationAlgorithm패키지를 로드한다.
explainer = lime_image.LimeImageExplainer()라임 설명체를 제공한다. 데이터의 종류에 따라 사용하는 함수도 다르다.
segmenter = SegmentationAlgorithm(
'slic',
n_segments=100,
compactness=1,
sigma=1)이미지 분할 알고리즘, segmenter 알고리즘이다. 이미지의 경우 어느 부분을 보고 정답을 예측했는지 보여주기 위해 해당 알고리즘이 중요한 역할을 한다.
라임은 기본적으로 4개의 알고리즘이 있으며, 해당 코드에서 사용한 알고리즘은 slic 알고리즘이다.
n_segmnets: 몇 조각으로 나눌 것인가compactness: 유사한 픽셀을 몇 개씩 합할 것인가
%%time
olivetti_test_index = 0
exp = explainer.explain_instance(
X_test[olivetti_test_index],
classifier_fn = simple_pipeline.predict_proba,
top_labels=6,
num_samples=1000,
segmentation_fn=segmenter)테스트 0번 이미지에 대해 설명 모델을 구축하는 코드이다. 인자 중 2개는 반드시 적어주어야 한다.
X_test[olivetti_test_index]
x_test만 아니라 index를 적어주어 확인해보고 싶은 이미지를 넣어주어야 한다.
classifier_fn = simple_pipeline.predict_proba
(2) code
model
train = keras.utils.image_dataset_from_directory("/content/drive/MyDrive/MyLecture/2022/ML2022/Pneumonia/chest_xray/train")
test = keras.utils.image_dataset_from_directory("/content/drive/MyDrive/MyLecture/2022/ML2022/Pneumonia/chest_xray/test")
val = keras.utils.image_dataset_from_directory("/content/drive/MyDrive/MyLecture/2022/ML2022/Pneumonia/chest_xray/val")디렉토리의 데이터를 불러와 자동으로 레이블을 붙인다.
BatchDataset element_spec=(TensorSpec(shape=(None, 256, 256, 3), dtype=tf.float32, name=None), TensorSpec(shape=(None,), dtype=tf.int32, name=None))
tensor 형태로 TensorSpec은 이미지를 불러오고, dtype는 레이블에 해당하는 값을 가져온다. 그래서 배열처럼 사용이 어렵다.
이때 image_dataset_from_directory의 API는 당음과 같다. default로 batch_size가 32개로 설정되어 있다.
tf.keras.utils.image_dataset_from_directory(
directory,
labels='inferred',
label_mode='int',
class_names=None,
color_mode='rgb',
batch_size=32,
image_size=(256, 256),
shuffle=True,
seed=None,
validation_split=None,
subset=None,
interpolation='bilinear',
follow_links=False,
crop_to_aspect_ratio=False,
**kwargs
)batch = train.as_numpy_iterator().next()TensorSpec으로 읽었지만 학습 시킬 때 numpy 배열 형태로 삽입해야 한다.
fig, ax = plt.subplots(3, 5, figsize=(15,10))
ax = ax.flatten()
for idx, img in enumerate(batch[0][:15]):
ax[idx].imshow(img.astype(int))
ax[idx].title.set_text(batch[1][idx])0번째는 이미지, 1번째는 레이블이 있기 때문에, idx는 이미지, img은 title.set_text의 값이 찍힌다.
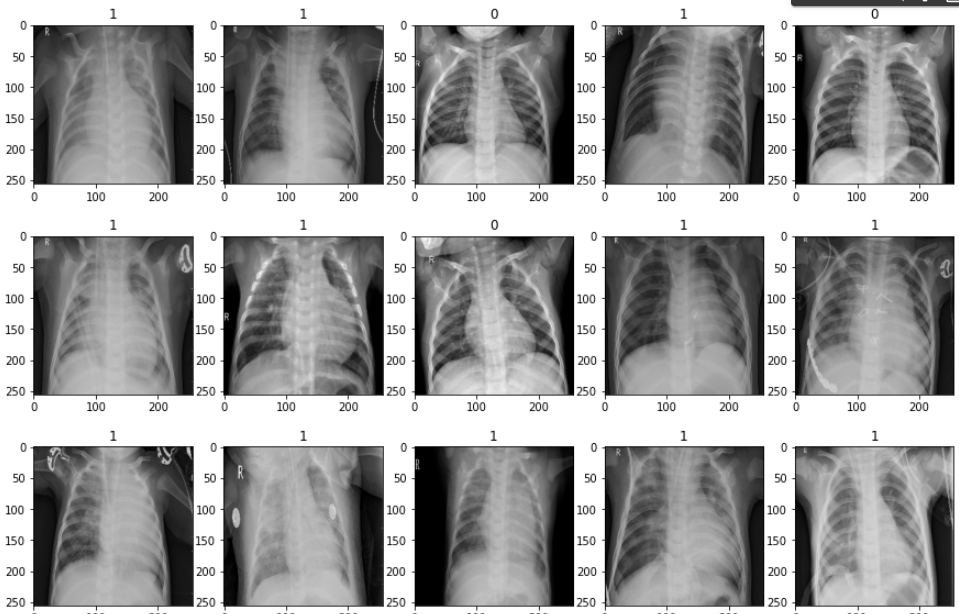
btach[0]가 이미지를 뿌리고, btach[1]는 y해당하며 idx만큼 16개를 설정한다.
train = train.map(lambda x, y:(x/255, y))
test = test.map(lambda x, y:(x/255, y))
val = val.map(lambda x, y:(x/255, y))정규화할 때 map함수 형식에 맞게 설정해야 한다.
history = model.fit(test, epochs=5, validation_data=val)모델 학습시킬 때 x_train과 y_train을 나누어 model.fit에 학습시켰지만 directory로 진행하는 경우 test 파일 통째로 집어넣는다.
bin_acc = BinaryAccuracy()
recall = Recall()
precision = Precision()
for batch in test.as_numpy_iterator():
X, y = batch
yhat = model.predict(X)
bin_acc.update_state(y, yhat)
recall.update_state(y, yhat)
precision.update_state(y, yhat)
print("Accuracy:", bin_acc.result().numpy(), "\nRecall:", recall.result().numpy(), "\nPrecision:", precision.result().numpy())출력층을 sigmoid 함수로 설정했기 때문에 classification_report를 사용할 때 0,1로 변환하여 사용해야 한다.
y_pred=np.where(yhat>0.5,1,0)
print(y_pred)다진 분류의 경우 np.argument함수를 쓴다.
LIME
from lime import lime_image
from lime.wrappers.scikit_image import SegmentationAlgorithm
explainer = lime_image.LimeImageExplainer()라임의 imageExplainer 생성한다.
# 이미지를 슈퍼픽셀로 분할하는 알고리즘 설정
# quickshift, slic, felzenswalb 등이 존재
segmenter = SegmentationAlgorithm('slic',
n_segments=100,
compactnes=1,
sigma=1) # 스무딩 역할: 0과 1사이의 floatSegmentationAlgorithm 생성한다.
n_segments: 이미지 분할 조각 개수compactnes: 유사한 파트를 합치는 함수, 보통 1이다.
%matplotlib inline
# 테스트셋의 18 번째 데이터 이용
test_idx = 2
exp = explainer.explain_instance(X[test_idx],
classifier_fn=model.predict,
top_labels=5,
num_samples=1000,
segmentation_fn=segmenter)top_labels: label 개수
from skimage.color import label2rgb
# 캔버스
fig, ((ax1, ax2), (ax3, ax4)) = plt.subplots(2, 2, figsize=(8, 8))
ax = [ax1, ax2, ax3, ax4]
for i in ax:
i.grid(False)
# 예측에 가장 도움되는 세그먼트만 출력
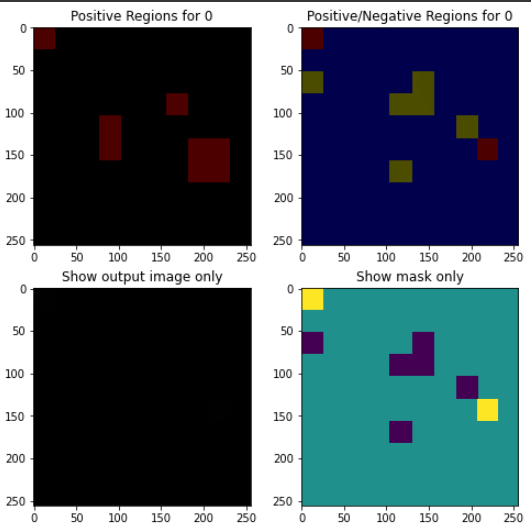
temp, mask = exp.get_image_and_mask(y[0],
positive_only=True, # 설명 모델이 결과값을 가장 잘 설명하는 이미지 영역만 출력
num_features=8, # 분할 영역의 크기
hide_rest=False) # 이미지를 분류하는 데 도움이 되는 서브모듈 외의 모듈도 출력
# label2rgb : 형광색 마스킹
ax1.imshow(label2rgb(mask, temp, bg_label=0), interpolation='nearest')
ax1.set_title('Positive Regions for {}'.format(y[0]))
# 모든 세그먼트 출력
temp, mask = exp.get_image_and_mask(y[2],
positive_only=False, # 설명 모델이 결과값을 가장 잘 설명하는 이미지 영역만 출력
num_features=8, # 분할 영역의 크기
hide_rest=False) # 이미지를 분류하는 데 도움이 되는 서브모듈 외의 모듈도 출력
ax2.imshow(label2rgb(4-mask, temp, bg_label=0), interpolation='nearest') # 역변환
ax2.set_title('Positive/Negative Regions for {}'.format(y[2]))
# 이미지만 출력
ax3.imshow(temp, interpolation='nearest')
ax3.set_title('Show output image only')
# 마스크만 출력
ax4.imshow(mask, interpolation='nearest') # 정수형 array
ax4.set_title('Show mask only')positive_only: 0이라고 예측하는 데 좋은 영향을 끼친 변수만 보여준다.- 못 맞혔는데 가장 영향을 많이 끼치는 변수가 문제점이다.