이진 논리회귀 실습 - 타이타닉 생존자 예측하기
1) 데이터 다운받기
import os
os.environ['KAGGLE_USERNAME'] = 'hyeonwoongjang' # username
os.environ['KAGGLE_KEY'] = '...' # key!kaggle datasets download -d heptapod/titanic
!unzip titanic.zip2) 필요한 패키지 임포트하기
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
from tensorflow.keras.optimizers import Adam, SGD
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler-
tensorflow로 논리 회귀 구현
-
from tensorflow.keras.models import Sequential : TensorFlow 라이브러리로부터
신경망 모델을 생성하기 위한 Sequential 모델을 가져옵니다. -
from tensorflow.keras.layers import Dense : 모델 내에
완전 연결 레이어(신경망 레이어)를 추가하기 위한 Dense 레이어를 가져옵니다. -
from tensorflow.keras.optimizers import Adam, SGD :
모델 학습에 사용할 최적화 알고리즘인 Adam과 SGD를 가져옵니다.
-
-
import numpy as np :
수치 계산을 위한 NumPy 라이브러리를 가져옵니다. -
import pandas as pd :
데이터 조작과 분석을 위한 Pandas 라이브러리를 가져옵니다. (데이터셋 csv 파일 읽기) -
import matplotlib.pyplot as plt :
데이터 시각화를 위한 Matplotlib 라이브러리를 가져옵니다. (그래프 그리기) -
import seaborn as sns :
데이터 시각화를 위한 Seaborn 라이브러리를 가져옵니다. (그래프 그리기) -
from sklearn.model_selection import train_test_split :
데이터를 Training Set과 Validation Set로 분할하는데 사용하는 train_test_split 함수를 가져옵니다. -
from sklearn.preprocessing import StandardScaler:
데이터를 표준화하는데 사용하는 StandardScaler를 가져옵니다. (전처리)
3) 데이터 로딩하기
df = pd.read_csv('train_and_test2.csv')- Pandas의 read_csv 함수를 사용하여 'train_and_test2.csv'라는 CSV 파일을 데이터프레임으로 불러옵니다.
df.head(5)- 데이터의 구조와 내용을 간단하게 확인하기 위해 데이터프레임 df의 처음 5개 행을 출력합니다.
4) 전처리하기 (데이터 정제)
A. 사용하는 칼럼(열)만 추출하기
df = pd.read_csv('train_and_test2.csv', usecols=[ # use_columns
'Age', # 나이
'Fare', # 승차 요금
'Sex', # 성별
'sibsp', # 타이타닉에 탑승한 형제자매, 배우자의 수
'Parch', # 타이타니게 탑승한 부모, 자식의 수
'Pclass', # 티켓 등급 (1, 2, 3등석)
'Embarked', # 탑승국
'2urvived' # 생존 여부 (0: 사망, 1: 생존)
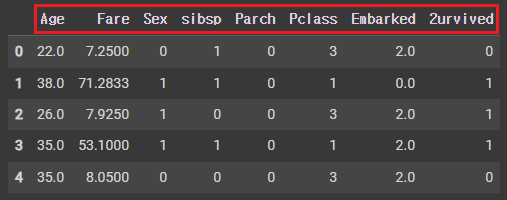
])
[ 간단하게 데이터셋 미리보기 ]
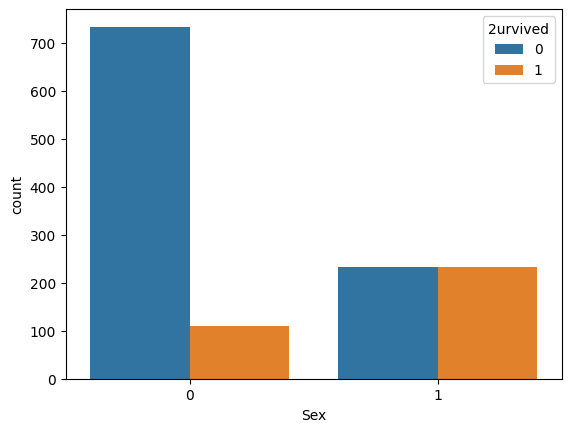
성별에 따른 생존자 수 : 각 성별 그룹 내에서 생존과 사망의 비율을 비교 : sns.countplot(x='Sex', hue='2urvived', data=df)
- sns.countplot(x='Sex', hue='2urvived', data=df) : Seaborn 라이브러리를 사용하여 카테고리형 데이터의 빈도를 시각화
- x='Sex': x축에 "Sex" 열의 데이터를 나타내는 막대 그래프를 생성하라는 명령
- hue='2urvived': 막대 그래프를 그릴 때 "2urvived" 열을 기준으로 데이터를 다르게 색칠하라는 명령
- data=df : 데이터를 df DataFrame에서 가져오라는 명령
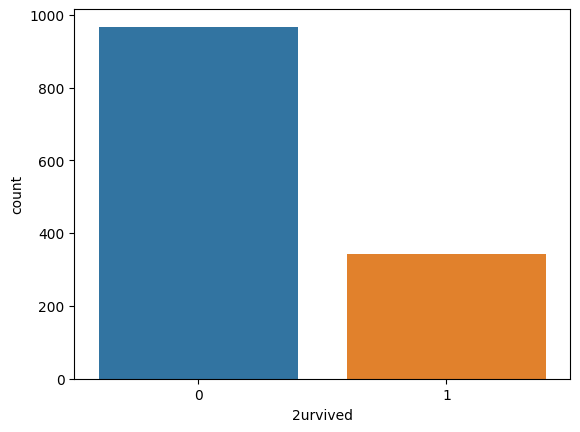
생존여부 클래스의 개수 확인 : sns.countplot(x=df['2urvived'])
- x=df['2urvived'] : x축에 "2urvived" 열의 데이터를 나타내는 막대 그래프를 생성하라는 명령
B. 비어있는 행 확인 & 없애기
- 비어있는 값(null 또는 na)이 있는지 확인 :
print(df.isnull().sum())- isnull() 메서드 : 각 열의 각 요소가 null값인지 확인
Age 0
Fare 0
Sex 0
sibsp 0
Parch 0
Pclass 0
Embarked 2 # 탑승지란이 비어있는 승객 두 명이 있음.
2urvived 0- 비어있는 값을 포함한 행을 제거 :
print(len(df)) # 1309
df = df.dropna() # DataFrame df에서 비어있는 값을 포함한 행을 제거한 새로운 DataFrame을 반환
print(len(df)) # 1307c. X, Y 데이터 분할
x_data = df.drop(columns=['2urvived'], axis=1) # '2urvived' 생존 여부 열을 Y 데이터로 지정하기 위해 X 데이터에서 제거
x_data = x_data.astype(np.float32) # x_data DataFrame 내의 데이터를 32비트 소수점 형식으로 변환 (32비트 형식으로 변환해야 tensorflow에서 Keras를 사용할 수 있음)
x_data.head(5) # 5개만 미리보기로 잘 적용되는지 확인 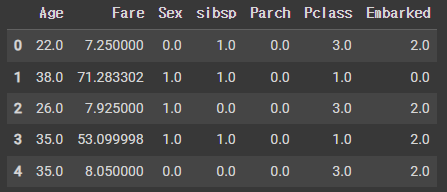
y_data = df[['2urvived']]
y_data = y_data.astype(np.float32)
y_data.head(5)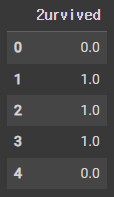
D. 표준화 (Standardization) : 평균을 0으로 맞추고 표준 편차를 1로 만들어 데이터 분포를 조절.
scaler = StandardScaler() # 데이터를 표준화(Standardization)하기 위한 StandardScaler 객체를 생성
x_data_scaled = scaler.fit_transform(x_data) # StandardScaler 객체를 사용하여 x_data DataFrame의 데이터를 표준화 : x_data의 데이터들의 단위가 다르기 때문
# 각 열의 데이터를 해당 열의 평균과 표준편차를 사용하여 조절 (데이터 스케일링)
print(x_data.values[0]) # before scaling : [22. 7.25 0. 1. 0. 3. 2. ]
print(x_data_scaled[0]) # after scaling : [-0.58026063 -0.5018386 -0.74189967 0.48027176 -0.44540733 0.8404475
0.6229398 ]E. 학습/검증 데이터 분할
x_train, x_val, y_train, y_val = train_test_split(x_data, y_data, test_size=0.2, random_state=2021) # 학습 세트 : 80% / 검증 세트 : 20% 으로 분할
print(x_train.shape, x_val.shape) # (1045, 7) (262, 7)
print(y_train.shape, y_val.shape) # (1045, 1) (262, 1)
# 1045개의 데이터로 학습을 시키고, 262개의 데이터로 모델의 성능을 검증 (tensorflow의 Keras를 사용하여 모델을 학습시킴) F. 모델 학습
model = Sequential([
Dense(1, activation='sigmoid') # 선형 회기를 거쳐 시그모이드 함수 사용 : 입력값을 0과 1 사이의 값으로 압축하여 모델의 출력값을 이진 클래스의 확률로 해석하기 위해서
])
model.compile(loss='binary_crossentropy', optimizer=Adam(learning_rate=0.01), metrics=['acc']) # Keras에서 0이냐 1이냐를 구분하는 이진 논리 회귀를 쓸 때, binary_crossentropy 손실 함수 사용
# metrics=['acc'] : 전체 샘플 중에서 모델이 올바르게 분류한 샘플의 비율을 계산하여 모델의 성능을 0에서 1사이로 측정합니다. 모델의 정확도가 높을수록(1에 가까울수록) 모델이 더 정확하게 분류합니다.
model.fit(
x_train,
y_train,
validation_data=(x_val, y_val), # 검증 데이터를 넣어주면 한 epoch이 끝날때마다 자동으로 검증
epochs=20 # epochs 복수형으로 쓰기!
)
#
Epoch 1/20
33/33 [==============================] - 1s 9ms/step - loss: 3.5066 - acc: 0.7177 - val_loss: 2.0245 - val_acc: 0.7481
Epoch 2/20
33/33 [==============================] - 0s 3ms/step - loss: 1.6454 - acc: 0.7187 - val_loss: 0.8011 - val_acc: 0.7748
...
33/33 [==============================] - 0s 3ms/step - loss: 0.4866 - acc: 0.7646 - val_loss: 0.4639 - val_acc: 0.7824
Epoch 20/20
33/33 [==============================] - 0s 3ms/step - loss: 0.4824 - acc: 0.7856 - val_loss: 0.4752 - val_acc: 0.7786
78%의 정확도로 생존 여부를 예측하는 모델이 됩니다.다항 논리회귀 실습 - 와인 종류 예측하기
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense
from tensorflow.keras.optimizers import Adam, SGD
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.preprocessing import OneHotEncoder
# 데이터셋 로드
df = pd.read_csv('Wine.csv')
df.head(5)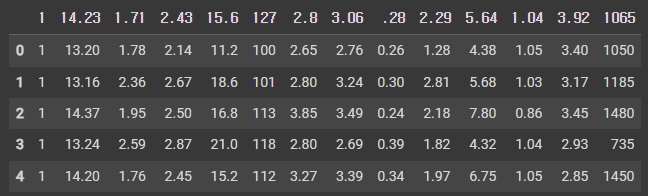
# 헤더의 내용을 채워주기 (보통은 다 세팅되어 있음.)
df = pd.read_csv('Wine.csv', names=[
'name' # 1, 2, 3으로 세 종류의 와인이 있음.
,'alcohol'
,'malicAcid'
,'ash'
,'ashalcalinity'
,'magnesium'
,'totalPhenols'
,'flavanoids'
,'nonFlavanoidPhenols'
,'proanthocyanins'
,'colorIntensity'
,'hue'
,'od280_od315'
,'proline'
])
df.head(5)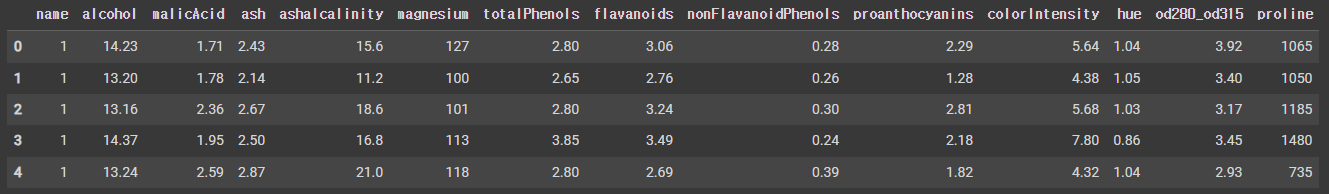
# 정답 라벨의 개수 확인
sns.countplot(x=df['name'])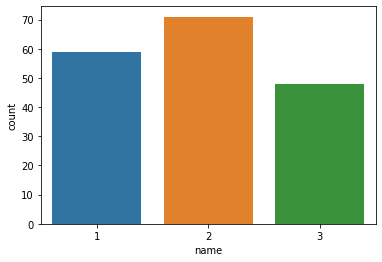
# 전처리 - 비어있는 행 확인
print(df.isnull().sum())
#
name 0
alcohol 0
malicAcid 0
ash 0
ashalcalinity 0
magnesium 0
totalPhenols 0
flavanoids 0
nonFlavanoidPhenols 0
proanthocyanins 0
colorIntensity 0
hue 0
od280_od315 0
proline 0
비어있는 행이 없다 = 좋은 데이터 (지워줄 데이터도 없음)
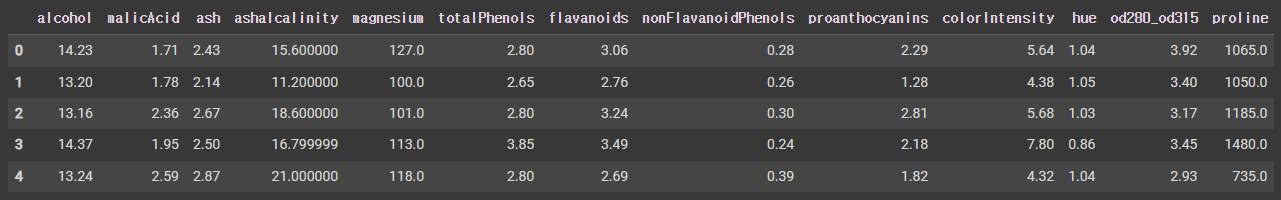
# X, y 데이터 분할
x_data = df.drop(columns=['name'], axis=1)
x_data = x_data.astype(np.float32)
x_data.head(5)
y_data = df[['name']]
y_data = y_data.astype(np.float32)
y_data.head(5)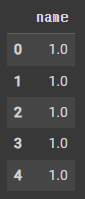
# 데이터 표준화
scaler = StandardScaler()
x_data_scaled = scaler.fit_transform(x_data)
print(x_data.values[0]) # 표준화 전 : [1.423e+01 1.710e+00 2.430e+00 1.560e+01 1.270e+02 2.800e+00 3.060e+00 2.800e-01 2.290e+00 5.640e+00 1.040e+00 3.920e+00 1.065e+03]
print(x_data_scaled[0]) # 표준화 후 : [ 1.5186119 -0.5622497 0.2320528 -1.1695931 1.9139051 0.8089973 1.0348189 -0.65956306 1.2248839 0.2517168 0.3621771 1.8479197 1.013009 ]
표준화된 데이터로 컴퓨터가 알아듣기 쉬워짐.
# One-hot encoding
encoder = OneHotEncoder()
y_data_encoded = encoder.fit_transform(y_data).toarray()
print(y_data.values[0]) # [1.]
print(y_data_encoded[0]) # [1. 0. 0.]
print(y_data_encoded[2]) # [0. 0. 1.]

# 학습/검증 데이터 분할
x_train, x_val, y_train, y_val = train_test_split(x_data_scaled, y_data_encoded, test_size=0.2, random_state=2021)
print(x_train.shape, x_val.shape) # (142, 13) (36, 13)
print(y_train.shape, y_val.shape) # (142, 3) (36, 3)
#
학습 데이터 : 142개 / 검증 데이터 : 36개 / 각 샘플 데이터가 가지는 특성 : 13개 / 각 샘플 데이터가 속하는 클래스 : 3개
# 모델 학습
model = Sequential([
Dense(3, activation='softmax')
]) # 이진 분류 문제에서는 하나의 출력 뉴런이 두 가지 결과 중 하나만 알아도 나머지를 예측하는 데 충분합니다.
# 출력 뉴런이 하나인 경우, 해당 뉴런의 출력 값은 0 또는 1로 해석됩니다.
# 예를 들어, 보통 0.5를 기준으로, 출력이 0.5보다 크면 하나의 클래스에 속한다고 판단하고, 0.5보다 작으면 다른 클래스에 속한다고 판단합니다.
# 다중 클래스 분류 문제에서는 3개의 클래스 중 하나를 선택하기 위해 3개의 출력 뉴런이 필요합니다.
model.compile(loss='categorical_crossentropy', optimizer=Adam(learning_rate=0.02), metrics=['acc'])
model.fit(
x_train,
y_train,
validation_data=(x_val, y_val), # 검증 데이터를 넣어주면 한 epoch이 끝날때마다 자동으로 검증
epochs=20 # epochs 복수형으로 쓰기!
)
#
Epoch 1/20
5/5 [==============================] - 1s 115ms/step - loss: 1.2892 - acc: 0.3798 - val_loss: 1.0719 - val_acc: 0.4444
Epoch 2/20
5/5 [==============================] - 0s 11ms/step - loss: 0.6915 - acc: 0.7560 - val_loss: 0.6363 - val_acc: 0.7222
...
12ms/step - loss: 0.0470 - acc: 1.0000 - val_loss: 0.1291 - val_acc: 0.9167
Epoch 20/20
5/5 [==============================] - 0s 12ms/step - loss: 0.0406 - acc: 1.0000 - val_loss: 0.1284 - val_acc: 0.9167
# 어떤 모르는 와인 데이터를 모델에 넣고 돌리면, 91.67%로 와인 1, 2, 3 중 어떤 와인에 속하는지 알 수 있는 모델을 만들었습니다.🥚🐣🐤🐥🐓🐔
