Devi GUI
2023 한양대학교 컴퓨터소프트웨어학부 졸업 프로젝트
Devi GUI
Devi GUI는 프로테오믹스의 MS2 데이터를 기반으로한 스펙트럼-펩타이드 매치 결과를 시각화하는 GUI입니다.
단백질은 우리 몸에서 효소, 호르몬, 항체 등의 다양한 역할을 하기 때문에 우리의 건강의 많은 영향을 줍니다. 따라서 단백질의 기능을 밝히기 위한 연구가 활발하게 진행되고 있는데, 단백질의 기능은 단백질의 구조에 달려있으며 단백질의 구조를 알기 위해서는 단백질의 아미노산 서열을 결정하는 과정이 필요합니다.
단백질의 서열을 결정하기 위한 방법으로는 질량분석(MS)기반의 방법이 쓰이는데, 바로 단백질을 아미노산 서열로 분해하여 질량분석기를 통해 질량을 측정하고 질량 분석기를 통해 얻은 정보를 이용해 기존에 밝혀진, 또는 가상의 아미노산 서열과 유사도를 분석하여 매치를 합니다. 질량분석기를 통해 얻은 정보를 '스펙트럼'이라고 하며, 스펙트럼을 아미노산 서열에 매치하는 방법은로는 기존에 실험을 통해 얻은(또는 AI를 통해 예측한) 스펙트럼과 매치하는 '스펙트럼 라이브러리 서치' 방식, 임의의 서열을 이론적인 스펙트럼 정보로 만들어 매치하는 '데이터베이스 서치' 방식이 있습니다.
Devi GUI는 스펙트럼 라이브러리 서치 엔진을 쉽게 실행하고, 그 매치 결과를 시각화하여 프로테오믹스 연구자들에게 편의를 제공하는 GUI입니다.
UI 및 사용 방법
View Spectra 탭

GUI에서 ‘View Spectra’ 탭([그림 2]의 화면)에서는 펩타이드-스펙트럼 각각의 매치를 시각화합니다.
상단 메뉴바에서 'Open > New Project'를 클릭하여 새로운 프로젝트를 실행할 수 있습니다. 인풋 다이얼로그에서 프로젝트 명과 각종 파라미터를 설명하고 쿼리 파일들(.mgf), 타켓 라이브러리 파일들(.msp), 디코이 라이브러리 파일들(.msp)을 임포트 한 후 실행하면 새로운 프로젝트가 생성되며 화면에 매치 결과가 시각화 됩니다.
프로테오믹스는 대용량의 단백체 데이터를 다루기 때문에 한번 매치 프로그램을 실행하는 데에는 상당한 시간이 소요될 수 있습니다. 따라서 Devi에는 이전에 한번 실행된 프로젝트는 간단히 프로젝트 파일을 불러오는 것으로 빠르게 결과를 시각화할 수 있는 기능을 제공합니다. 상단 메뉴바에 'Open > Open Project'를 클릭하고 프로젝트 파일(.devi)을 임포트하면 빠른 시간 내에 매치 결과를 볼 수 있습니다.
더 자세한 사용 설명은 Wiki를 참고 해주세요.
Summary 탭
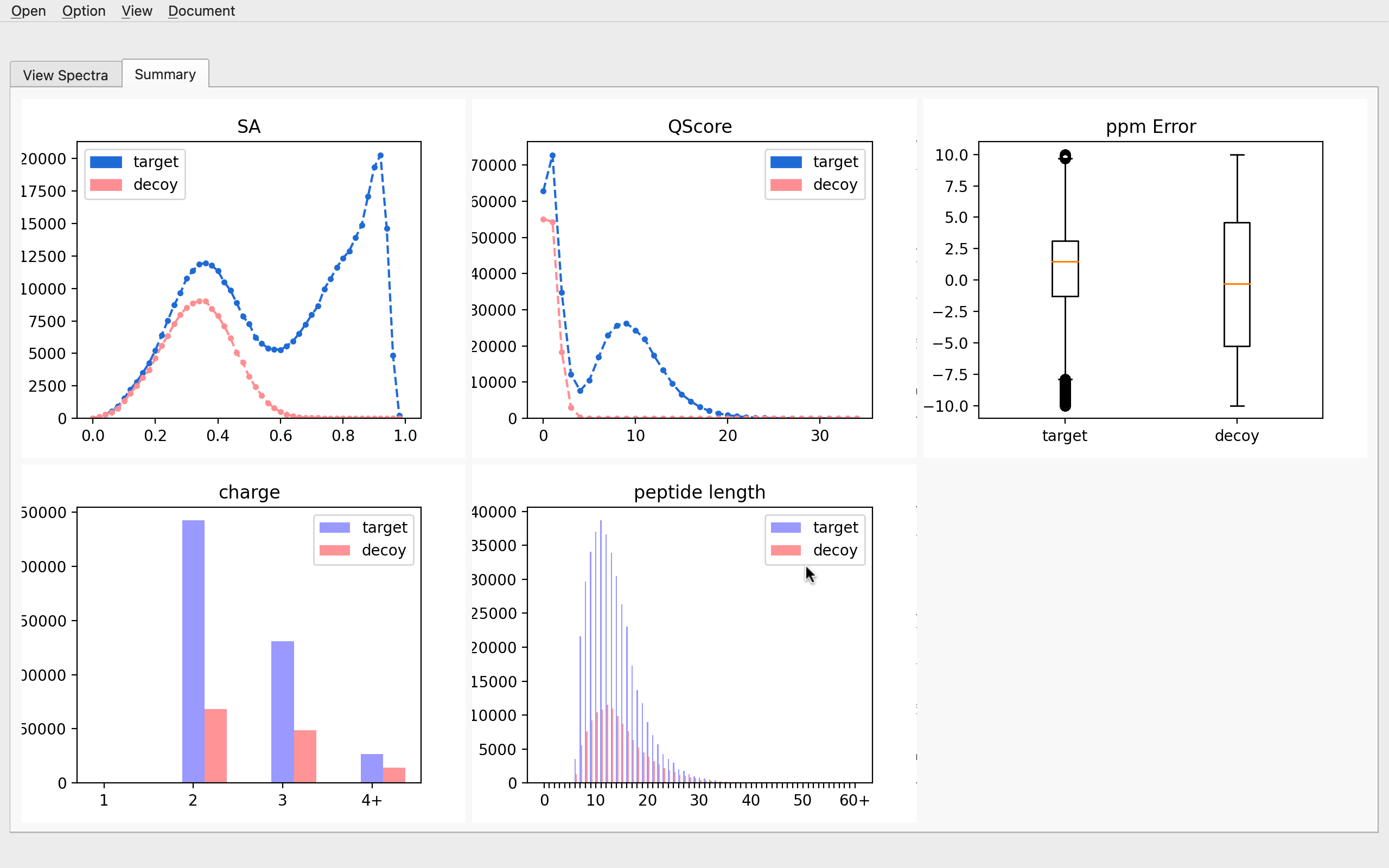
Summary 탭에서는 실행된 프로젝트의 매치 분포를 시각화하여 연구자가 매치 결과에 대한 전체적인 이해를 할 수 있도록 돕습니다.
쿼리 스펙트럼과 라이브러리 스펙트럼이 얼마나 유사한지를 나타내는 SA Score와 QSocre, 매치가 얼만큼의 에러를 가지는지를 나타내는 ppm Error, 매치된 스팩트럼의 전하의 개수와 매치된 펩타이드의 길이 분포를 시각화합니다.
Devi의 구현
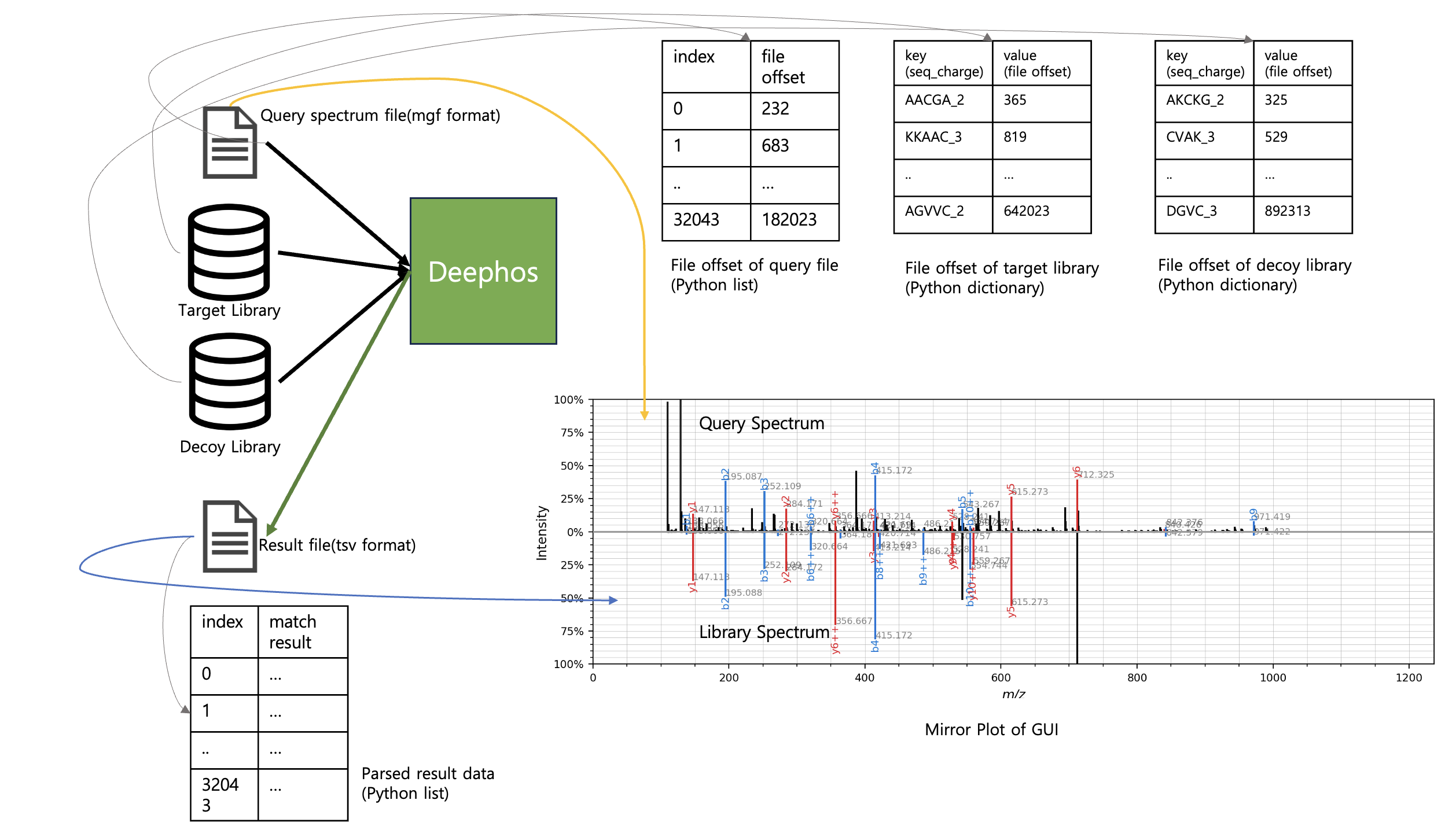
GUI를 통해 쿼리 스펙트럼 파일과 타겟-디코이 스펙트럼 라이브러리 파일을 선택한 후 펩타이드 오차 범위 등과 같은 각종 파라미터를 입력하여 서치 엔진을 실행합니다. 그러면 서치 엔진은 쿼리 스펙트럼과 라이브러리 스펙트럼의 매치 결과를 담고 있는 결과 파일을 생성합니다.
서치 엔진의 실행이 끝나면 쿼리 스펙트럼 파일, 결과 파일, 타겟 스펙트럼 라이브러리 파일, 디코이 스펙트럼 라이브러리 파일을 모두 전체 스캔하고 문자열을 처리하여 나온 정보를 파이썬 자료구조(딕셔너리, 리스트 등)에 저장합니다. 쿼리 스펙트럼 파일은 각각의 스펙트럼의 m/z-intensity 정보가 시작되는 위치의 파일 오프셋을 리스트에 저장합니다. 파일 오프셋을 저장하는 이유는 이후 GUI 스펙트럼을 시각화 할 때 필요한 m/z, intensity 정보를 그 때 그 때 쿼리 파일을 스캔하여 가져올 것이기 때문입니다. 결과 파일의 각 매치는 딕셔너리에 저장됩니다. 딕셔너리는 내부적으로 해시 맵으로 구현되어 있으므로 키-값의 형태로 저장이 되는데 결과 파일의 모든 항목에 대한 정보를 딕셔너리에 저장하므로, 키는 ‘File’, ‘Peptide’ 등과 같은 항목을 나타내는 문자열, 값은 그에 해당하는 값입니다. 이러한 각 매치에 대한 딕셔너리를 모두 리스트에 저장합니다. 타겟-디코이 스펙트럼 라이브러리는 각각 타겟과 디코이 정보를 저장하는 딕셔너리에 저장이 됩니다. 키는 ‘펩타이드 서열-전하량’ 문자열이며 값은 피크의 개수와 라이브러리 파일에서 m/z, intensity 정보가 시작되는 파일 오프셋입니다.
이렇게 정보들을 저장해 놓은 정보들은 GUI에 스펙트럼에 대한 각종 정보를 표시할 때 사용됩니다. 결과 파일의 정보를 저장한 파이썬 리스트를 순차 탐색하며 그 모든 정보를 GUI에 표 형식으로 표시합니다. 사용자가 표에서 특정 펩타이드-스펙트럼 매치를 클릭하면 쿼리 파일의 스펙트럼 정보 시작 파일 오프셋을 저장해 놓은 리스트에 접근해 파일 오프셋을 얻고, 쿼리 파일을 열어서 해당 오프셋으로 파일 포인터를 이동하여 선택된 스펙트럼의 정보를 모두 읽어 미러 플롯 상단에 시각화 합니다. 또한 타겟 또는 디코이 라이브러리 파일에서의 스펙트럼 정보 시작 파일 오프셋을 저장해 놓은 리스트에 접근해 파일 오프셋을 얻고, 라이브러리 파일을 열어서 앞에 설명한 과정과 동일하게 스펙트럼 정보를 읽고 미러 플롯 하단에 시각화 한다. 스펙트럼 정보의 시각화는 파이썬의 라이브러리인 ‘spectrum_utils’를 이용하였습니다.
전체 매치의 분포를 요약하는 페이지의 그래프는 파이썬 라이브러리인 ‘matplotlib’을 이용하였습니다.
Dependencies
Devi는 파이썬으로 개발되었습니다.
Devi를 개발하며 이용한 라이브러리는 다음과 같습니다.
Package Version
------------------------- -------
altgraph 0.17.4
appdirs 1.4.4
contourpy 1.1.0
cycler 0.11.0
fastobo 0.12.2
fonttools 4.41.1
importlib-metadata 6.8.0
importlib-resources 6.0.0
kiwisolver 1.4.4
lark 1.1.7
llvmlite 0.40.1
macholib 1.16.3
matplotlib 3.7.2
numba 0.57.1
numpy 1.24.4
packaging 23.1
pandas 2.0.3
Pillow 10.0.0
pip 23.2.1
pyinstaller 6.1.0
pyinstaller-hooks-contrib 2023.10
pyparsing 3.0.9
PyQt6 6.5.2
PyQt6-Qt6 6.5.2
PyQt6-sip 13.5.2
PySide6 6.5.2
PySide6-Addons 6.5.2
PySide6-Essentials 6.5.2
pyteomics 4.6
python-dateutil 2.8.2
pytz 2023.3
setuptools 58.0.4
shiboken6 6.5.2
six 1.16.0
spectrum-utils 0.4.2
tzdata 2023.3
zipp 3.16.2