오늘의 후기👍
우하하 velog 글씨색 넣는 법 배워왔어요
오늘은 어제보단 할만했습니다~!!!
팀원들과 함께 머리를 맞대면서 코드를 짜보니까 되게 얻는게 많았달까
전공자이신 팀원분이 리뷰하실 때 엄청나게 귀를 귀울여서 들었다
코드가 정말 다름.. 뭔가 간결하고.. 아무튼 멋지셨다...
우리 조는 비전공자가 대부분인 조였는데 그래서 더 다양한 궁금증과
해결방법이 나타난 느낌 완전 럭키비키⭐
그리고 주피터로.. 시험본다해서 해봤는데...
코랩처럼 막 괄호 한 쪽을 쓰면 반대편 괄호를 자동으로 닫아준다던지
그런 서비스가 없나요 혹시
너무 불친절해요 💢💢
Q3.winequality_red.csv를 판다스 데이터 프레임으로 불러와서 wine에 선언하는 코드를 작성하고 실행하시기 바랍니다.
wine = pd.read_csv(path+'winequality_red.csv', sep=';')
#판다스 설명서 read_csv누르면 나옴 sep의 defalt는 ',' (csv파일이 기본적으로 쉼표구분)
csv가 comma-seperate values라는 사실!
Q12. 데이터 프레임 wine의 컬럼 density와 pH를 표준화(standardization) 하시기 바랍니다
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler()
wine[['density', 'pH']]=scaler.fit_transform(wine[['density', 'pH']])
print(wine['density']) #1차원 시리즈
print(wine[['density']]) #데이터프레임
# wine['density'] = scaler.fit_transform(wine['density'])
#오류발생 (2차원 배열 또는 데이터프레임을 예상하는데, 시리즈 형태(1차원)가 들어왔기때문)
#scaler.fit_transform은 1차원 시리즈가 아닌 2차원 배열, 데이터프레임 형태여야
이 부분 챗gpt덕분에 이해했다.
Q17. RandomForest의 Tree 개수에 따른 accuracy를 시각화하시기 바랍니다.
#for문 연습해야겠다..
for i in range(len(accs)):
acc = accs[i]
# print(acc)
plt.plot(i, acc, marker='o')
plt.show()
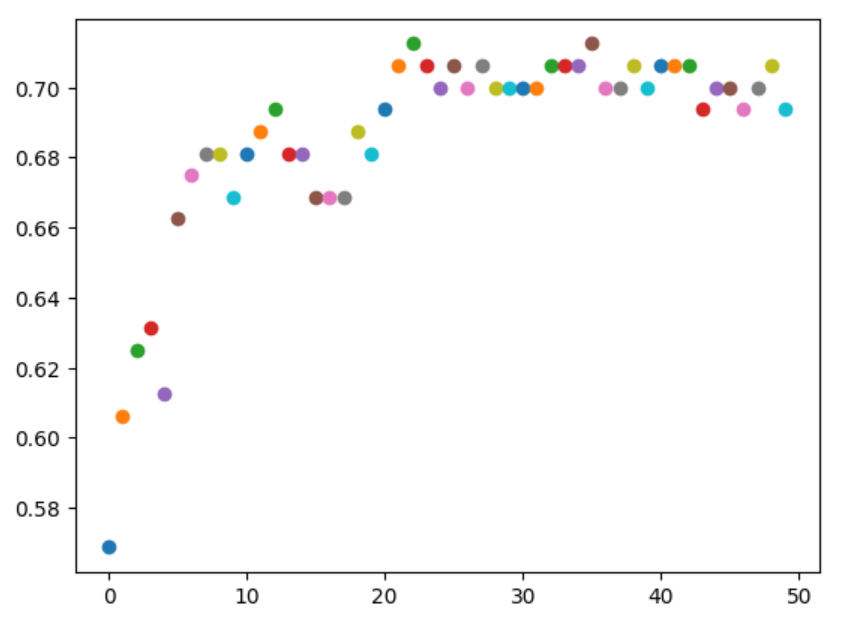
for문 연습해보려고 그래프도 for문으로 그려볼까 했는데
선그래프가 산점도처럼 나와버렸다..... 알록달록귀여움
plt.plot(range(1,51), accs, marker='o')
plt.show()
#그냥 이렇게 그리면된다!Q18. 아래 조건에 맞추어 뉴럴네트워크 모델을 학습시키시기 바랍니다.
model = Sequential([
Dense(64, activation='relu',input_shape=(x_train.shape[1],)),
Dense(32, activation='relu'),
Dense(10, activation='softmax')
])
model.compile(optimizer='adam', loss='sparse_categorical_crossentropy', metrics=['acc'])
es = EarlyStopping(monitor='val_loss', patience=50, restore_best_weights=True, verbose=1)
history = model.fit(x_train, y_train, epochs=2000, verbose=1,
batch_size = 1, validation_data=(x_test, y_test), callbacks=[es])
여기서 loss를 'sparse_categorical_crossentropy'가 아니라
'categorical_crossentropy'로 설정하면 오류가 발생했는데 그 이유는
y의 값이 정수형일때는 전자로 설정하고
만약 원-핫 인코딩을 했다면 후자로 설정해야하기때문이다
끝!