시험기간 다 끝난 이후에 다시 시작하는.. 나홀로 프로젝트.. 이제 진짜 얼마 안남았다.
프로젝트도 얼마 안남았는데(아마),, 마감일은 더 얼마 안남았다. 큰일났다. 후달달
나는 할 수 있다!!!!!!! 할 수 있다고!!!!!!!!!!
밤새고, 독서실에서 했다가, 집에서 했다가,, 날짜 개념이 사라진 탓에 그냥 17-19일은 통째로 작성합니당!
8장. PJ1_악성코드 탐지 모델(특징 공학)
아주 큰 결심을 하였다. 책에서 주는 가이드라인과 샘플 코드들을 통하여 프로젝트를 따라가고 있었는데, 계속 버전 문제와 내가 구한 샘플들과의 차이들 때문에 시간이 너무 지체되었다. 오류를 수정할수록 점점 의도한 코드와 동떨어져가는 느낌을 받아서, 그냥 내 방식대로 특징 공학을 하기로 하였다!
내가 구했던 특징들 pe, ngram, image 특징들을 사용하여 svm, randomforest, naivebayes, dnn, cnn, avg 모델에 각각 넣어 높은 정확도를 가진 아이를 선택할 것이다.
암튼!
시작해봅시다!
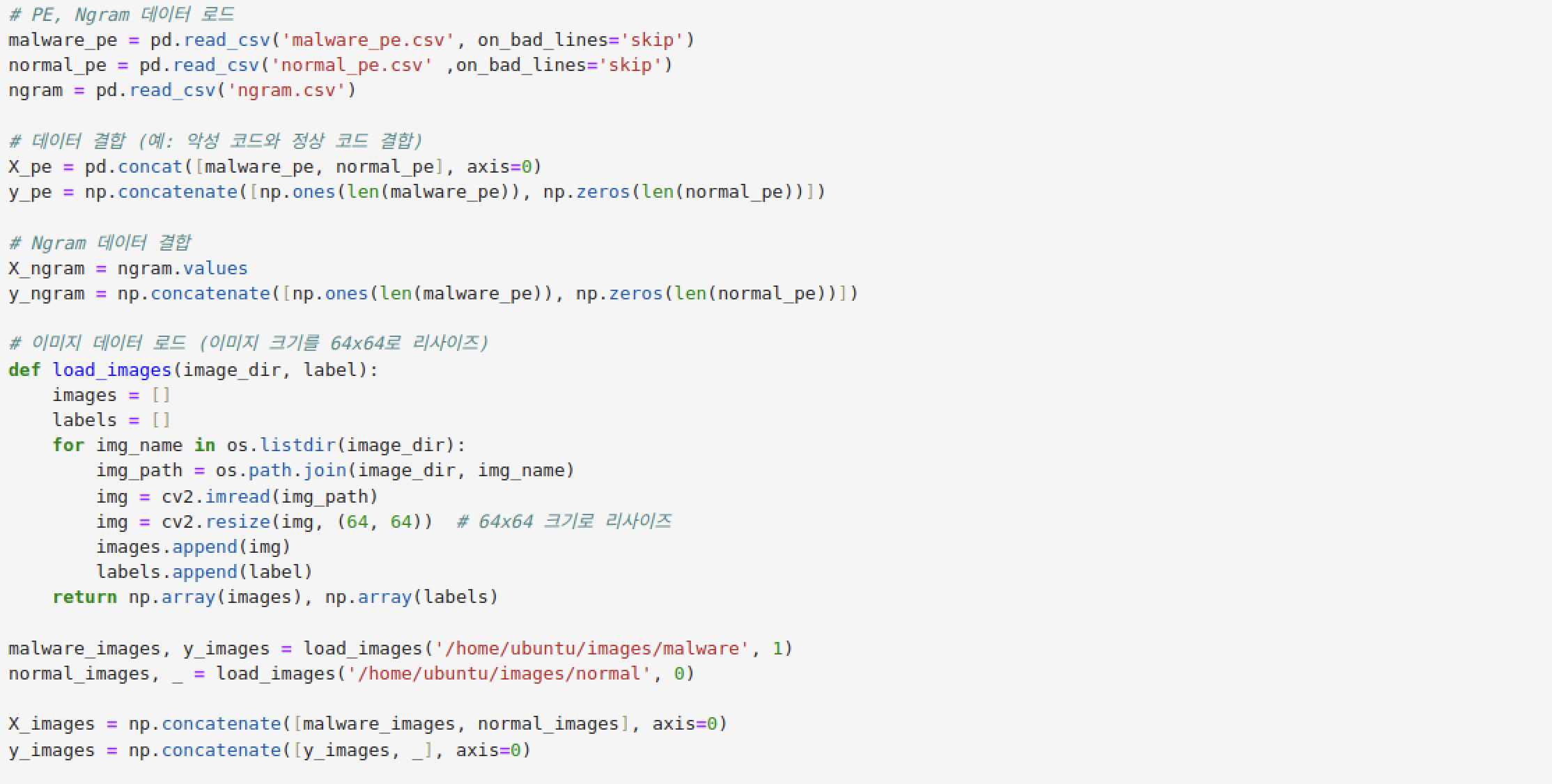
- PE, Ngram 데이터 로드: malware_pe.csv, normal_pe.csv, ngram.csv 파일을 읽어옵니다.
- PE 데이터셋 결합: 악성코드(malware_pe)와 정상(normal_pe) 데이터를 결합하여 X_pe를 만들고, 레이블(y_pe)을 생성합니다. 악성코드는 1, 정상은 0으로 레이블을 지정합니다.
- Ngram 데이터셋 준비: ngram.csv 데이터를 읽고, X_ngram에 값을 할당한 후, PE와 동일한 레이블을 y_ngram에 생성합니다.
- 이미지 데이터 로드: load_images 함수는 지정된 디렉토리에서 이미지를 64x64 크기로 읽고, 레이블을 할당하여 X_images와 y_images 배열을 만듭니다. 악성코드는 1, 정상은 0으로 레이블을 지정합니다.
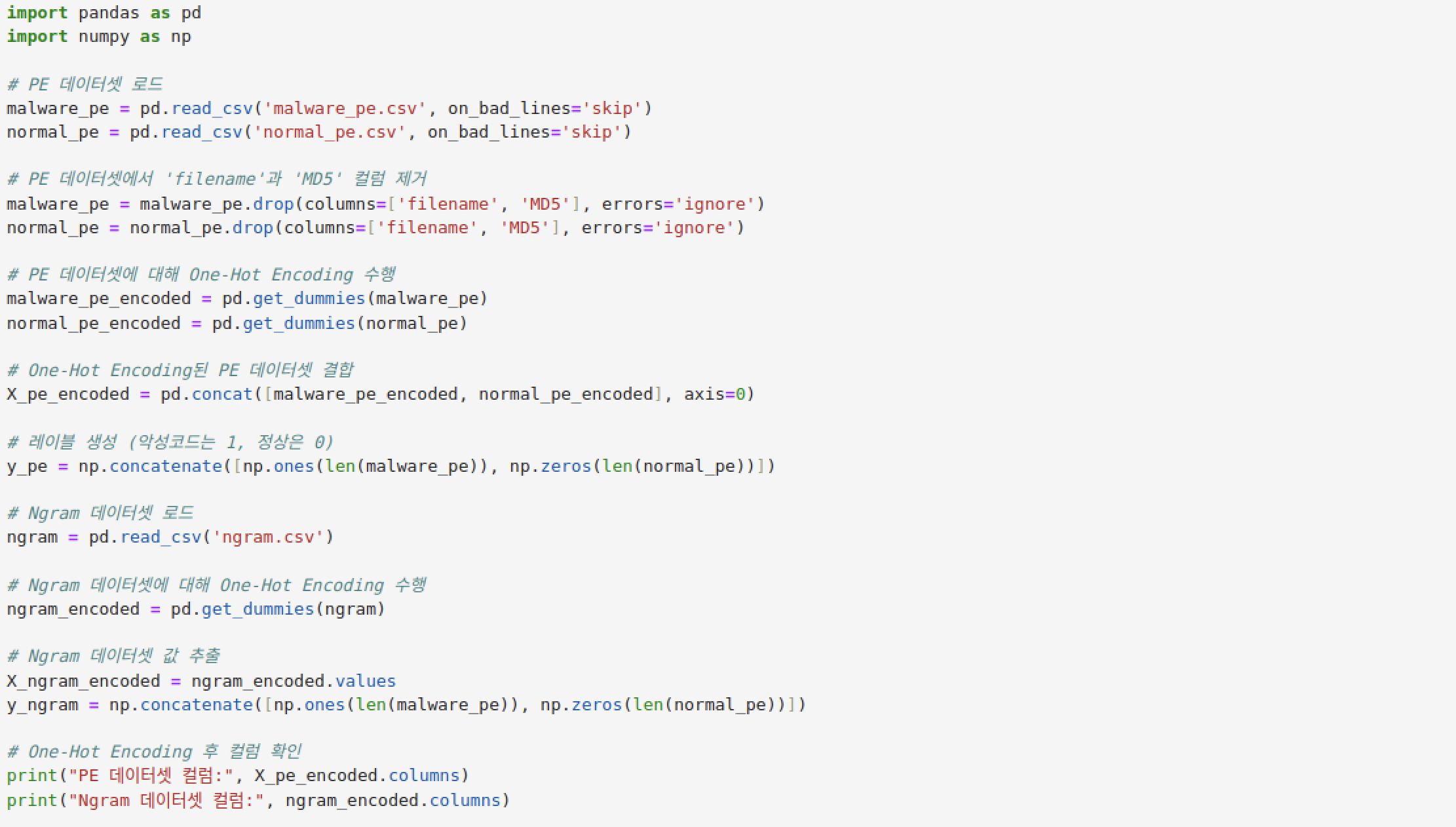
- PE 데이터 로드: malware_pe.csv와 normal_pe.csv 파일을 로드합니다.
- 불필요한 컬럼 제거: filename과 MD5 컬럼을 제거합니다.
- One-Hot Encoding: PE 데이터셋을 One-Hot Encoding하여 malware_pe_encoded와 normal_pe_encoded를 생성합니다.
- PE 데이터 결합: One-Hot Encoding된 malware_pe_encoded와 normal_pe_encoded를 결합하여 X_pe_encoded를 만듭니다.
- 레이블 생성: 악성코드는 1, 정상은 0으로 레이블을 생성하여 y_pe에 저장합니다.
- Ngram 데이터 로드 및 One-Hot Encoding: ngram.csv 데이터를 로드하고 One-Hot Encoding하여 ngram_encoded를 생성합니다.
- Ngram 데이터 준비: One-Hot Encoding된 ngram_encoded의 값을 X_ngram_encoded에 할당하고, 레이블을 y_ngram에 생성합니다.
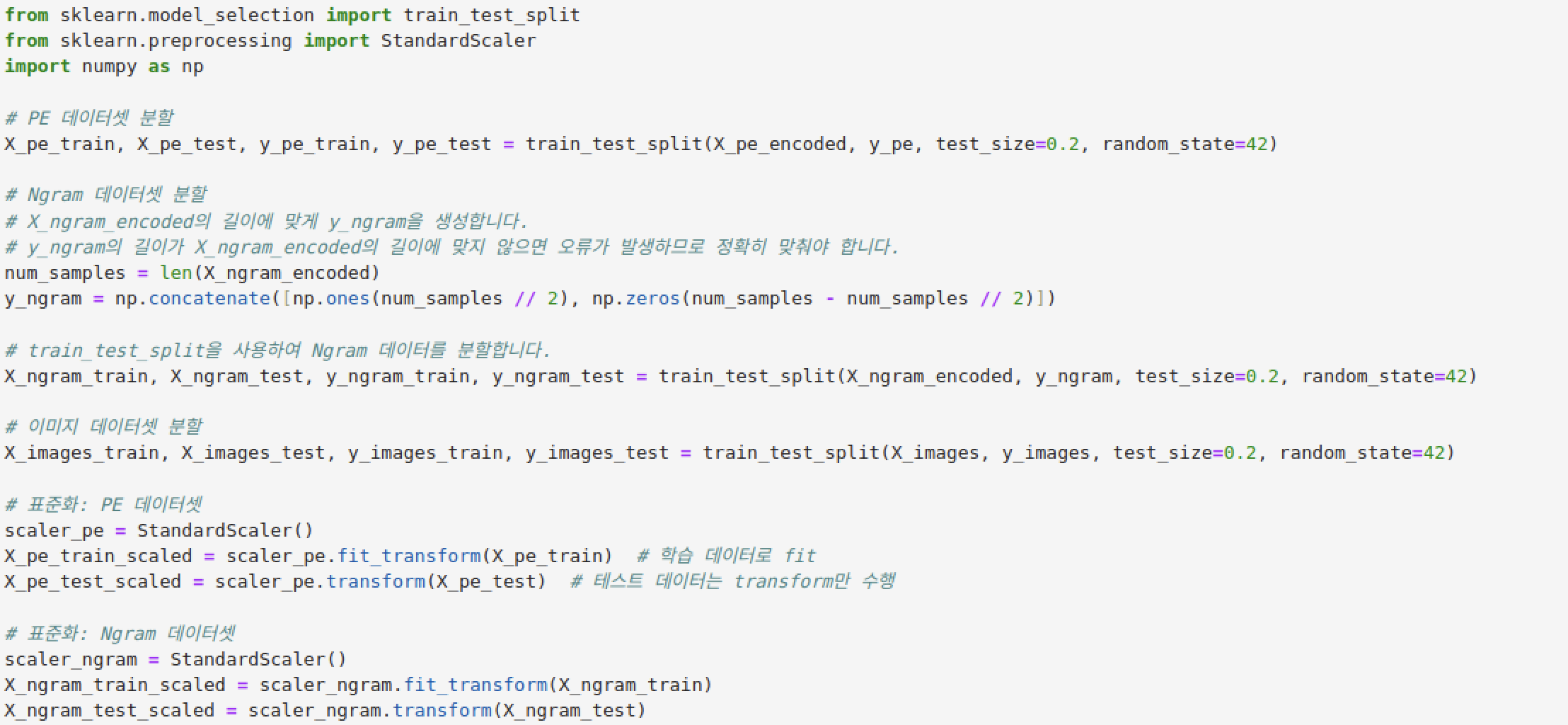
- 데이터 분할 (PE 데이터): train_test_split을 사용해 PE 데이터를 훈련 세트와 테스트 세트로 80:20 비율로 분할합니다.
- Ngram 레이블 생성: Ngram 데이터의 샘플 수를 기준으로 악성코드는 1, 정상은 0으로 레이블을 생성합니다.
- 데이터 분할 (Ngram 데이터): train_test_split을 사용해 Ngram 데이터를 훈련 세트와 테스트 세트로 80:20 비율로 분할합니다.
- 이미지 데이터 분할: train_test_split을 사용해 이미지 데이터를 훈련 세트와 테스트 세트로 80:20 비율로 분할합니다.
- 표준화 (PE 데이터): StandardScaler를 사용해 PE 훈련 데이터와 테스트 데이터를 표준화합니다.
- 표준화 (Ngram 데이터): StandardScaler를 사용해 Ngram 훈련 데이터와 테스트 데이터를 표준화합니다.
여기까지가 기본 세팅이다!
svm (pe, ngram)
randomforest (pe, ngram)
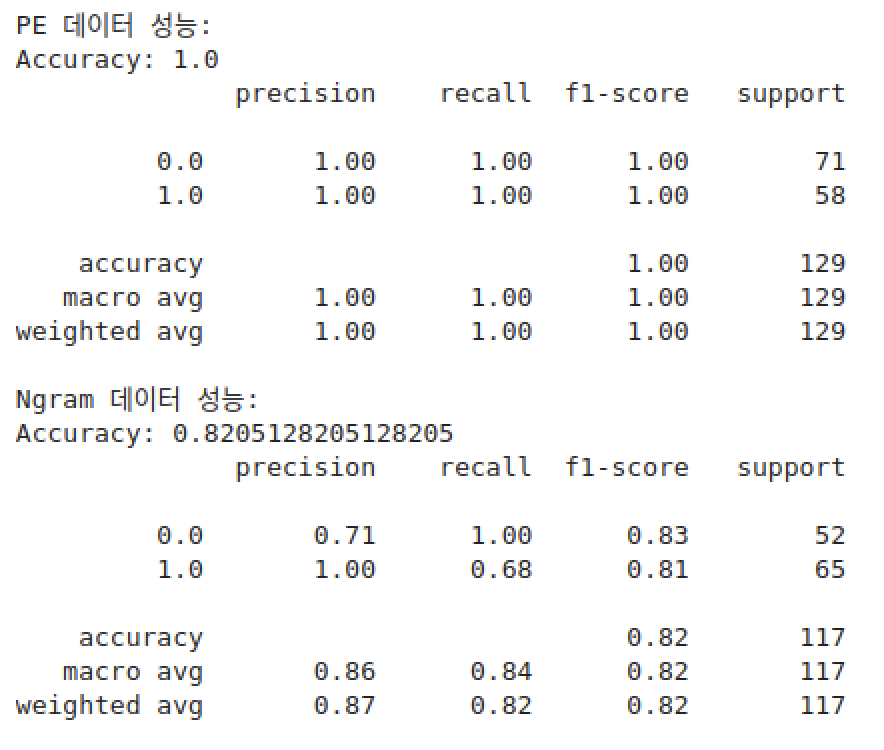
naivebayes (pe, ngram)

dnn (pe, ngram)

cnn (image)

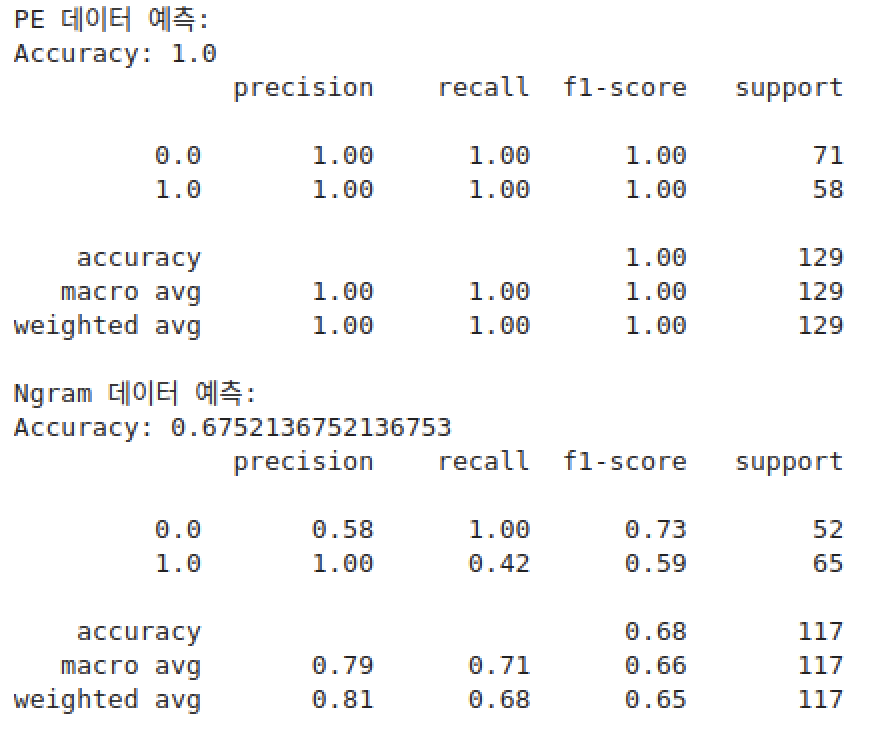
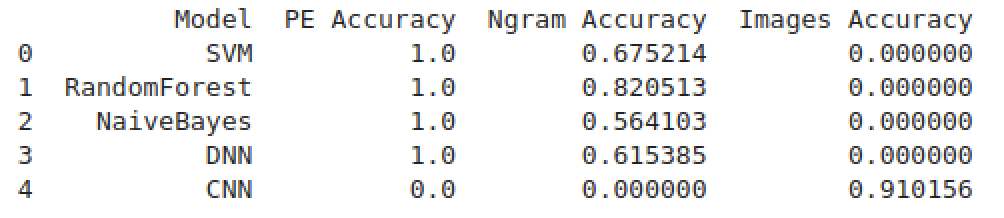
모델은 svm, randomforest, naivebayes, dnn, cnn을 돌렸다.
근데 뭔가 잘못되어서 pe가 모든 모델에서 과적합이 되었다. 열심히 해결해보려고 했는데, 시간도 부족하고 능력도 부족해서 결국 ngram 특징을 선택하기로 하였다ㅎㅎㅎ..
특징공학 결과
9장. PJ1_모델링
이제 모델링을 시작해볼건데, 특징공학때와 마찬가지로 책에서 어느정도 가이드라인을 잡고 8장의 결과를 기반으로 스스로 모델링을 진행해보려고 한다.
모델링의 방법에는 크게 4가지가 있다고 할 수 있다.
1. 정적 특징과 동적 특징을 하나의 특징으로 묶은 뒤 하나의 모델에 학습시키는 방법
2. 여러 모델의 결과를 토대로 새로운 결과 값을 도출하는 방법
3. 여러 모델을 직렬로 연결하는 방법
4. 클로스터링 모델을 통해 데이터를 여러 개의 군집으로 나눈 후, 각 군집에 맞는 분류 모델을 각각 학습시키는 방법
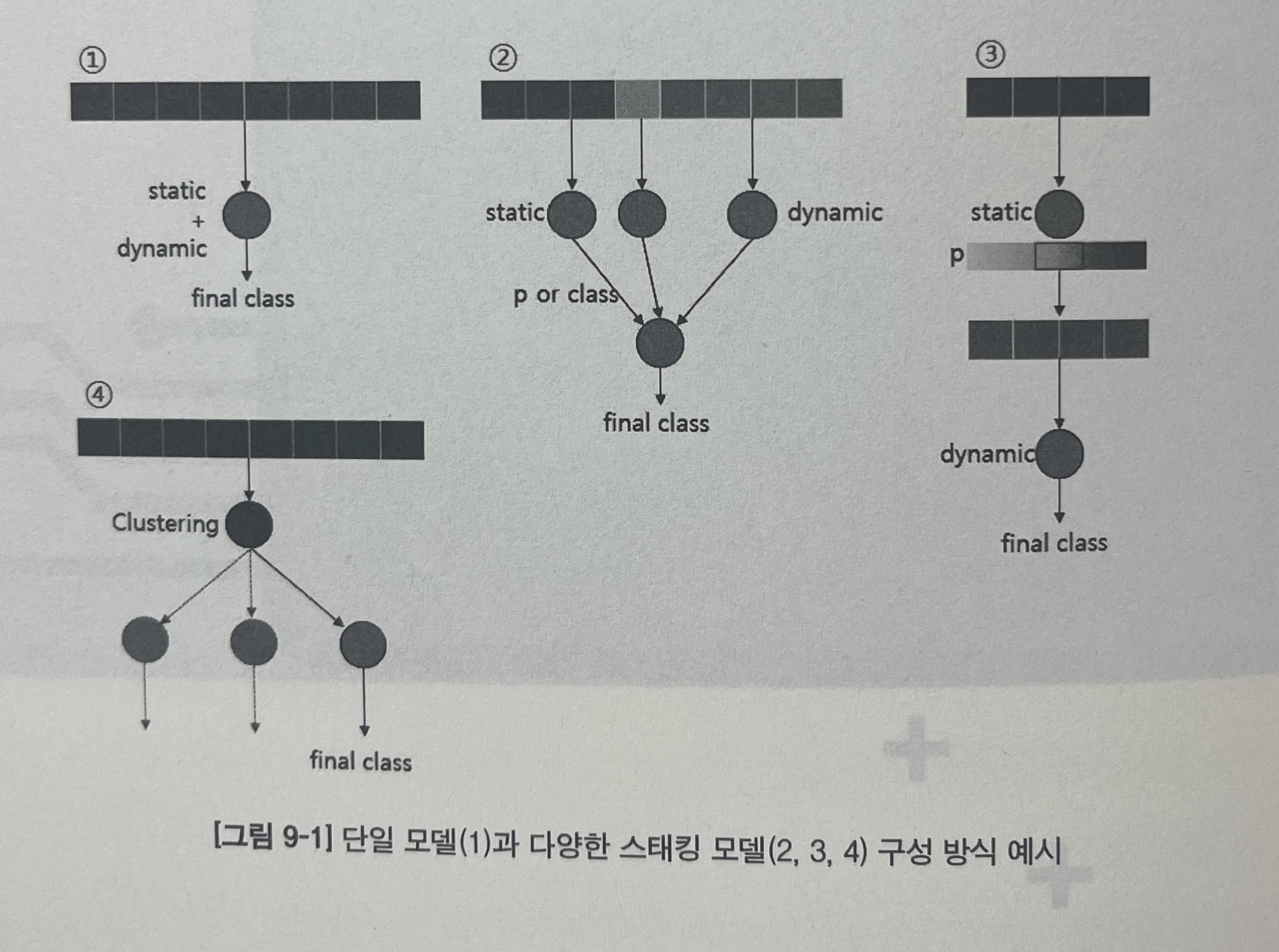
단일 모델
나는 위에서 언급했지만,, 반강제적으로 ngram 특징을 사용하는 randomforest 모델을 선택하게되었다. 특징이 정해진 상태라면 모델 매개변수를 조정해 정확도를 더욱 높일 수 있다.
RandomForestClassifier(n_estimators, criterion, max_depth, min_samples_split, min_samples_leaf, min_weight_fraction_leaf, max_features, max_leaf_nodes, min_impurity_decrease, min_impurity_split, bootstrap, oob_score, n_jobs, random_state, verbose, warm_start, class_weight)위의 코드가 랜덤포레스트의 결과에 영향을 줄 수 있는 여러 매개변수 옵션이다.
- n_estimators : 트리의 개수
- criterion : 트리 분할을 결정하는 함수
- max_depth : 트리의 최대 깊이
- min_samples_split : 내부 노드를 분할에 필요한 최소 샘플 개수
- min_samples_leaf : 단말 노드에 필요한 최소 샘플 개수
- min_weight_fraction_leaf : 단말 노드에 필요한 가중치 합의 최소 가중치 비율
- max_features : 최적의 트리 분할에 사용할 특징 개수
- max_leaf_nodes : 최대 단말 노드 개수
- min_impurity_decrease : 노드 분할 기준 (불순도 감소 정도)
- min_impurity_split : 트리 신장을 멈추는 임계치
- bootstrap : 부트스트랩(중복허용 샘플링) 샘플 사용 여부
- oob_score : 일반화된 정확도 예측을 위한 out-of-bag 샘플 사용 여부
- n_jobs : 병렬 작업 개수
- random_state : 랜덤 시드
- verbose : 자세히 보기 정도
- warm_start : 이전 트리 생성에 사용한 해결책 재사용 여부
- class_weight : 클래스 가중치 (멀티클래스 분류)
성능에 가장 큰 영향을 미치는 옵션은 n_estimator와 max_features 이다.
n_estimator는 판단에 사용할 트리의 개수로, 값이 클수록 더 좋은 결과를 얻을 수 있지만 그만큼 계산 시간이 길어진다는 단점이 있다.
max_features는 노드 분할 시 고려할 랜덤 특징 조합 크기를 의미하몀 분류 문제의 경우 sqrt(n_features)를 사용하는 것이 가장 좋다.
모델 매개변수가 달라짐에 따라 변화하는 정확도는 sklearn에서 제공하는 GridSearchCV를 이용하면 간단하게 분석할 수 있다. GridSearchCV 함수에 인자로 전달하는 cv는 Cross Validation (교차검증)의 약자로, 10-Fold 교차 검증을 수행한 결과를 토대로 최적의 매개변수 조합을 찾는다는 의미로 해석할 수 있다.
n_estimator는 100,200,500,1000 이고 max_features는 'sqrt'랑 None으로 설정하였다.
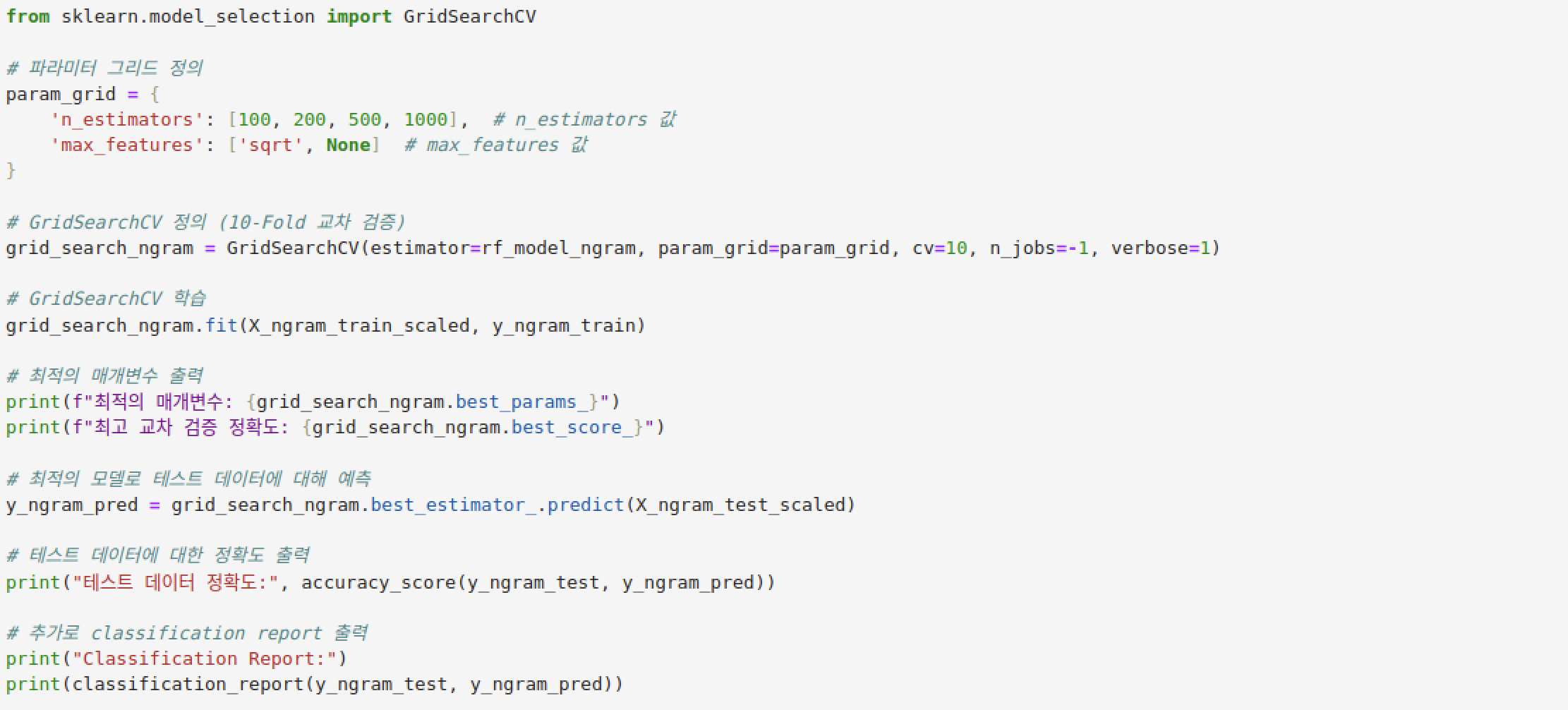

결과를 보면, 최고 교차 검증 정확도가 굉장히 많이 올랐으며, 테스트 데이터 정확도 역시 기존(Accuracy: 0.8205128205128205) 보다 조금 더 오른 것을 확인할 수 있다.
스태킹 모델
이 파트의 경우.. 그냥 한 번 시도해 보는 것이다. pe추출이 잘못되었기ㅠㅠ 때문에 나는 사용할 수 있는 특징이 ngram밖에 없긴한데.. 일단 도전!!!!!
pe 특징과 ngram 특징을 하나의 데이터로 합친 후 (열 병합), 학습 데이터와 테스트 데이터로 분리한다 (행 분할). 랜덤 포레스트와 SVM으로 학습 데이터를 학습시켜서 (GridSearchCV 사용) 새로운 특징 데이터를 생성시킨다. predict 함수 대신 predict_proba 함수를 사용하면 판단 결과가 아닌 각 클래스별 확률 값(악성일 확률, 정상일 확률)을 구할 수 있다. x_train과 x_test를 모두 predict_proba 함수에 전달해서 학습 데이터와 테스트 데이터의 특징 개수가 동일하게 한 뒤, DNN 모델에 학습 데이터를 넣어 학습한 후 테스트 데이터로 모델을 평가한다.


그렇다!!!!
import numpy as np
from sklearn.ensemble import RandomForestClassifier
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
from sklearn.metrics import classification_report
from tensorflow.keras.models import Sequential
from tensorflow.keras.layers import Dense, Dropout
from tensorflow.keras.optimizers import Adam
rf_params = {
'n_estimators': [100, 200],
'max_features': ['sqrt', None]
}
rf_model = GridSearchCV(RandomForestClassifier(random_state=42), rf_params, cv=5, n_jobs=-1, verbose=1)
rf_model.fit(X_train_scaled, y_train)
rf_train_features = rf_model.best_estimator_.predict_proba(X_train_scaled)
rf_test_features = rf_model.best_estimator_.predict_proba(X_test_scaled)
\uc0dd\uc131
svm_params = {
'C': [0.1, 1, 10],
'kernel': ['linear', 'rbf']
}
svm_model = GridSearchCV(SVC(probability=True, random_state=42), svm_params, cv=5, n_jobs=-1, verbose=1)
svm_model.fit(X_train_scaled, y_train)
svm_train_features = svm_model.best_estimator_.predict_proba(X_train_scaled)
svm_test_features = svm_model.best_estimator_.predict_proba(X_test_scaled)
train_features = np.hstack([rf_train_features, svm_train_features])
test_features = np.hstack([rf_test_features, svm_test_features])
dnn_model = Sequential([
Dense(128, activation='relu', input_dim=train_features.shape[1]),
Dropout(0.5),
Dense(64, activation='relu'),
Dropout(0.5),
Dense(1, activation='sigmoid')
dnn_model.compile(optimizer=Adam(learning_rate=0.001), loss='binary_crossentropy', metrics=['accuracy'])
dnn_model.fit(train_features, y_train, epochs=10, batch_size=32, validation_split=0.2, verbose=1)
test_loss, test_accuracy = dnn_model.evaluate(test_features, y_test, verbose=0)
print(f"DNN Test Accuracy: {test_accuracy:.4f}")
y_pred = (dnn_model.predict(test_features) > 0.5).astype(int)
print("Classification Report:")
print(classification_report(y_test, y_pred))
결과는~~~!
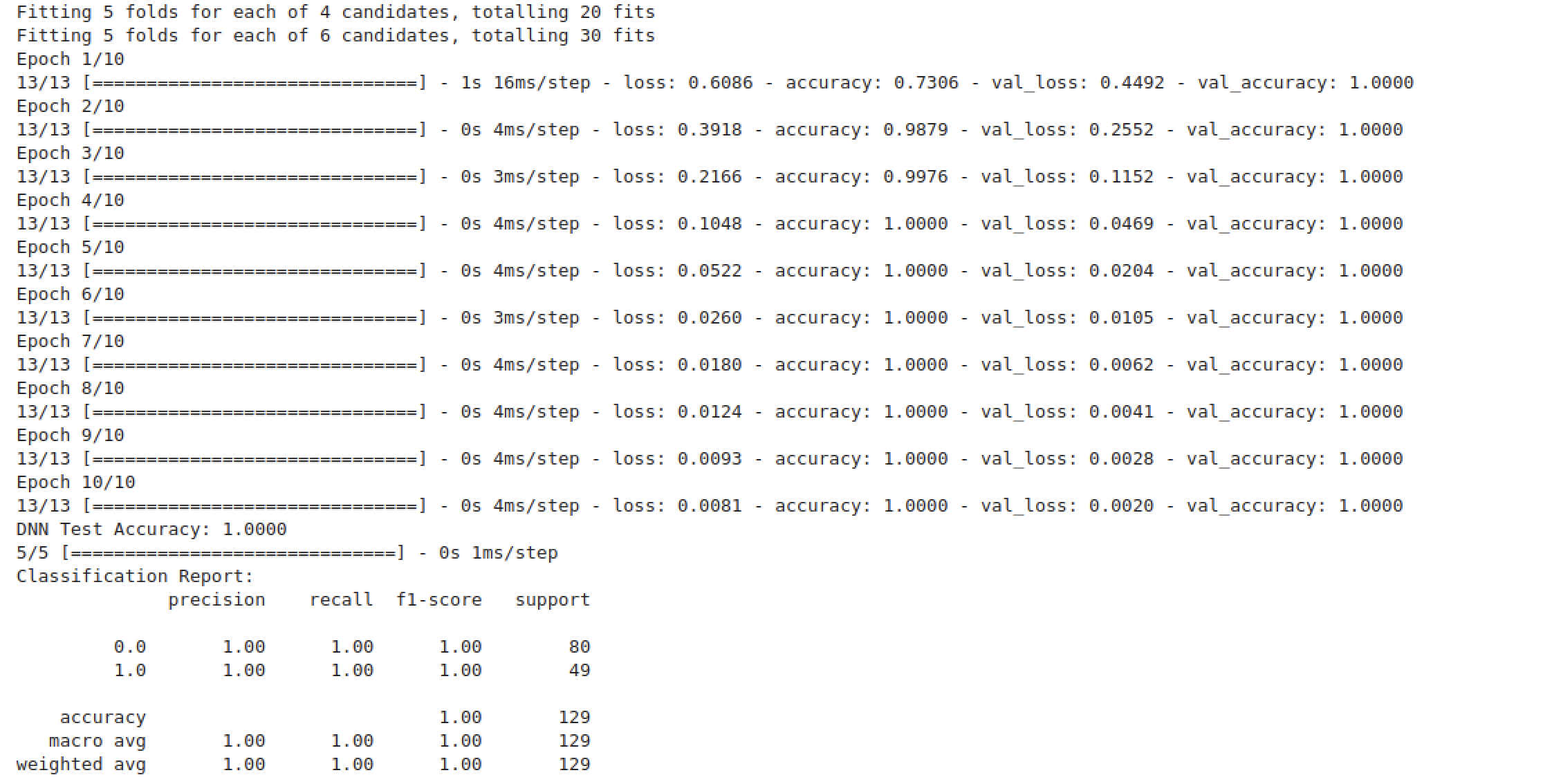
실패했습니다~!!!!
아니!!!!!!!!!!!!!!!!!!!!!!! 대체 뭐가 문제인거지?!?!?!?!?!? 저 pe파일 진짜 화나네
그냥 내가 만든 모델이 완벽한거라고 생각하자ㅎㅎ
실패했어도,, 실패한대로 기록은 올려놔야겠다. 스태킹 모델이라는 것에 대해 배울 수 있었던 시간인걸로~~ 그런걸로..~
모델 배치
학습한 모델을 실제 애플리케이션에 반영하는 것이 목표라면,
- 모델 배치 방식 : 재사용 가능한 모델 변수를 어떻게 저장하고 불러올 것인가?
- 특징 공학 과정을 코드화 : 학습 데이터와 테스트 데이터는 동일한 과정으로 처리해야 함
- 예외 처리 : 발생 가능한 모든 예외를 처리해야 함
- 기능 배치 방식 : 특징 추출과 판단 기능을 로컬에 탑재 or 원격에서 수행
백신 프로그램 내부에 엔진 형태로 모델을 탑재하려면 우선 학습 결과물이 되는 모델 변수를 별도의 파일로 저장해야한다. sklearn의 joblib 기능을 이용해 학습 결과를 저장하고 있는 모델 변수(clf)를 파일로 저장한다.
지금까지 만든 코드들 중에서 실제로 백신 엔진에 탑재할 부분만 하나의 코드로 모아보겠다.
모델은 단일모델(ngram + 랜덤포레스트)을 사용할 것이다. 이때 필요한 파일은 다음과 같다.
- 특징 추출 : ngram.py에 정의된 ngram 특징 추출 기능
- 모델 파일 : 랜덤포레스트 모델을 저장한 joblib 파일
- One-hot 인코딩 범주 : peid.yara 패턴 중 실제 특징으로 사용한 패턴 목록 파일
- 판단 기능 : single_model.ipynb 코드를 모듈로 작성
특징 추출의 경우, ngram.py에서 특징 추출 부분만 골라서 새롭게 extract_ngram.py를 작성해주었다.
import os
import pefile
import operator
from itertools import chain
from capstone import Cs, CS_ARCH_X86, CS_MODE_32
class NGRAM_features:
def __init__(self):
self.gram = dict()
def gen_list_n_gram(self, num, asm_list):
for i in range(0, len(asm_list), num):
yield asm_list[i:i+num]
def n_grams(self, num, asm_list, ex_mode):
gram = self.gram if ex_mode == 1 else dict()
gen_list = self.gen_list_n_gram(num, asm_list)
for lis in gen_list:
lis = " ".join(lis)
gram[lis] = gram.get(lis, 0) + 1
return gram
def get_ngram_count(self, headers, grams, label):
patterns = [grams.get(pat, 0) for pat in headers]
patterns.append(label)
return patterns
def get_opcodes(self, mode, file):
asm = []
try:
pe = pefile.PE(file)
except pefile.PEFormatError:
print(f"[ERROR] PE file format error: {file} is not a valid PE file or is empty.")
return []
ep = pe.OPTIONAL_HEADER.AddressOfEntryPoint
end = pe.OPTIONAL_HEADER.SizeOfCode
ep_ava = ep + pe.OPTIONAL_HEADER.ImageBase
for section in pe.sections:
addr = section.VirtualAddress
size = section.Misc_VirtualSize
if ep > addr and ep < (addr + size):
ep = addr
end = size
data = pe.get_memory_mapped_image()[ep:ep + end]
temp = data.hex()
temp = [temp[i:i + 2] for i in range(0, len(temp), 2)]
if mode:
return temp
# Capstone Disassembler
md = Cs(CS_ARCH_X86, CS_MODE_32)
md.detail = False
for insn in md.disasm(data, 0x401000):
asm.append(insn.mnemonic)
return asm
def extract_features(file_path, ngram_extractor, ngram_size=4):
data = []
try:
byte_code = ngram_extractor.get_opcodes(0, file_path)
if not byte_code:
return None
grams = ngram_extractor.n_grams(ngram_size, byte_code, 0)
data = grams
except Exception as e:
print(f"Error while processing {file_path}: {e}")
return data
def create_feature_vector(grams, headers):
return grams.get_ngram_count(headers, grams, 0) if grams else []joblib 파일의 경우,

코드를 통하여 만들어 내었고,
One-hot 인코딩 범주 역시 patterns.csv 파일로 저장해놨다.
그럼 이제 남은 것은 "판단 기능 : single_model.ipynb 코드를 모듈로 작성" 이다.

이런식으로! 물론 틀린 추측을 하기도 하지만, 어느정도 알맞은 판단을 하는 모델을 만들어냈다.
10장. PJ1_백신에 엔진 추가하기
악성코드 탐지 모델 구축의 최종 목적은 백신이 그 기능을 활용할 수 있도록 만드는 것이다. 프로젝트에서는 여러 오픈소스 백신들 중 키콤 백신(Kicom AV)을 활용한다. 프로젝트의 목표는 내가 만든 모델을 백신에 반영해보는 연습을 하는 것이므로, 백신 구조에 맞는 완전한 연동은 하지 않는다.
키콤 백신은 파이썬으로 제작된 오픈소스 백신으로, 소스코드는 크게 엔진(Engine) 부분과 도구(Tools) 폴더로 구성되며, 도구 폴더는 악성코드 패턴 생성 및 키 생성 도구를 포함한다. 엔진 폴더는 다시 kavcore와 plugins 폴더로 나뉜다.
책에서는 키콤 0.31 버전을 이용하여 실습을 진행하였는데,

2024.12 기준 v0.33c 인지라,, 어차피 가이드라인이니까 나는 0.33 버전으로 진행하려한다.
pip install pylzma psutil
./build.sh build
python k2.py
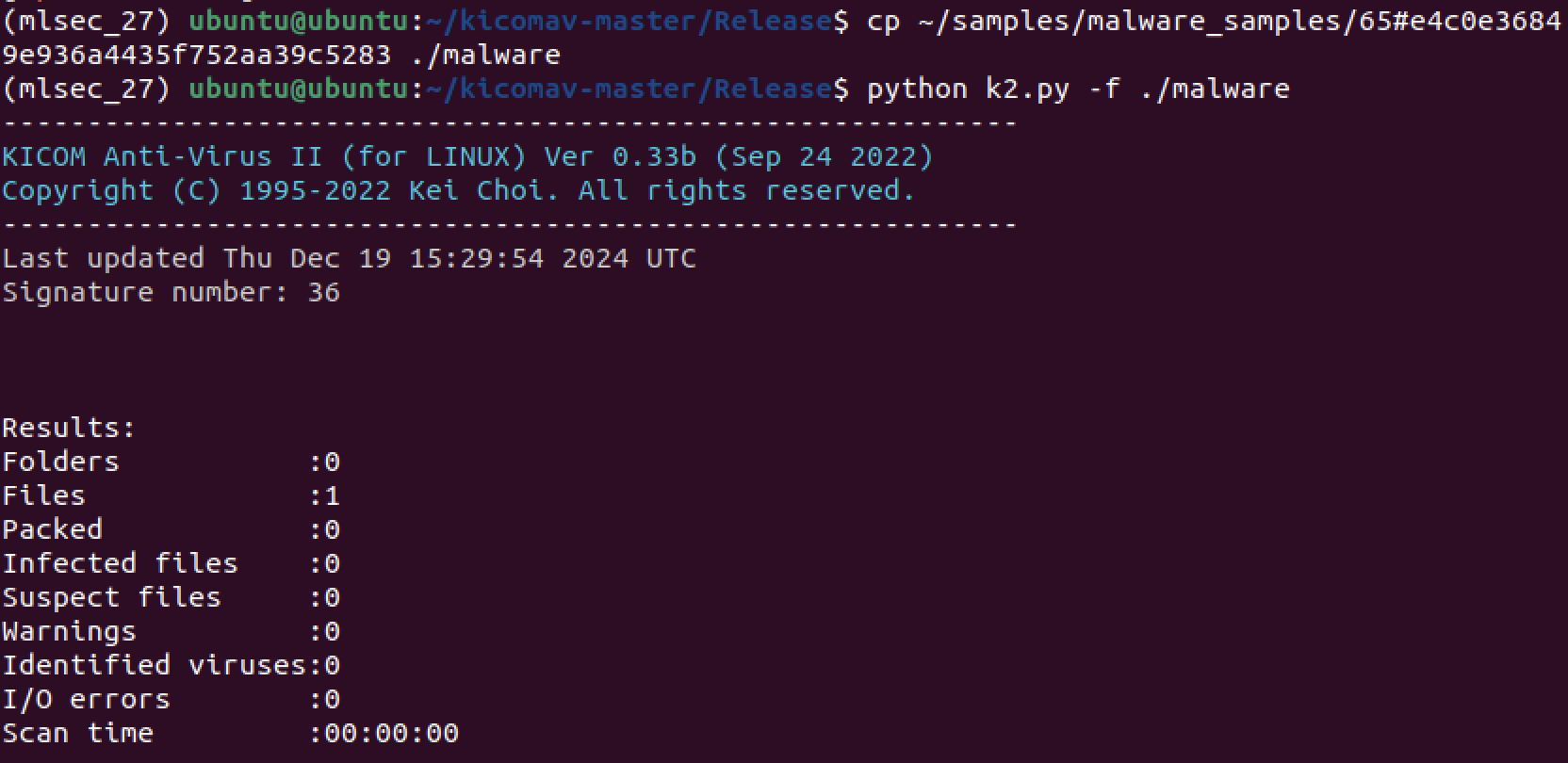
키콤 백신을 한 번 사용해 보기 위하여 샘플들 중 하나를 가지고 와서 돌려보았다.
Signature number: 36 >> 확인할 수 있다.
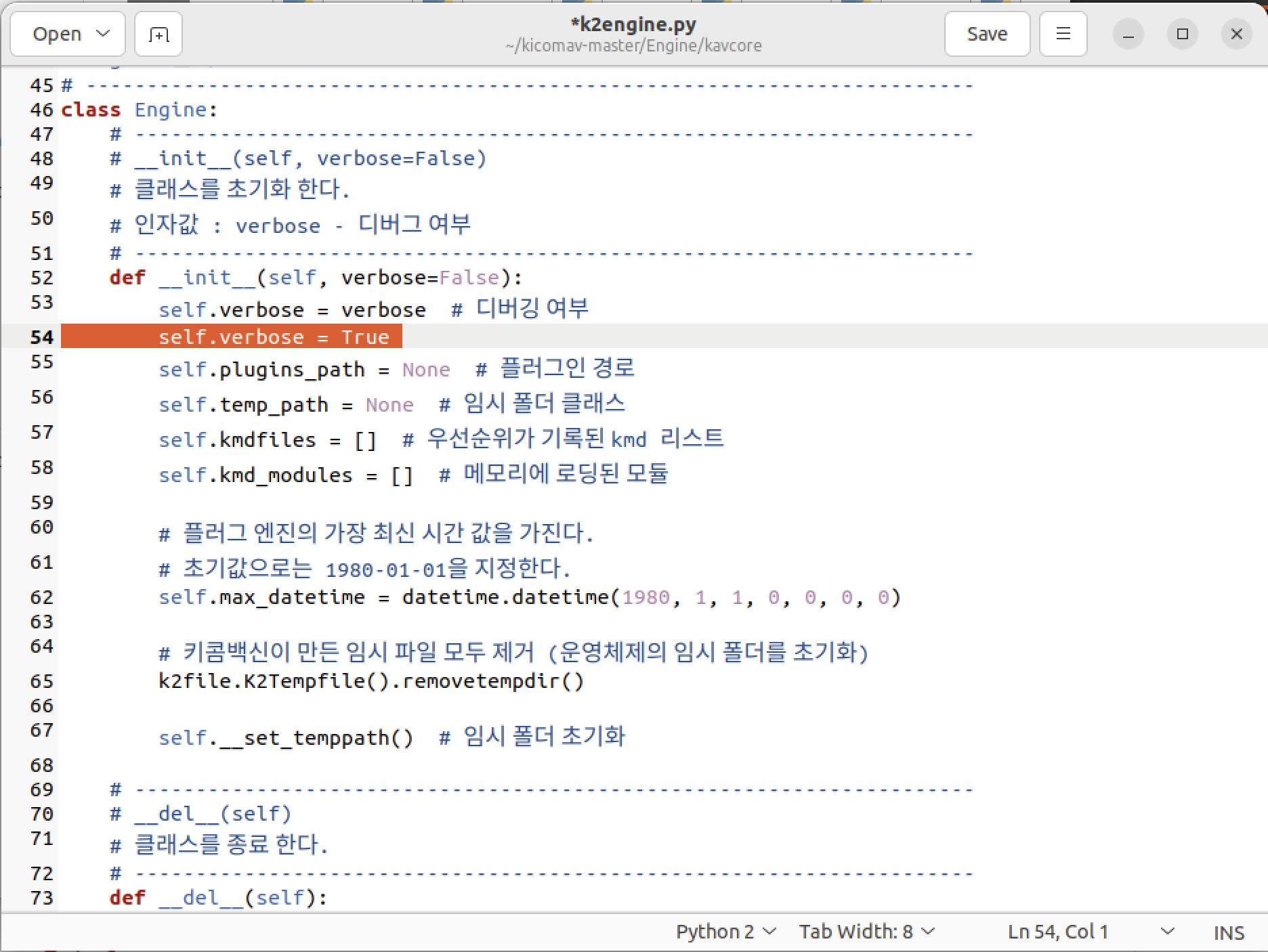
self.verbose = Truek2engine.py 파일에 verbose 옵션을 True로 추가하였다. 이를 통하여 디버깅 메세지를 통해서 키콤 백신이 malware 파일을 검사하는 과정을 알아낼 수 있다.
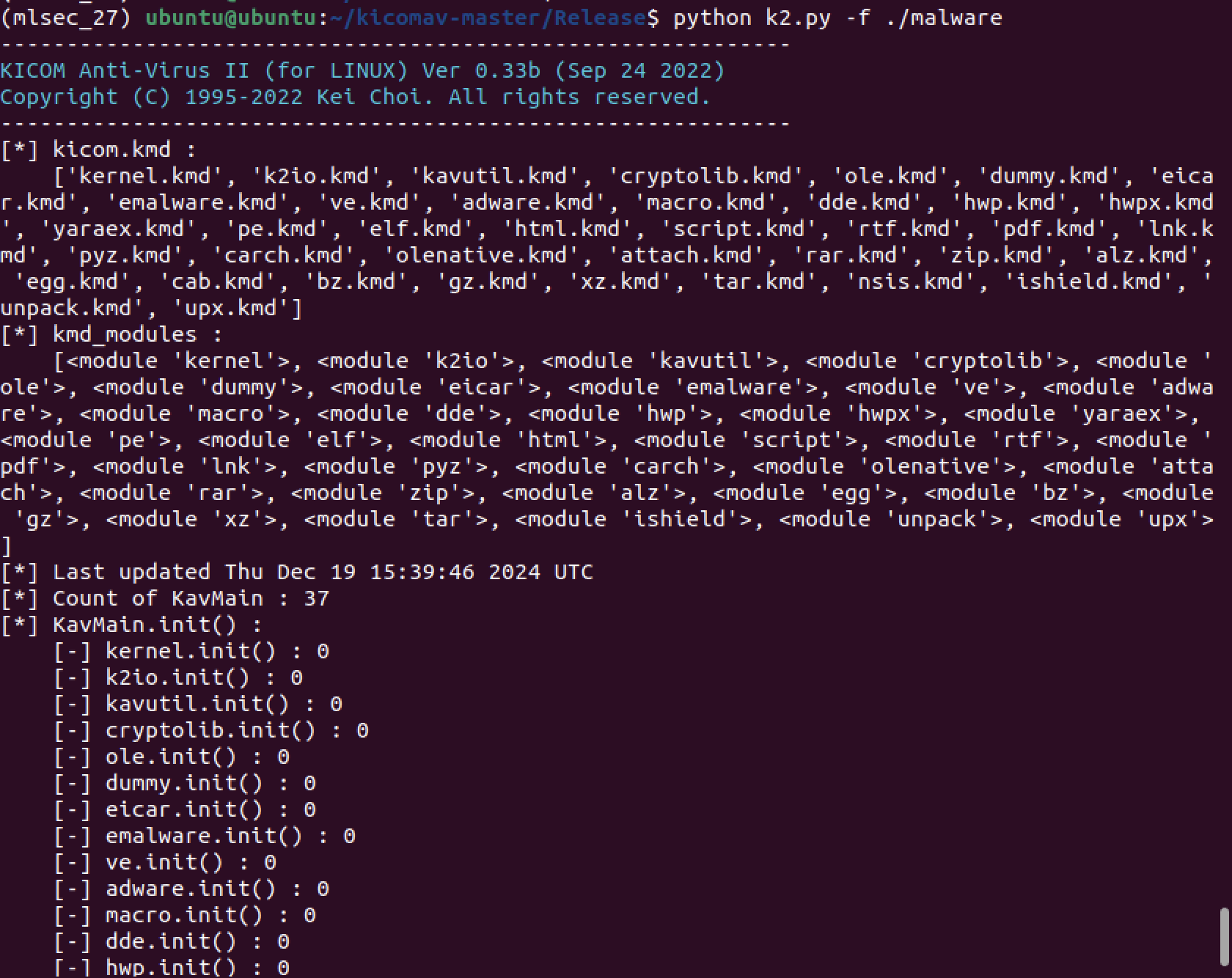
디버깅 모드 활성화 후 키콤 백신 실행 화면이다.
- kicom.kmd 목록 : kicom.kmd 파일에 기록되어 있는 플러그인 목록
- kmd_modules 목록 : 키콤 백신에 성공적으로 로드된 플러그인 목록
- Last update : 최종 업데이트 시간
- Count of KavMain : KavMain 개수 = 성공적으로 로드된 플로그인의 개수
- KavMain.init() 목록 : 로드된 모든 플러그인 내의 KavMain 클래스를 하나씩 초기화
- Count of KavMain.init() : 초기화된 KavMain 클래스 개수
- KavMain.__scan_file() : 악성코드 스캔 기능이 실행되었음을 알리는 메시지
- Results : 플로그인에 대상 파일을 검사한 결과
- KavMain.uninit() : 로드된 모든 플러그인을 해제
k2engine 내의 Engine 클래스는 kicom.kmd 파일 내에 기록된 플로그인 목록을 확인한 후 해당 플러그인을 하나씩 로드한다. 그리고 모듈을 초기화하고, 검사 대상 파일을 각 플러그인 모듈에 하나씩 대입해 악성코드를 검사한다. 검사가 완료된 후 탐지 결과를 보여주고 백신을 종료한다.
플러그인 추가 방법
1. 추가하려는 기능이 기존 시스템에 매끄럽게 연동될 수 있도록 기존 코드 분석
2. 새로운 플로그인 코드를 형식에 맞게 정의 (KavMain 클래스)
3. 플러그인 우선순위를 고려해 kicom.Ist에 새롭게 생성한 플러그인 파일 이름 기록
4. 플러그인 엔진 코드 작성 및 추가로 필요한 파일을 plugins 폴더에 복사
5. 엔진 동작 테스트
