들어가며
Causal model은 causal quantities를 확인하기 위해 필수적이다. 이전 내용에서는 Causal model에 대한 graphical intuition을 확인했다. 이번 글에서는 더 나아가 두 가지 내용에 대해 설명할 예정이다.
1. Indentify causal quantities
2. Formalize causal models
4.1 -operator and Interventional Distributions
일반적으로 확률에 대한 계산을 할 때, 우리는 조건부 확률 (conditional probability)을 흔하게 접할 수 있다. 그러나 이렇게 conditioning을 하는 것과 intervening을 하는 것은 전혀 다른 이야기다. 라는 conditioning을 진행하는 것은, 단순하게 treatment t를 부여받은 분포에만 관심을 가지겠다는 뜻이다. 우린 앞으로 intervention을 표현할 때 -operator를 활용할 것이다. 다음과 같이 표현한다.
Conditioning on 는 전체 모집단 혹은 관측한 데이터에서 treatment t를 부여받은 subset이 된다.
Intervention on 는 전제 모집단에 대해 로 생각한다는 뜻이다. 즉, Observation이 아닌 Doing(intervention)을 통해 인과관계를 도출하기 위한 과정 중 하나라고 생각 할 수 있다. 아래 그림을 통해 더 직관적으로 이해할 수 있다.
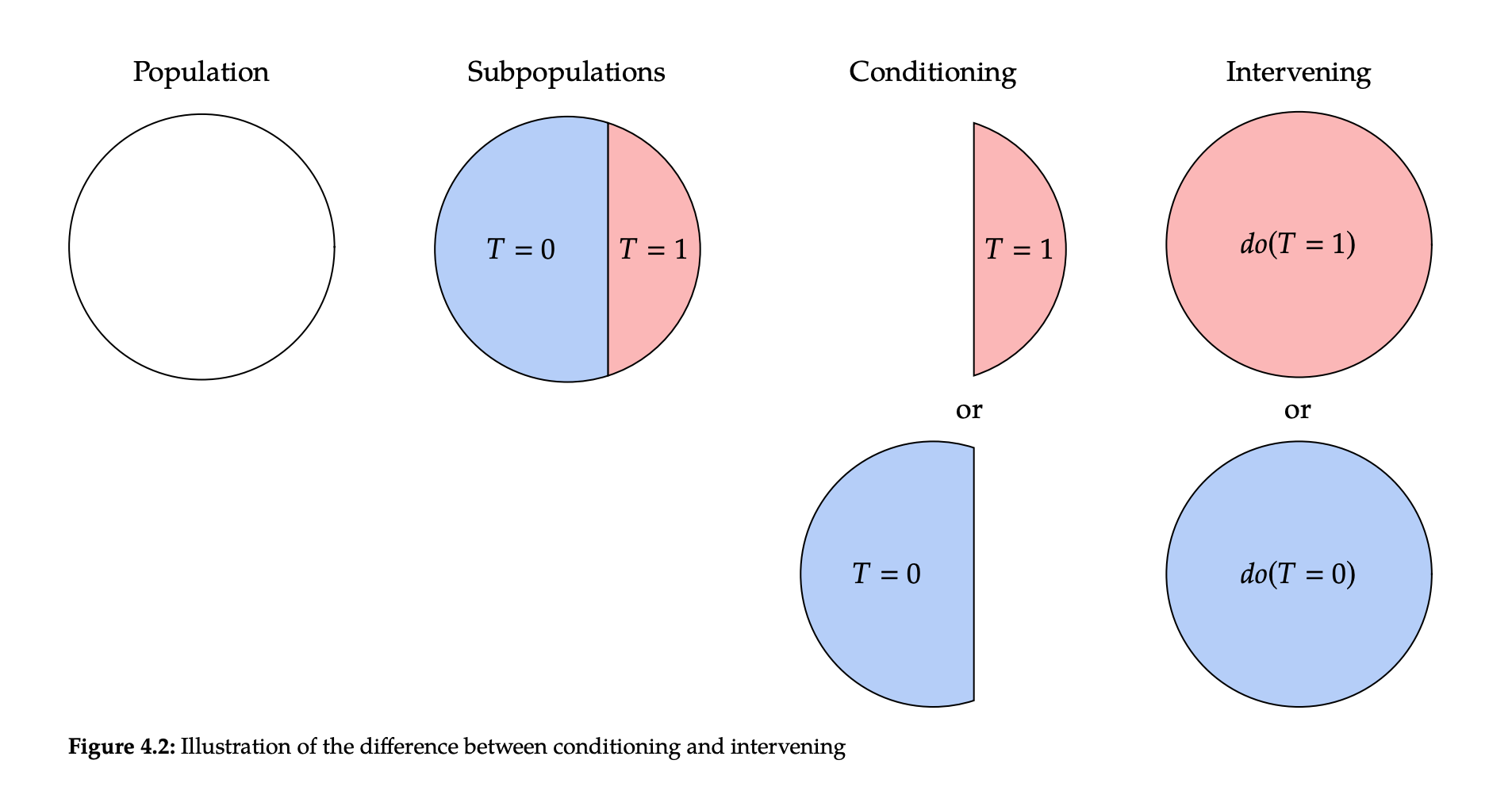
Observational distribution에 대해,
로 표현 가능 하다.
Interventional distribution의 경우,
로 표현 가능하다.
-operator를 통해 얻은 Causal effect를 Causal Estimand 라고 부르며, 그렇지 않은 관측치들에 대해서는 Statistical Estimand 라고 부른다. Identification의 가정하에, 우리는 Causal Estimand를 Statistical Estimand로 바꿀 수 있다. Treatment에 영향을 줄 수 있는 모든 변수의 효과를 무시하게 만들어주는게 바로 이 연산자다.
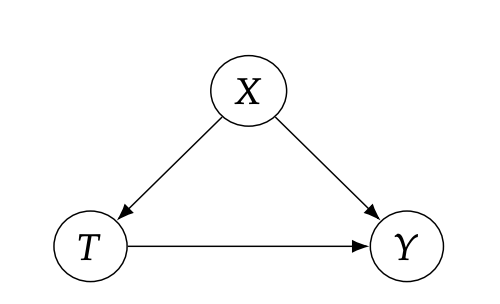
위 그래프에서 인과관계를 살펴보자. 만약, 노드가 존재하지 않았다면 를 통해 인과효과를 파악할 수 있었을 것이다. 하지만 Confounder인 의 존재로 인해, Backdoor path가 생기게 되어 인과효과를 파악하기 어려워진다. 여기서 -operator가 빛을 발휘한다. 바로 그래프 내의 와 를 잇는 edge를 삭제할 수 있게 된다는 점이다.
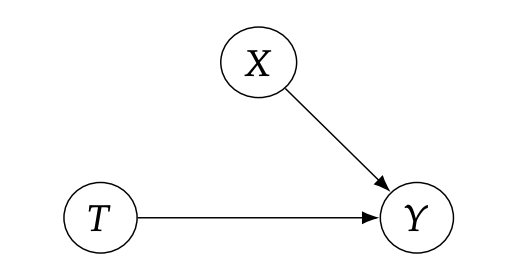
이런 그래프가 되는 것이다. 가 적용되었다고 생각하면 된다. 우리는 를 얻게 되며, 이 값이 바로 Causal effect라고 할 수 있다.
4.2 Main Assumption: Modularity
Causal Inference를 위해서는 정말 많은 가정들이 기반이 되어야 한다. 앞에서 계속 다뤘던 것처럼, interventions에 대해서도 'interventions are local' 이라는 가정이 들어가게 된다. Intervention node 에 대해서 가 미치는 영향에만 변화가 생기고 나머지 노드에서 받는 영향은 유지된다는 가정이다. 지금부터 다룰 Modularity assumption은 바로 이 'Inventions are local' 을 일반화한 가정이라고 생각하면 된다. 가정은 다음과 같다.
Assumption Modularity
If we intervene on a set of nodes , setting them to constants, then for all , we have the following:
1. If , then remains unchaged.
2. If , then if is the value that was set to by the intervention; otherwise, .
즉, 우리는 , , 가 서로 연관되지 않은 distribution임을 알 수 있다.
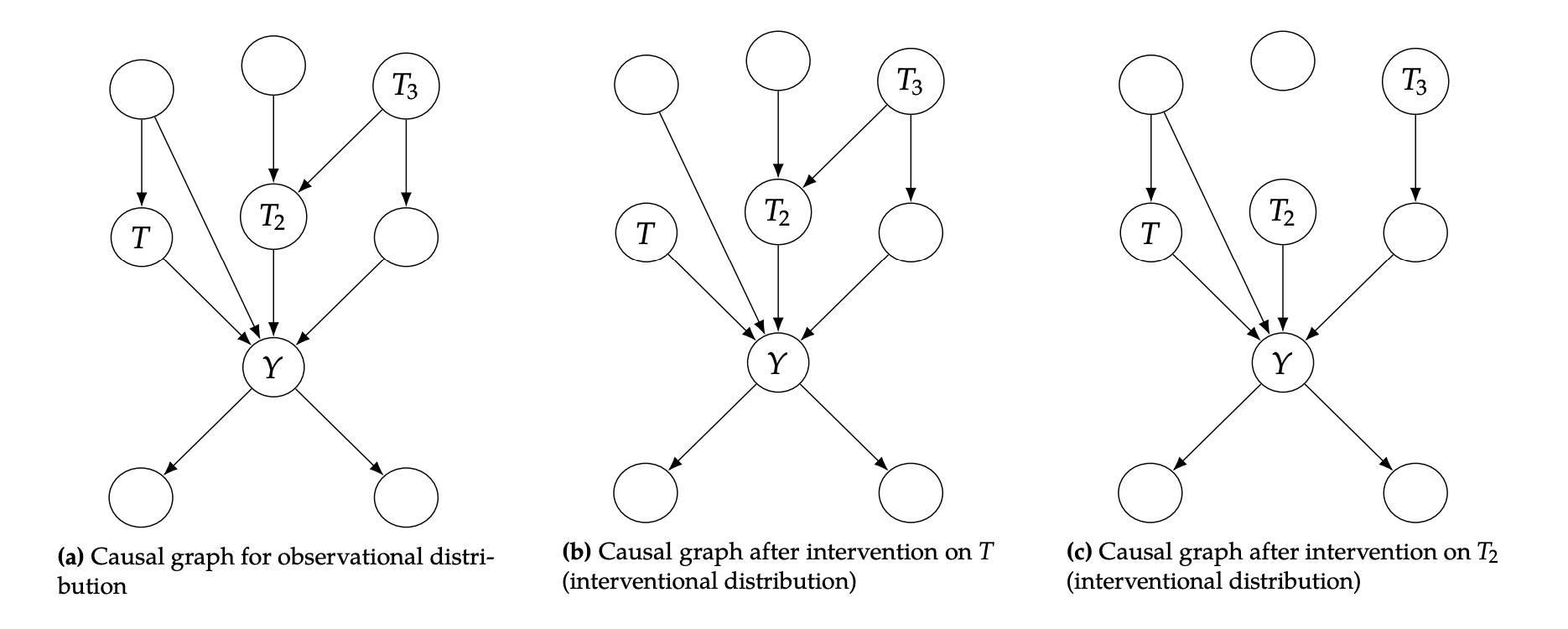
위 그림에서의 (b)와 (c)같이 edge가 제거된 그래프를 Manipulated graph라고 한다.
4.3 Truncated Factorization
앞에서 다룬 Bayesian network factorization을 떠올려보자.
여기서 Modularity assumption을 적용할 수 있다. Intervention이 적용된 nodes set 에 포함되는 노드들은, 이 되어버리기 때문에, factorization에서 생략할 수 있다.

위와 같은 식이 도출된다.
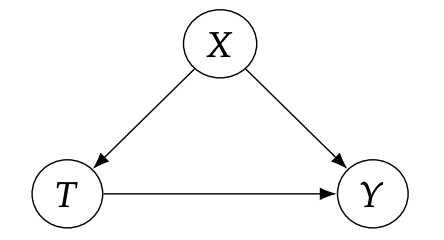
위 그래프에 대해 를 통해,
Bayesian network factorization:
Truncated factorization:
Marginalize:
를 확인 가능하다.
4.4 Backdoor Adjustment
이전 챕터에서 다뤘던 내용 처럼 backdoor path를 막는 것은 인과관계를 도출하기 위해 필수적이다. 이전 챕터에서는 conditioning을 통해 임의로 path를 차단하면서 potential outcome을 도출해 냈다. 이번 챕터에서는 -operator를 통해 edge 자체를 삭제함과 동시에 backdoor path를 없애는 방식으로 optential outcome을 도출한다.
먼저, Backdoor Adjustment에 대해서 알아야 한다.
Backdoor Adjustment
Given the modularity assumption, that satisfies the backdoor criterion, and positivity, we can identify the causal effect of on :
Proof of backdoor adjustment
여기서 드는 의문점이 있다. Conditioning을 통한 Potential outcome과 backdoor adjustment를 통해 얻을 수 있는 결과값이 같다면, 왜 굳이 -operator를 통해 backdoor path를 막는 것인지 의문이 든다. 뭐, chapter 14에서 더 자세하게 다룬다고는 하니까 믿고 기다려보기로 한다...
4.5 Structural Causal Models (SCMs)
지금까지 다룬 내용을 Structural causal model로 만들었다고 이해하면 된다. 인과관계에서는 역이 성립하지 않는다. 다시 말해, 라고 해서 가 인과관계에서는 성립하지 않는다.
그래서 다음과 같은 notation을 사용한다.
위 수식은 아래 그림에 대한 내용이다.
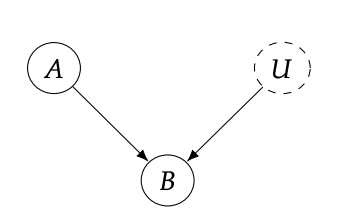
이 수식을 좀 더 확장해서 다음과 같은 그래프에 대해서 표현 할 수 있다.
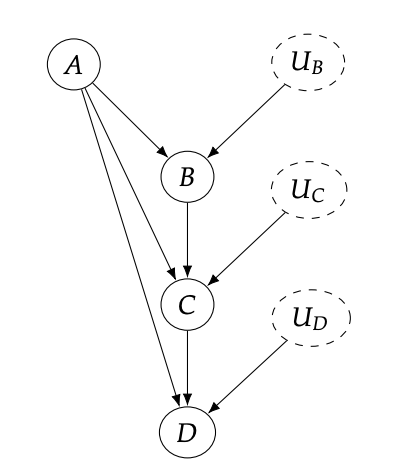
위 그래프에 대해서 우리는
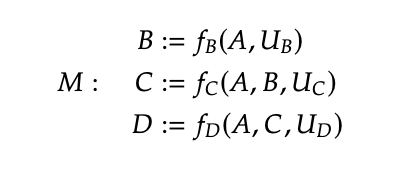
이렇게 표현 할 수 있다.
Collider의 경우 앞에서 다뤘던 것 처럼, block 하는 순간 부모 노드들 사이의 independent가 없어지게 된다. 그렇기에 SCMs에서도 collider에 유의해야 한다.
에 대해서 intervention on 를 진행하면 다음과 같이 표현 된다.
4.6 Example Application of Backddor Adjustment

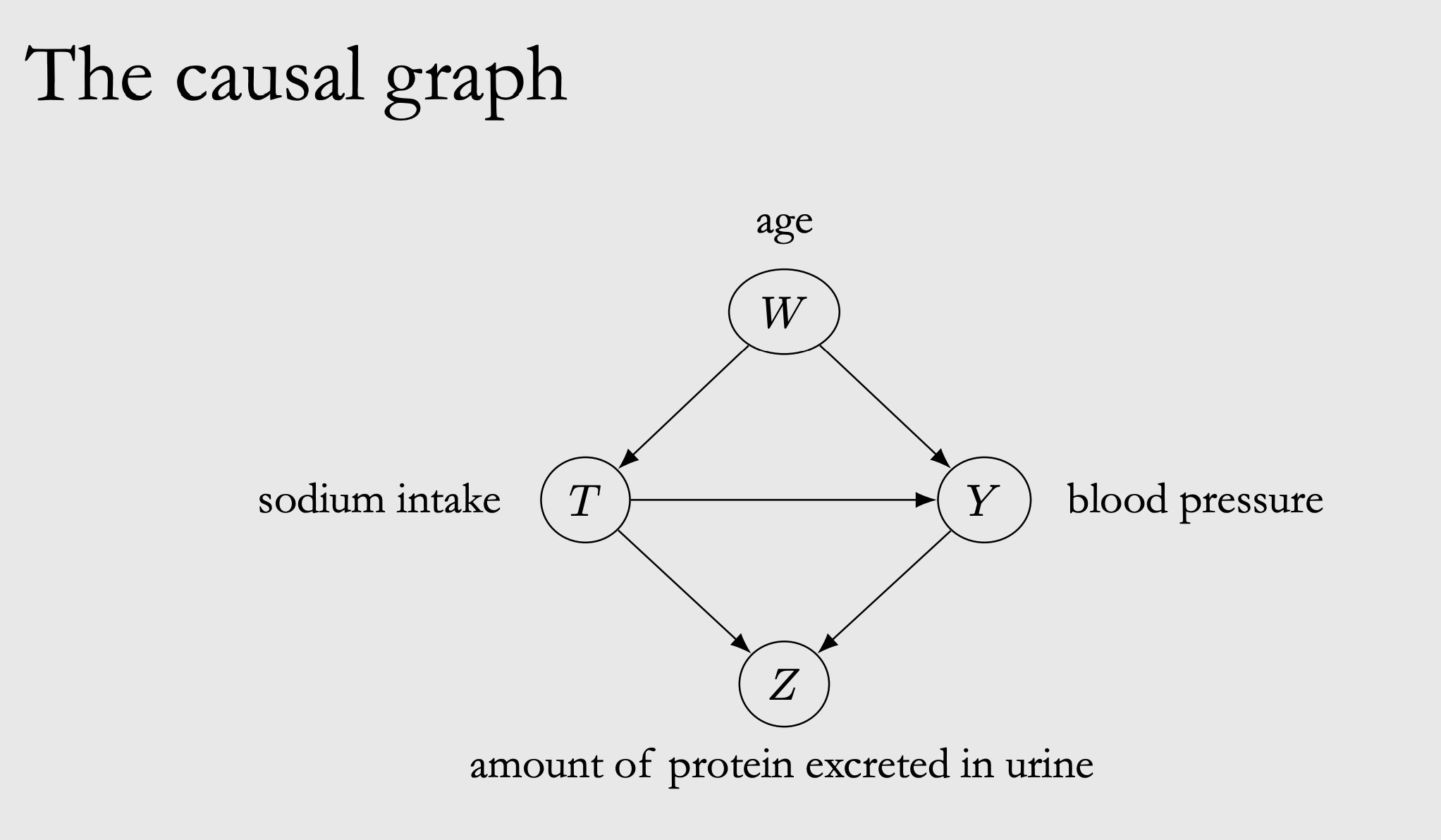

"""
Estimating the causal effect of sodium on blood pressure in a simulated example
adapted from Luque-Fernandez et al. (2018):
https://academic.oup.com/ije/article/48/2/640/5248195
"""
import numpy as np
import pandas as pd
from sklearn.linear_model import LinearRegression
def generate_data(n=1000, seed=0, beta1=1.05, alpha1=0.4, alpha2=0.3, binary_treatment=True, binary_cutoff=3.5):
np.random.seed(seed)
age = np.random.normal(65, 5, n)
sodium = age / 18 + np.random.normal(size=n)
if binary_treatment:
if binary_cutoff is None:
binary_cutoff = sodium.mean()
sodium = (sodium > binary_cutoff).astype(int)
blood_pressure = beta1 * sodium + 2 * age + np.random.normal(size=n)
proteinuria = alpha1 * sodium + alpha2 * blood_pressure + np.random.normal(size=n)
hypertension = (blood_pressure >= 140).astype(int) # not used, but could be used for binary outcomes
return pd.DataFrame({'blood_pressure': blood_pressure, 'sodium': sodium,
'age': age, 'proteinuria': proteinuria})
def estimate_causal_effect(Xt, y, model=LinearRegression(), treatment_idx=0, regression_coef=False):
model.fit(Xt, y)
if regression_coef:
return model.coef_[treatment_idx]
else:
Xt1 = pd.DataFrame.copy(Xt)
Xt1[Xt.columns[treatment_idx]] = 1
Xt0 = pd.DataFrame.copy(Xt)
Xt0[Xt.columns[treatment_idx]] = 0
return (model.predict(Xt1) - model.predict(Xt0)).mean()
if __name__ == '__main__':
binary_t_df = generate_data(beta1=1.05, alpha1=.4, alpha2=.3, binary_treatment=True, n=10000000)
continuous_t_df = generate_data(beta1=1.05, alpha1=.4, alpha2=.3, binary_treatment=False, n=10000000)
ate_est_naive = None
ate_est_adjust_all = None
ate_est_adjust_age = None
for df, name in zip([binary_t_df, continuous_t_df],
['Binary Treatment Data', 'Continuous Treatment Data']):
print()
print('### {} ###'.format(name))
print()
# Adjustment formula estimates
ate_est_naive = estimate_causal_effect(df[['sodium']], df['blood_pressure'], treatment_idx=0)
ate_est_adjust_all = estimate_causal_effect(df[['sodium', 'age', 'proteinuria']],
df['blood_pressure'], treatment_idx=0)
ate_est_adjust_age = estimate_causal_effect(df[['sodium', 'age']], df['blood_pressure'])
print('# Adjustment Formula Estimates #')
print('Naive ATE estimate:\t\t\t\t\t\t\t', ate_est_naive)
print('ATE estimate adjusting for all covariates:\t', ate_est_adjust_all)
print('ATE estimate adjusting for age:\t\t\t\t', ate_est_adjust_age)
print()
# Linear regression coefficient estimates
ate_est_naive = estimate_causal_effect(df[['sodium']], df['blood_pressure'], treatment_idx=0,
regression_coef=True)
ate_est_adjust_all = estimate_causal_effect(df[['sodium', 'age', 'proteinuria']],
df['blood_pressure'], treatment_idx=0,
regression_coef=True)
ate_est_adjust_age = estimate_causal_effect(df[['sodium', 'age']], df['blood_pressure'],
regression_coef=True)
print('# Regression Coefficient Estimates #')
print('Naive ATE estimate:\t\t\t\t\t\t\t', ate_est_naive)
print('ATE estimate adjusting for all covariates:\t', ate_est_adjust_all)
print('ATE estimate adjusting for age:\t\t\t\t', ate_est_adjust_age)
print()