
5-3. GenomicRangeQuery
For example, consider string S = CAGCCTA and arrays P, Q such that:
P[0] = 2 Q[0] = 4
P[1] = 5 Q[1] = 5
P[2] = 0 Q[2] = 6The answers to these M = 3 queries are as follows:
The part of the DNA between positions 2 and 4 contains nucleotides G and C (twice), whose impact factors are 3 and 2 respectively, so the answer is 2.
The part between positions 5 and 5 contains a single nucleotide T, whose impact factor is 4, so the answer is 4.
The part between positions 0 and 6 (the whole string) contains all nucleotides, in particular nucleotide A whose impact factor is 1, so the answer is 1.
Write a function:
class Solution { public int[] solution(String S, int[] P, int[] Q); }
that, given a non-empty string S consisting of N characters and two non-empty arrays P and Q consisting of M integers, returns an array consisting of M integers specifying the consecutive answers to all queries.
Result array should be returned as an array of integers.
For example, given the string S = CAGCCTA and arrays P, Q such that:
P[0] = 2 Q[0] = 4
P[1] = 5 Q[1] = 5
P[2] = 0 Q[2] = 6the function should return the values [2, 4, 1], as explained above.
Write an efficient algorithm for the following assumptions:
N is an integer within the range [1..100,000];
M is an integer within the range [1..50,000];
each element of arrays P and Q is an integer within the range [0..N - 1];
P[K] ≤ Q[K], where 0 ≤ K < M;
string S consists only of upper-case English letters A, C, G, T.
DNA 서열은 A, C, G, T 문자로 구성된 문자열로 표현될 수 있습니다. 각 문자는 연속적인 뉴클레오타이드의 종류를 나타냅니다. 각 뉴클레오타이드는 영향 인자(impact factor)를 가지며, 이는 정수입니다. A, C, G, T 유형의 뉴클레오타이드는 각각 1, 2, 3, 4의 영향 인자를 가집니다. 여러분은 주어진 DNA 서열의 특정 부분에 포함된 뉴클레오타이드의 최소 영향 인자를 묻는 여러 쿼리에 답해야 합니다.
DNA 서열은 N개의 문자로 구성된 비어 있지 않은 문자열 S = S[0]S[1]…S[N-1]로 주어집니다. M개의 쿼리가 있으며, 각 쿼리는 M개의 정수로 구성된 비어 있지 않은 배열 P와 Q로 주어집니다. K번째 쿼리(0 ≤ K < M)는 P[K]와 Q[K] 사이(포함)의 DNA 서열에 포함된 뉴클레오타이드의 최소 영향 인자를 찾아야 합니다.
예를 들어, 문자열 S = CAGCCTA와 배열 P, Q가 다음과 같다고 가정합니다
P[0] = 2 Q[0] = 4
P[1] = 5 Q[1] = 5
P[2] = 0 Q[2] = 6
이 M = 3개의 쿼리에 대한 답은 다음과 같습니다:
위치 2에서 4 사이의 DNA 부분에는 뉴클레오타이드 G와 C(두 번)가 포함되어 있으며, 이들의 영향 인자는 각각 3과 2이므로 답은 2입니다.
위치 5에서 5 사이의 부분에는 단일 뉴클레오타이드 T가 포함되어 있으며, 이의 영향 인자는 4이므로 답은 4입니다.
위치 0에서 6 사이의 전체 문자열에는 모든 뉴클레오타이드가 포함되어 있으며, 특히 영향 인자가 1인 뉴클레오타이드 A가 포함되어 있으므로 답은 1입니다.
예를 들어, 문자열 S = CAGCCTA와 배열 P, Q가 다음과 같다면:
P[0] = 2 Q[0] = 4
P[1] = 5 Q[1] = 5
P[2] = 0 Q[2] = 6
함수는 [2, 4, 1] 값을 반환해야 합니다.
다음 가정을 위한 효율적인 알고리즘을 작성하세요:
N은 [1…100,000] 범위 내의 정수입니다.
M은 [1…50,000] 범위 내의 정수입니다.
배열 P와 Q의 각 요소는 [0…N - 1] 범위 내의 정수입니다.
P[K] ≤ Q[K], 여기서 0 ≤ K < M입니다.
문자열 S는 대문자 영어 문자 A, C, G, T로만 구성됩니다.
문제풀이
import java.util.*;
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
int M = P.length;
String str = "";
ArrayList<Integer> result = new ArrayList<>();
for(int i = 0; i < M; i++){
str = S.substring(P[i], Q[i] + 1);
if(str.contains("A")){
result.add(1);
}
else if(str.contains("C")){
result.add(2);
}
else if(str.contains("G")){
result.add(3);
}
else if(str.contains("T")){
result.add(4);
}
}
int[] resultArray = new int[result.size()];
for(int i = 0; i < result.size(); i++) {
resultArray[i] = result.get(i);
}
return resultArray;
}
}반복문에서 문자열 S를 자른 문자열에 A,C,G,T가 포함되어있는지 확인하고 해당 값에 맞는 정수를 배열에 넣어 값을 리턴해줬습니다.
제출결과
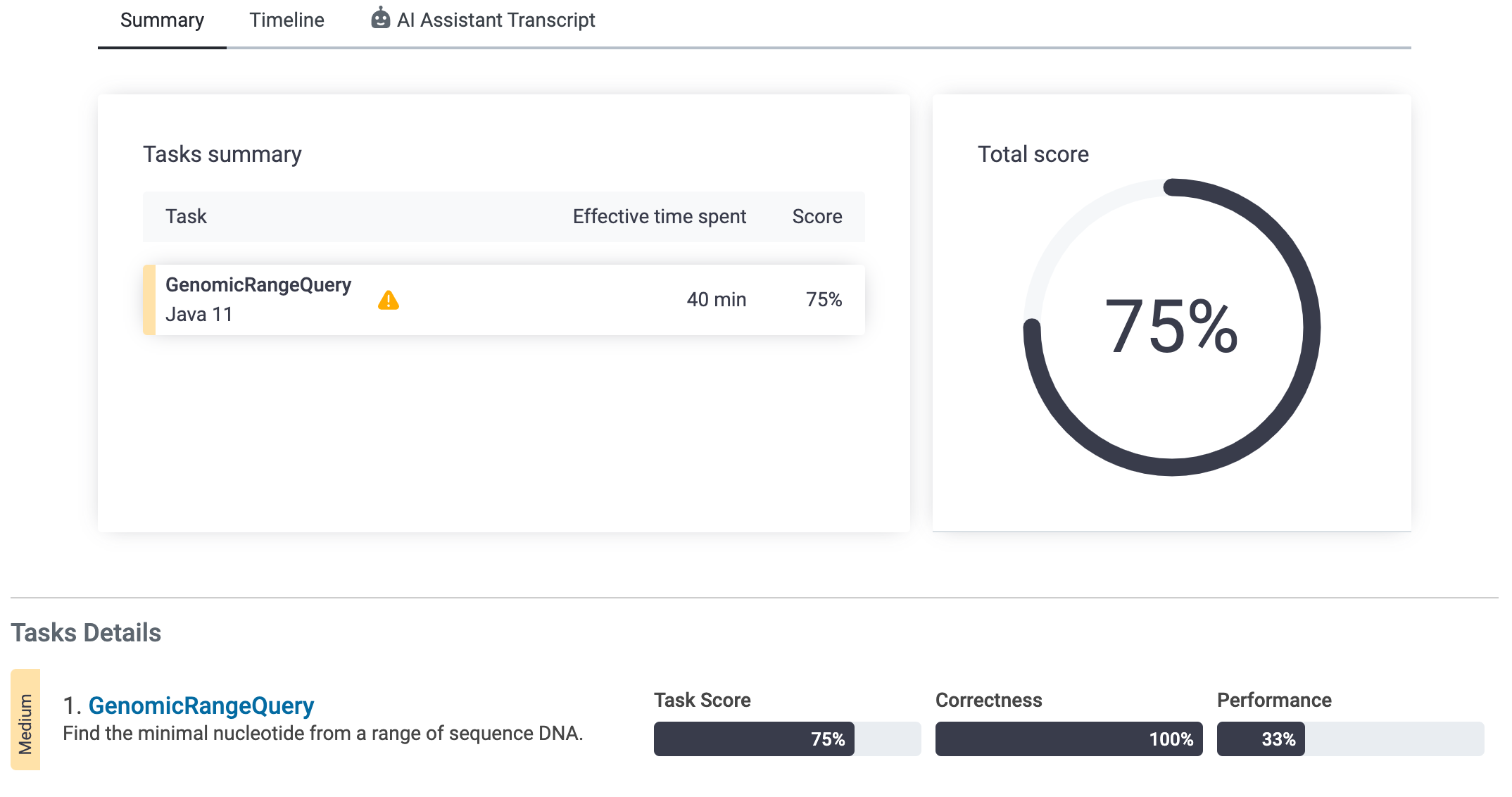
타임아웃 에러발생

해시맵으로 변경해도 동일하게 75%..
검색해보니 누적 개수를 저장하는 방식으로 문제를 풀 수 있다는 내용을 알았다.
수정된 코드
import java.util.*;
class Solution {
public int[] solution(String S, int[] P, int[] Q) {
int N = S.length();
int M = P.length;
int[] result = new int[M];
// 누적 개수 배열 생성
int[][] prefixSums = new int[4][N + 1];
for (int i = 0; i < N; i++) {
char nucleotide = S.charAt(i);
for (int j = 0; j < 4; j++) {
prefixSums[j][i + 1] = prefixSums[j][i];
}
if (nucleotide == 'A') {
prefixSums[0][i + 1]++;
} else if (nucleotide == 'C') {
prefixSums[1][i + 1]++;
} else if (nucleotide == 'G') {
prefixSums[2][i + 1]++;
} else if (nucleotide == 'T') {
prefixSums[3][i + 1]++;
}
}
// 각 쿼리에 대해 최소 영향 인자 찾기
for (int i = 0; i < M; i++) {
int start = P[i];
int end = Q[i] + 1;
if (prefixSums[0][end] - prefixSums[0][start] > 0) {
result[i] = 1;
} else if (prefixSums[1][end] - prefixSums[1][start] > 0) {
result[i] = 2;
} else if (prefixSums[2][end] - prefixSums[2][start] > 0) {
result[i] = 3;
} else if (prefixSums[3][end] - prefixSums[3][start] > 0) {
result[i] = 4;
}
}
return result;
}
}
각 쿼리에 대해 부분 문자열을 생성하고 검사하는 대신, 누적 합(prefix sum)을 사용하여 각 쿼리에 대해 빠르게 최소 영향 인자를 찾을 수 있다
제출결과
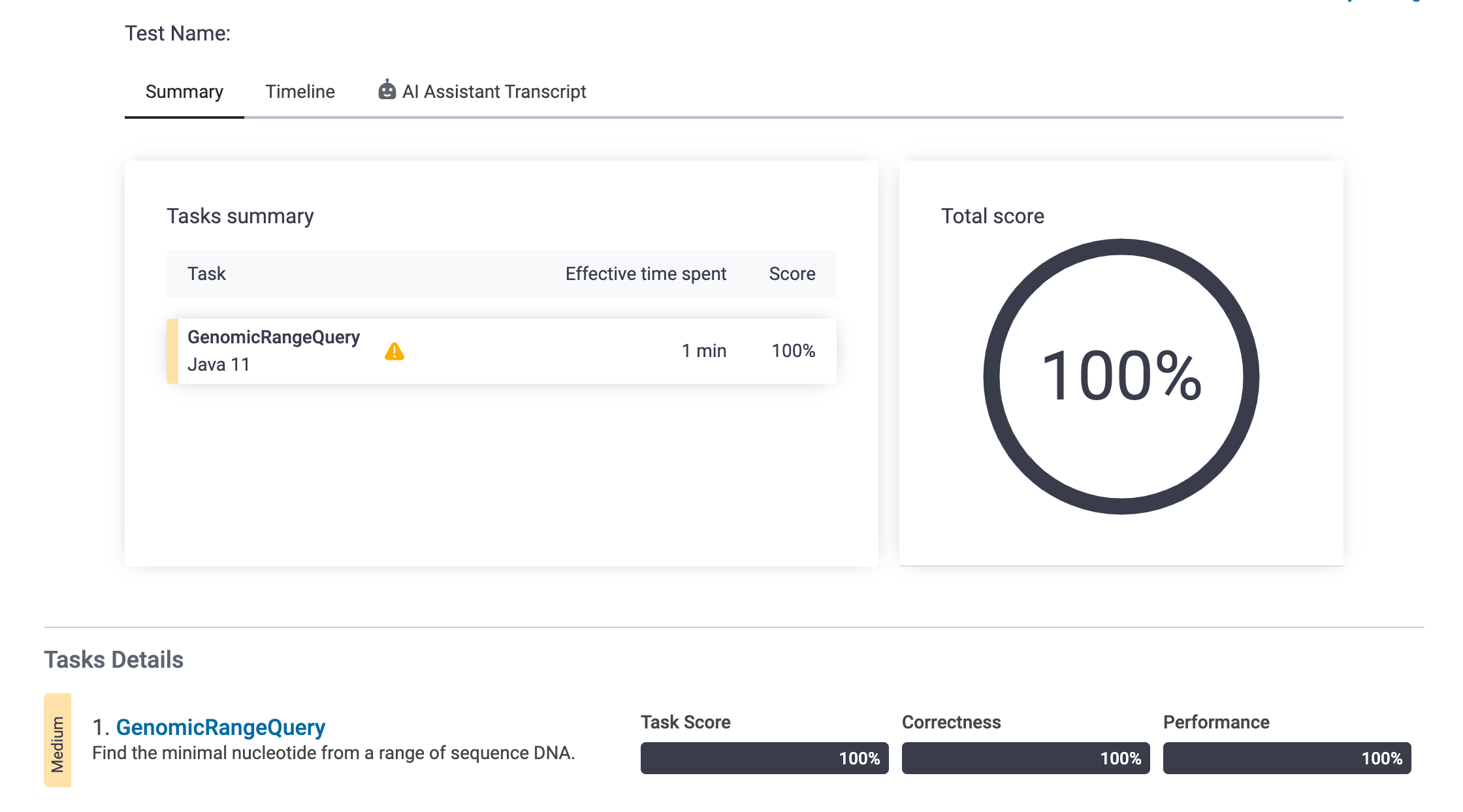
문제풀어보기-> https://app.codility.com/programmers/lessons/5-prefix_sums/genomic_range_query/
