
Logistic_Regression
Logistic Regression이란?
Logistic Regression은 선형 회귀의 확장으로, 주로 이진 분류 문제에 사용됩니다. 이 알고리즘은 데이터의 선형 결합을 확률로 변환하여, 이를 통해 어떤 클래스에 속할 확률을 예측합니다. 예측된 확률을 기반으로 0.5보다 크면 양성 클래스, 0.5보다 작으면 음성 클래스로 분류합니다.
import numpy as np
z = np.arange(-10, 10, 0.01)
g = 1 / (1 + np.exp(-z))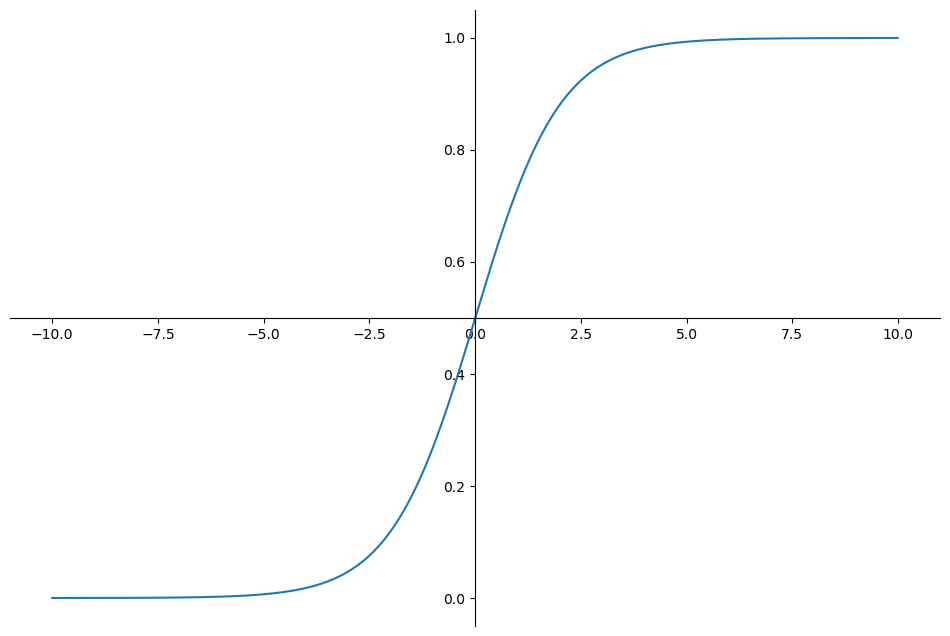
시그모이드 함수
와인분류
Logistic_Regression을 사용하여 와인 맛등급 분류해보기
wine['taste'] = [1 if grade > 5 else 0 for grade in wine['quality']]
X = wine.drop(['quality', 'taste'], axis=1)
y = wine['taste']grade를 기준으로 5보다 높으면 1 낮으면 0으로 정의해줍니다.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=13)이후 테스트 세트와 훈련 세트로 분류
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score
# solver 최적화 알고리즘 선텍
# liblinear 데이터수가 적으면 기본적으로 선택
lr = LogisticRegression(solver='liblinear', random_state=13)
lr.fit(X_train, y_train)
y_pred_tr = lr.predict(X_train)
y_pred_test = lr.predict(X_test)
print('Train ACC : ', accuracy_score(y_train, y_pred_tr))
print('Test ACC : ', accuracy_score(y_test, y_pred_test))로지스틱 분류 모델을 선언하고 solver='liblinear' 파마메터를 넣어주고 정확도를 측정합니다.
Train ACC : 0.7425437752549547
Test ACC : 0.7438461538461538
이번에는 스케일링을 거친 이후에 로지스틱 회귀 모델을 만들어보겠습니다.
from sklearn.pipeline import Pipeline
from sklearn.preprocessing import StandardScaler
estimators = [('scaler', StandardScaler()),
('clf',LogisticRegression(solver='liblinear', random_state= 13))]
pipe = Pipeline(estimators)
pipe.fit(X_train, y_train)
y_pred_tr = pipe.predict(X_train)
y_pred_test = pipe.predict(X_test)
print('Train ACC : ', accuracy_score(y_train, y_pred_tr))
print('Test ACC : ', accuracy_score(y_test, y_pred_test))Train ACC : 0.7444679622859341
Test ACC : 0.7469230769230769
스케일링으로는 크게 바뀌지 않는걸 알수있다.
from sklearn.tree import DecisionTreeClassifier
wine_tree = DecisionTreeClassifier(max_depth=2, random_state=13)
wine_tree.fit(X_train, y_train)
models = {'Logistic Regression': pipe,
'desion tree': wine_tree}이번에는 DecisionTree와 Logistic 모델을 같이 사용하여 정확도를 비교해보도록하겠습니다.
from sklearn.metrics import roc_curve
plt.figure(figsize=(10,8))
plt.plot([0,1],[0,1], label='random_guess')
for model_name, model in models.items():
pred = model.predict_proba(X_test)[:,1]
fpr, tpr, thesholds = roc_curve(y_test, pred)
plt.plot(fpr, tpr, label=model_name)
plt.grid()
plt.legend()
plt.show()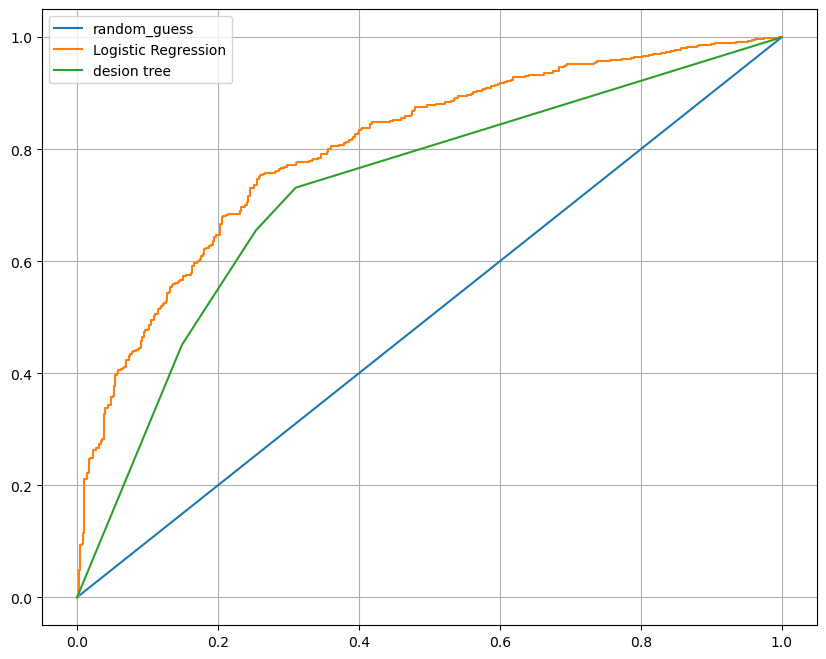
roc curve를 그려 직관적으로 비교해보면 DecisionTree 모델보다 로지스틱 회귀 모델이 더 정확도가 높음을 알수있다.
당뇨병 예측
이번에는 로지스틱 회귀모델로 당뇨병 여부를 예측하는 모델을 만들어 보도록하겠습니다
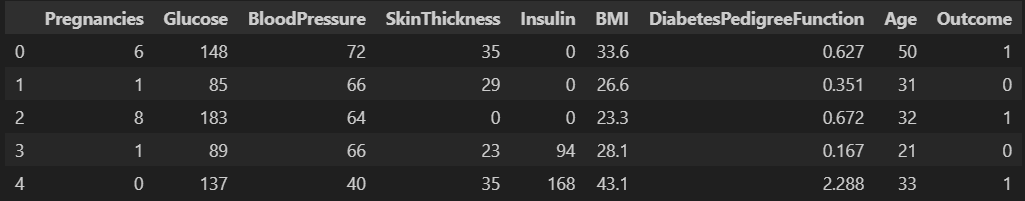
데이터 세트의 정보
Pregnancies (임신 횟수): 해당 개체의 임신 횟수입니다.
Glucose (혈장 포도당 농도): 2시간 동안 경과된 경구 포도당 내성 검사 후 혈장 포도당 농도입니다.
BloodPressure (혈압): 해당 개체의 이완기 혈압입니다.
SkinThickness (피부 두께): Triceps skinfold thickness로 측정된 값입니다.
Insulin (인슐린): 2시간 동안 경과된 경구 포도당 내성 검사 후 혈청 인슐린 농도입니다.
BMI (체질량 지수): 체중(kg)을 키(m)의 제곱으로 나눈 값으로, 비만도를 측정하는 지표입니다.
DiabetesPedigreeFunction (당뇨병 혈통 기능): 가족력을 고려하여 당뇨병이 발병할 확률을 계산하는데 사용되는 값입니다.
Age (나이): 해당 개체의 나이입니다.
Outcome (결과): 당뇨병 여부를 나타내는 이진 변수입니다. 1은 당뇨병이 발병했음을 나타내고, 0은 발병하지 않았음을 나타냅니다.
히트맵으로 상관관계 비교하기
import seaborn as sns
plt.figure(figsize=(12,8))
sns.heatmap(pima.corr(), cmap='YlGnBu')
plt.show()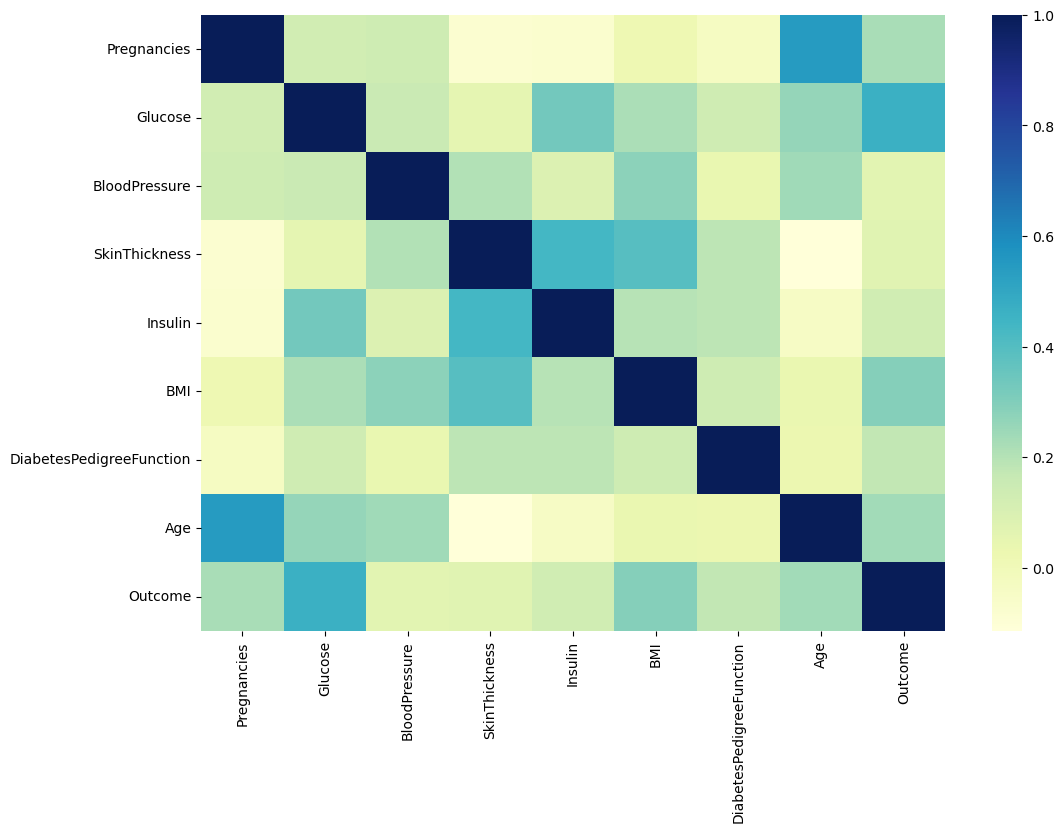
0인 데이터가 있는 컬럼을 조회
(pima==0).astype(int).sum()Pregnancies 111
Glucose 5
BloodPressure 35
SkinThickness 227
Insulin 374
BMI 11
DiabetesPedigreeFunction 0
Age 0
Outcome 500
dtype: int64
당뇨병 예측에 필요한 핵심 컬럼중에서 0인 값들에 평균값들로 채워줍니다.
zero_features = ['Glucose', 'BloodPressure', 'SkinThickness', 'BMI']
pima[zero_features] = pima[zero_features].replace(0, pima[zero_features].mean())
(pima==0).astype(int).sum()이후 학습세트와 훈련세트로 분류해줍니다.
X = pima.drop('Outcome', axis=1)
y = pima['Outcome']
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.2, random_state=13, stratify= y)스케일링과 모델을 선언하여 파이프를 만들어줍니다.
estimators = [('scaler', StandardScaler()),
('clf', LogisticRegression(solver='liblinear', random_state=13))]
pipe_lr = Pipeline(estimators)
pipe_lr.fit(X_train, y_train)
pred = pipe_lr.predict(X_test)이후 만든 모델로 정확도를 측정해줍니다
from sklearn.metrics import (accuracy_score, recall_score, precision_score, roc_auc_score, f1_score)
print('Accuracy', accuracy_score(y_test, pred))
print('Recall', recall_score(y_test, pred))
print('Precision', precision_score(y_test, pred))
print('Auc_score', roc_auc_score(y_test, pred))
print('F1 score', f1_score(y_test, pred))Accuracy 0.7727272727272727
Recall 0.6111111111111112
Precision 0.7021276595744681
Auc_score 0.7355555555555556
F1 score 0.6534653465346535
중요도 측정
coeff = list(pipe_lr['clf'].coef_[0])
labels = list(X_train.columns)
features = pd.DataFrame({'features':labels, 'importance':coeff})
features.sort_values(by=['importance'], ascending=True, inplace=True)상관관계의 데이터를 coeff 리스트에 넣고 크기순으로 정렬해줍니다.
features['positive'] = features['importance'] > 0
features.set_index('features', inplace=True)
features이후에 해당데이터를 0보다 큰지 작은지로 분류하여 컬럼과의 새로운 컬럼을 만들어주고 시각화 해주면
features['importance'].plot(kind='barh',
figsize=(11,6),
color=features['positive'].map({True:'blue',
False:'red'}))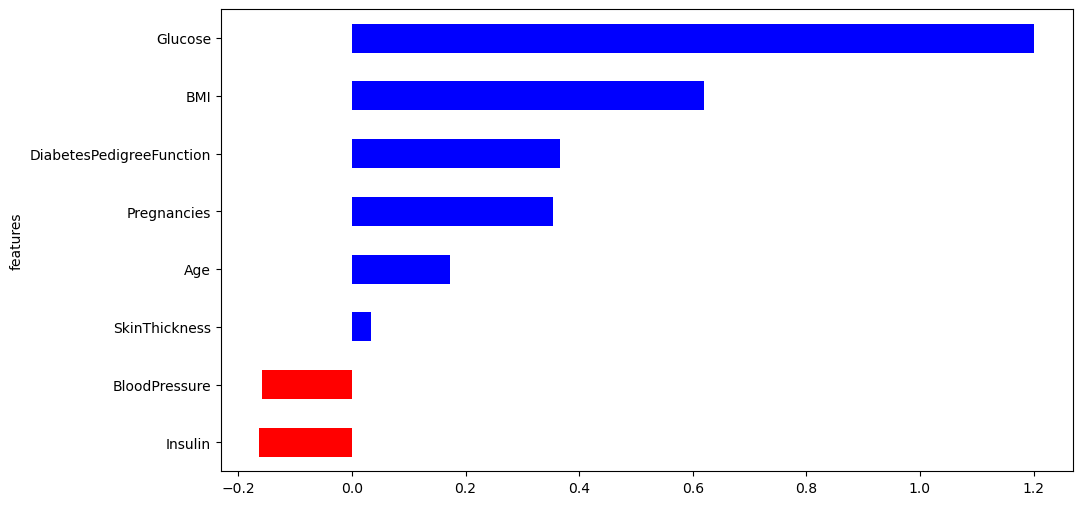
다음 결과를 보면 혈장 포도당 농도와 BMI가 당뇨와 관계가 깊다는 결론을 얻을수 있었습니다.
precision과 recall
이어서 와인 데이터를 이용해서 모델의 성능을 종합적으로 평가해보겠습니다.
from sklearn.metrics import classification_report
print(classification_report(y_test, lr.predict(X_test)))여기서 각 행은 클래스에 대한 정보를 나타냅니다. 각 열은 다음과 같은 의미를 가집니다:
precision: 해당 클래스를 양성으로 예측한 샘플 중 실제로 양성인 비율 (정밀도)
recall: 해당 클래스의 실제 양성 중 모델이 양성으로 예측한 비율 (재현율)
f1-score: 정밀도와 재현율의 조화 평균
support: 해당 클래스의 실제 샘플 수
macro avg
(0.68 + 0.77) / 2
weighted avg
0.68 (477/1300) + 0.77 (823/1300)
from sklearn.metrics import confusion_matrix
confusion_matrix(y_test, lr.predict(X_test))[275, 202],
[131, 692]
confusion_matrix는
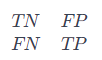
True Negative (TN): 실제 음성이며 모델이 정확하게 음성으로 예측한 경우
False Positive (FP): 실제 음성이지만 모델이 잘못해서 양성으로 예측한 경우 (오류 유형 I)
False Negative (FN): 실제 양성이지만 모델이 잘못해서 음성으로 예측한 경우 (오류 유형 II)
True Positive (TP): 실제 양성이며 모델이 정확하게 양성으로 예측한 경우
를 시각화 해줍니다.
thresholds값에 따라서 recalls, precision의 변화를 시각화 해보겠습니다.
import matplotlib.pyplot as plt
from sklearn.metrics import precision_recall_curve
%matplotlib inline
plt.figure(figsize=(10,8))
pred = lr.predict_proba(X_test)[:, 1]
precisionm, recalls, thresholds = precision_recall_curve(y_test, pred)
plt.plot(thresholds, precisionm[:len(thresholds)], label = 'precision')
plt.plot(thresholds, recalls[:len(thresholds)], label = 'recall')
plt.grid()
plt.legend()
plt.show() 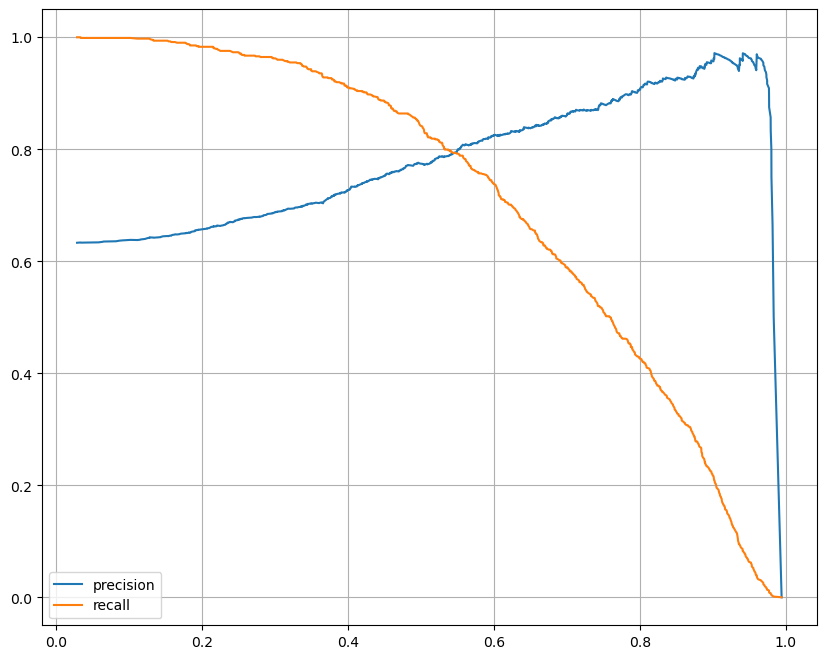
pred_proba = lr.predict_proba(X_test)
pred_proba[0.40526731, 0.59473269],
[0.50957556, 0.49042444],
[0.10215001, 0.89784999],
...,
[0.22540242, 0.77459758],
[0.67366935, 0.32633065],
[0.31452992, 0.68547008]
threshold 바꿔보기
from sklearn.preprocessing import Binarizer
binarizer = Binarizer(threshold=0.6).fit(pred_proba)
pred_bin = binarizer.transform(pred_proba)[:,1]
print(classification_report(y_test, lr.predict(X_test)))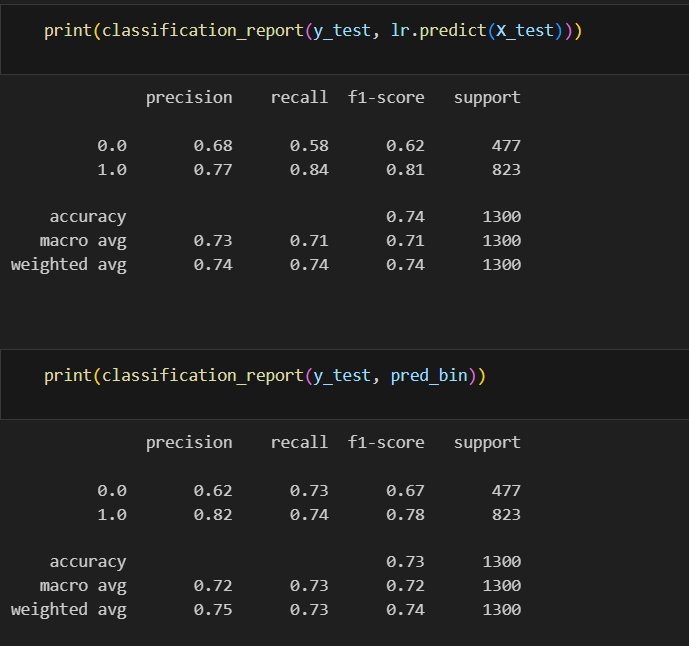
threshold값에 따른 결과값 확인하기
solver 파라메터
solver='liblinear'는 로지스틱 회귀의 최적화 문제를 해결하는 데 사용되는 알고리즘을 지정하는 매개변수입니다. liblinear는 작은 데이터셋에서 잘 동작하며 이진 분류 문제에 특히 효과적입니다.
