1. 문제
위스콘신 유방암 데이터셋을 대상으로 분류기법 2 개를 적용하여 기법별 결과를 비교하고 시각화하시오. (R 과 python 버전으로 모두 실행)
-종속변수는 diagnosis: Benign(양성), Malignancy(악성)
2. 의사결정나무 문제풀이
2.1 풀이 과정(R)
데이터 준비
-> factor 형을 numeric 형으로 변환
-> 학습, 테스트 데이터 생성
-> 의사결정나무 rpart 활용하여 생성
-> 시각화
# (분류분석/시각화)
# 2. 위스콘신 유방암 데이터셋을 대상으로 분류기법 2 개를 적용하여 기법별 결과를
# 비교하고 시각화하시오. (R 과 python 버전으로 모두 실행)
# -종속변수는 diagnosis: Benign(양성), Malignancy(악성)
library(car)
library(lmtest)
library(ROCR)
library(SyncRNG)
am <- read.csv("wdbc_data.csv") # diagnosis 변수를 1, 0 으로 변경
am$diagnosis_sub <-ifelse(am$diagnosis == 'M', 1, 0)
# diagnosis 변수를 숫자로 변경했으니 필요없는 변수는 제거
am <- subset(am, select = -c(diagnosis, id))
str(am)
# 학습셋, 테스트셋 생성 v <- 1:nrow(am)
s <- SyncRNG(seed=42) s$shuffle(v)[1:5]
idx <- s$shuffle(v)[1:round(nrow(am)*0.7)] head(idx)
length(idx)
am$diagnosis_sub <- as.factor(am$diagnosis_sub)
head(am[-idx[1:length(idx)],]) idx[1:length(idx)]
trData <- am[idx[1:length(idx)],] teData <- am[-idx[1:length(idx)],]
#============ 의사결정나무 ============#
library(rpart)
library(caret)
data_lm <- rpart(diagnosis_sub~., data = trData) data_lm
rpart_lm <-predict(data_lm, newdata = teData, type = 'class')
table(teData$diagnosis_sub, rpart_lm)
teData$diagnosis_sub <- as.factor(teData$diagnosis_sub)
confusionMatrix(rpart_lm, teData$diagnosis_sub)
# Accuracy : 0.924
# 시각화
library(rpart.plot)
rpart.plot(data_lm)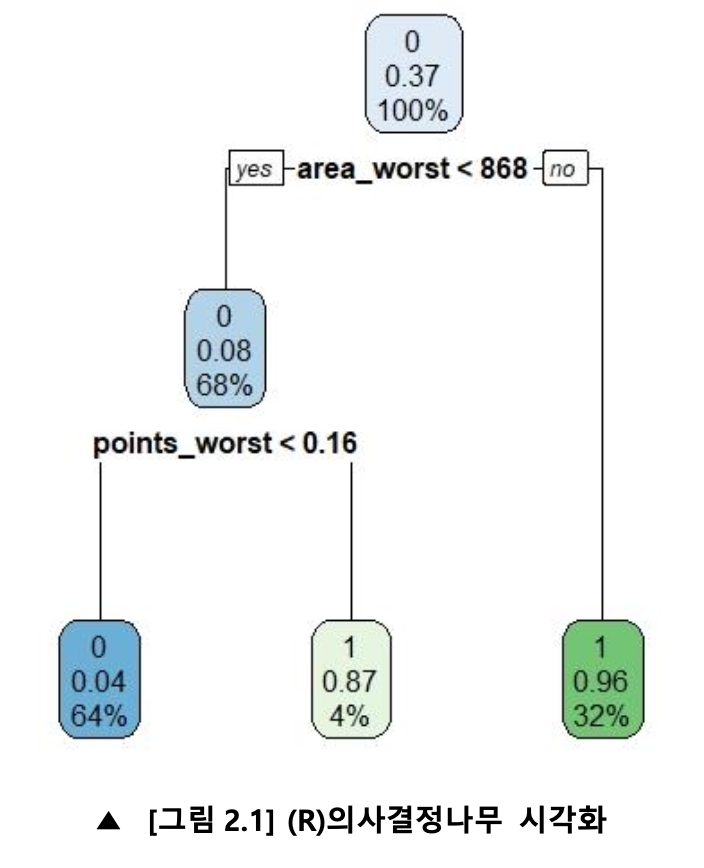
2.2 풀이 과정(python)
데이터 준비
-> 만들어진 데이터 확인 후 결측값 확인
-> 문자형태 값 → 숫자 형태로 변환 (1,0)
-> 값을 새로운 컬럼’진단’에 담아준다
-> 필요 없는 컬럼 삭제
-> 학습, 테스트 데이터 생성
-> 의사결정나무 모델 만들기
-> 예측
-> 시각화
# 필요한 모듈 임포트
import sklearn.datasets as d
import pandas as pd
import numpy as np
import matplotlib as mpl
import matplotlib.pyplot as plt
from sklearn import metrics
from sklearn.model_selection import cross_val_score
from SyncRNG import SyncRNG
from sklearn.tree import DecisionTreeClassifier
from sklearn.tree import DecisionTreeRegressor,plot_tree from sklearn.tree import export_graphviz
#데이터 셋 로드
wdbccancer = pd.read_csv("wdbc_data.csv")
# 결측값 확인 wdbccancer.isnull().sum()
# 문자형태인 타기 값을 숫자 형태로 변환 wdbccancer['diagnosis'].unique()
#M=1(악성종양,암),B=0 으로 변환
def func1(row):
if 'M' in row :
return 1
else :
return 0
# 변환된 결과를 데이터프레임에 'diagnosis' 이라는 새로운 컬럼을 만들어 넣어준다
wdbccancer['diagnosis'] = wdbccancer['diagnosis'].apply(func1)
wdbccancer.head()
# 필요없는 컬럼 삭제
wdbc = wdbccancer.drop(columns=['id']) wdbc.head()
# 분류보델 구축 - 결정트리
from sklearn.tree import DecisionTreeClassifier
#데이터 셋 7:3으로 분할 v=list(range(1,len(wdbccancer)+1)) s=SyncRNG(seed=42) s.shuffle(v)[:5]
ord=s.shuffle(v) idx=ord[:round(len(wdbccancer)*0.7)] idx[:5]
# 인덱스 수정-R 이랑 같은 데이터 가져오기 위해
for i in range(0,len(idx)):
idx[i]=idx[i]-1
# 학습데이터, 테스트데이터 생성
train=wdbc.loc[idx] # 70% train=train.sort_index(ascending=True) test=wdbc.drop(idx) # 30%
train_x=train.drop(['diagnosis'],axis=1) train_y=train['diagnosis']
test_x=test.drop(['diagnosis'],axis=1) test_y=test['diagnosis']
#============ 의사결정나무 ============#
# 의사결정나무(Decision Tree) 객체 생성 및 훈련
# 모델만들기
reg = DecisionTreeRegressor(max_depth=2).fit(train_x,train_y)
#예측
pred=reg.predict(test_x)
p1=pred.round()
p2=np.array(test_y)
# 정확도
import sklearn.metrics as metrics metrics.accuracy_score(p2,p1)
#변수 중요도
for i,col in enumerate(train_x.columns):
print(f'{col} 중요도 : {reg.feature_importances_[i]}')
#시각화하기
fig = plt.Figure(facecolor="white") plot_tree(reg,feature_names=train_x.columns,filled=True) plt.show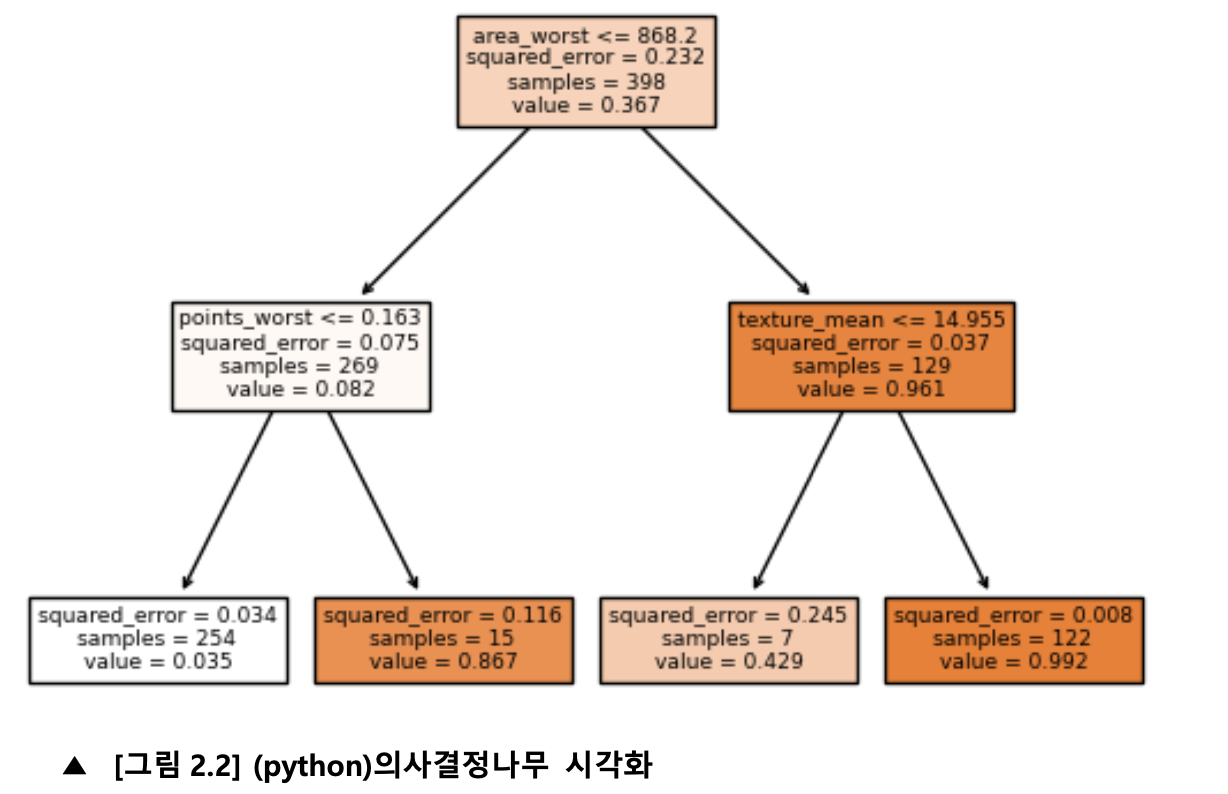
3. SVM 문제풀이
3.1 풀이 과정(R)
데이터준비
-> linear 커널을 사용하여 튜닝
-> svm 실행
-> 예측 및 정확도 확인
-> 시각화
#============ SVM ============#
library(e1071)
library(caret)
# linear 커널 사용 튜닝
result1 <- tune.svm(diagnosis_sub~., data=trData, gamma=2^(-5:0), cost = 2^(0:4),
kernel="linear") # 튜닝된 파라미터 확인
result1$best.parameters
# SVM 실행
normal_svm1 <- svm(diagnosis_sub~., data=trData, gamma=0.0625, cost=1, kernel = "linear")
# 결과 확인 summary(normal_svm1)
# sv index 확인 normal_svm1$index
#SVM으로 예측
normal_svm1_predict <- predict(normal_svm1, teData)
# 테이블 확인
table(teData$diagnosis_sub, normal_svm1_predict)
# 정확도
confusionMatrix(normal_svm1_predict, teData$diagnosis_sub) # Accuracy : 0.9825
# 시각화
library(dplyr)
x11()
am %>%
ggplot(aes(x=radius_mean ,y=texture_mean,colour=diagnosis_sub)) + geom_point() +
theme_minimal(base_family="NanumGothic")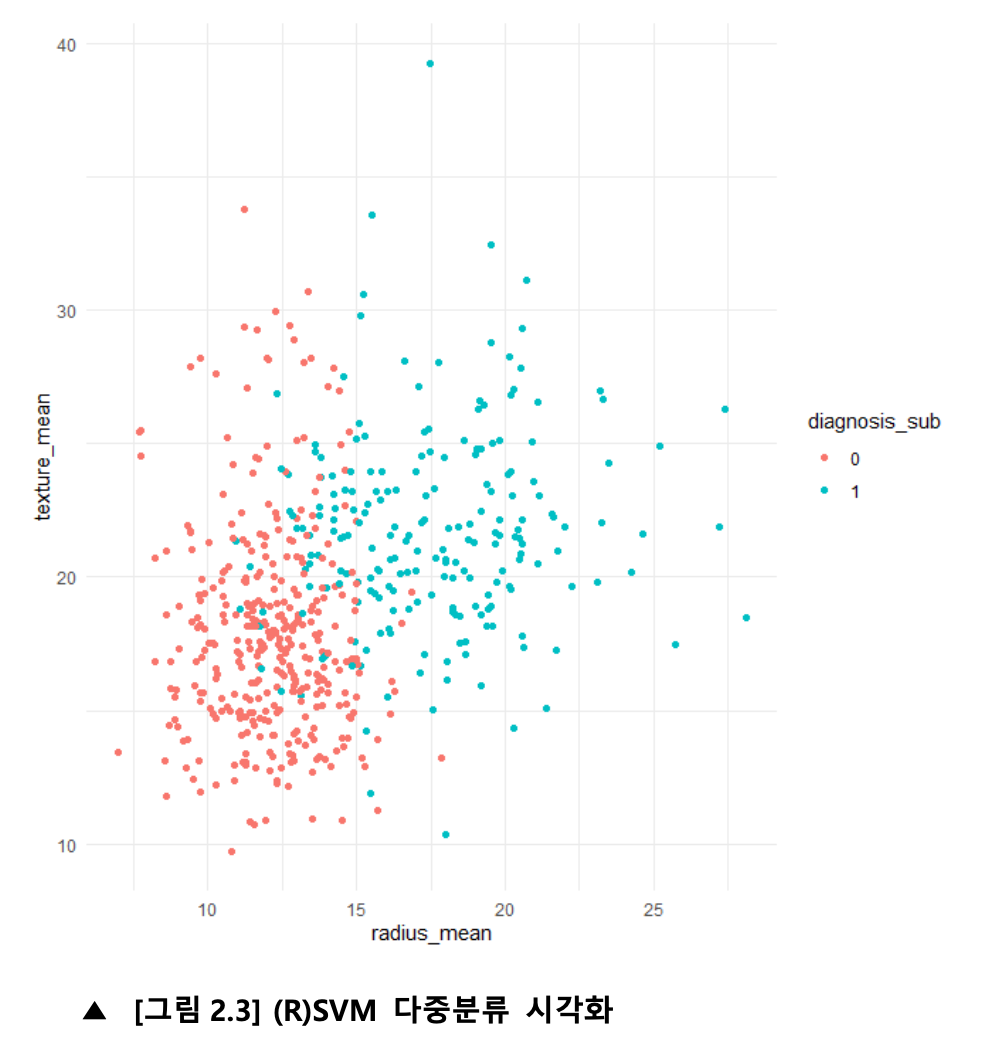
3.2 풀이 과정(python)
데이터 준비
-> Kernel 을 linear & rbf 로 선형분리
-> 교차 검증, 높은 평균을 가진 SCORE 을 선택
-> 학습, 테스트 데이터 생성 토대로 시각화
# SVM ======================================================
from sklearn.svm import LinearSVC
from sklearn.svm import SVC
import sklearn.svm as svm
import sklearn.metrics as mt
from sklearn.model_selection
import cross_val_score, cross_validate
#데이터 셋 로드
wdbccancer_svm = pd.read_csv("wdbc_data.csv") wdbccancer_svm
# 아이디는 유방암을 예측하는데 전혀 도움이 되지 않기 때문에 무시
# diagnosis M = 1 (악성종양,암), B = 0
# 결측값 확인 wdbccancer_svm.isnull().sum()
# 문자형태인 타기 값을 숫자 형태로 변환
wdbccancer_svm['diagnosis'].unique() #M=1(악성종양,암),B=0 으로 변환
def func1(row):
if 'M' in row :
return 1
else :
return 0
# 변환된 결과를 데이터프레임에 '진단' 이라는 새로운 컬럼을 만들어 넣어준다
wdbccancer_svm['진단'] = wdbccancer_svm['diagnosis'].apply(func1)
wdbccancer_svm.head()
# 필요없는 컬럼 삭제
wdbc = wdbccancer_svm.drop(columns=['id','diagnosis'])
wdbc.head()
# 분류보델 구축 - 결정트리
from sklearn.tree import DecisionTreeClassifier
# 훈련 검증용 데이터 분리
from sklearn.model_selection import train_test_split
#데이터 셋 7:3으로 분할
X = wdbc.drop(['진단'], axis = 1) # 목표변수
y = wdbc['진단'] # 설정변수
X_train,X_test,y_train,y_test = train_test_split (X,y, test_size = 0.3 , random_state = 1004)
# test_size: 테스트 셋 구성의 비율
# SVM, kernel = linear 로 선형분리
svm_model = svm.SVC(kernel='linear', random_state=100)
# 교차검증
scores = cross_val_score(svm_model,X,y,cv=5) scores
pd.DataFrame(cross_validate(svm_model,X,y,cv=5)) print("교차검증 평균: ", scores.mean())
# 0.9508461419034312
# SVM, kernel = 'rbf'로 비선형분리 진행 svm_model2 = svm.SVC(kernel='rbf')
# 교차검증
scores2 = cross_val_score(svm_model2, X, y, cv=5) scores2
pd.DataFrame(cross_validate(svm_model2, X, y, cv=5)) print('교차검증 평균: ', scores2.mean())
# 0.9156652693681104
######### 선형 분리에서 높은 평균 SCORE , 선형분리가 적합한 케이스이다.
import matplotlib.pyplot as plt
plt.figure(figsize=(7,7))
X1=np.array(X)
y1=np.array(y)
plt.scatter(X1[:,0],X1[:,1],c = y1) plt.xlabel("radius_mean")
plt.ylabel("texture_mean")
plt.show()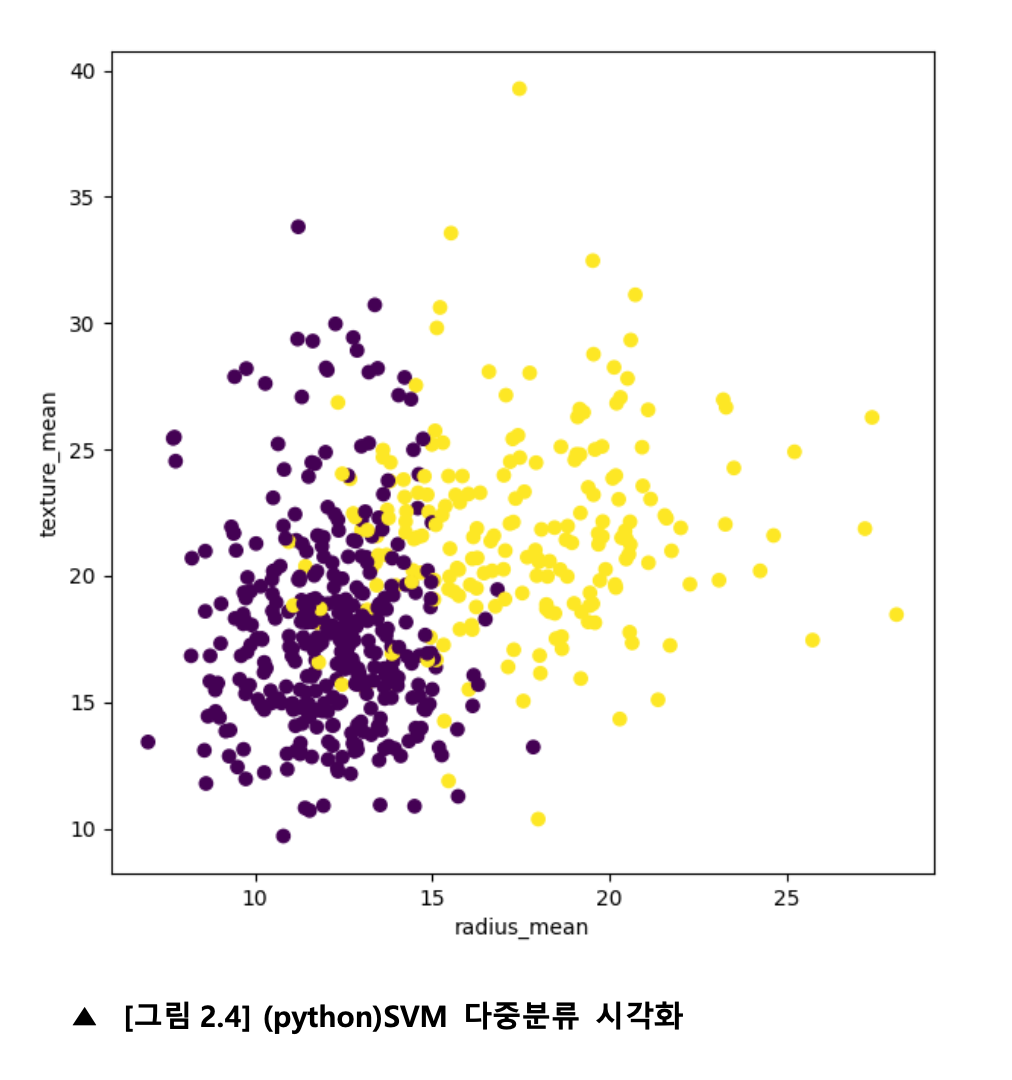
4. 결과 분석
의사결정나무와 SVM 기법을 활용하여 위스콘신 유방암 데이터셋을 분류하였다.
해당 데이터는 유방암으로 의심되는 종양의 모양에 따라 양성과 악성을 구분하는 데이터다.
종속변수 diagnosis 에 해당되는 값을 범주형 변수로 바꿔주기 위해서 B(Benign) 양성을 0 으로, M(Malignancy) 음성을 1 로 변경해주었고, 분석하기에 앞서 먼저 데이터를 R 과 python 에서 발생 난수의 순서까지 동일하지 않기 때문에 동일하게 가질 수 있도록 패키지 SyncRNG 를 사용하여 동일한 난수를 통해 분류해냈다.
R 에서는 데이터프레임이 1부터 시작하기 때문에python에서 0행과 R에서 1행이 같게 되므로 같은 인덱스 번호를 가진다면 -1 을 해주어 train 과 test 에 같은 데이터를 담아내도록 하였다.
그 결과 거의 비슷한 의사결정나무 그래프를 얻어냈음을 [그림 2.1], [그림 2.2]를 통해 확인할 수 있다.
