

답안코드
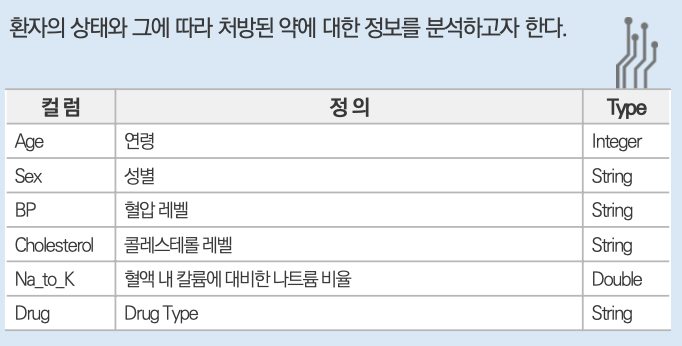
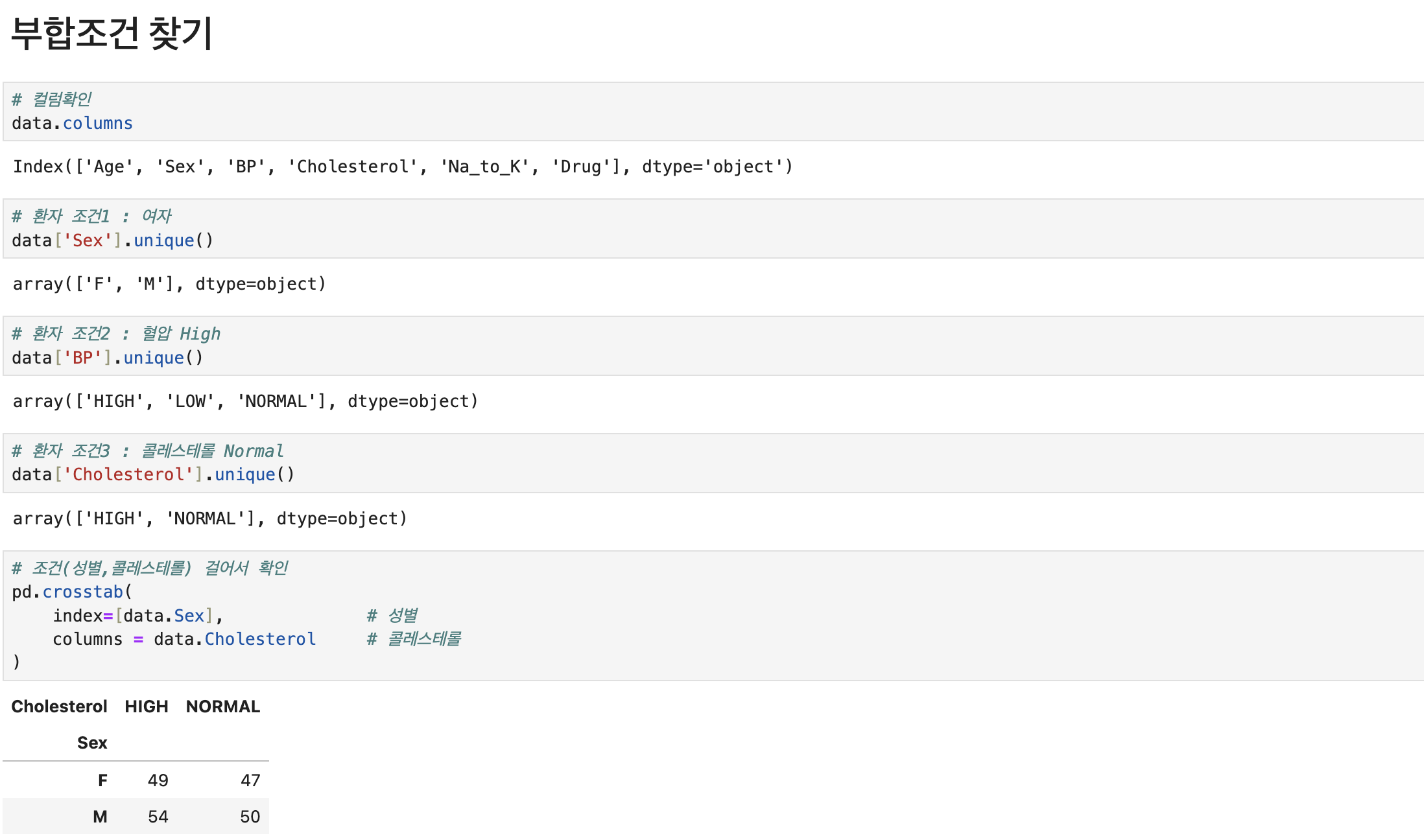
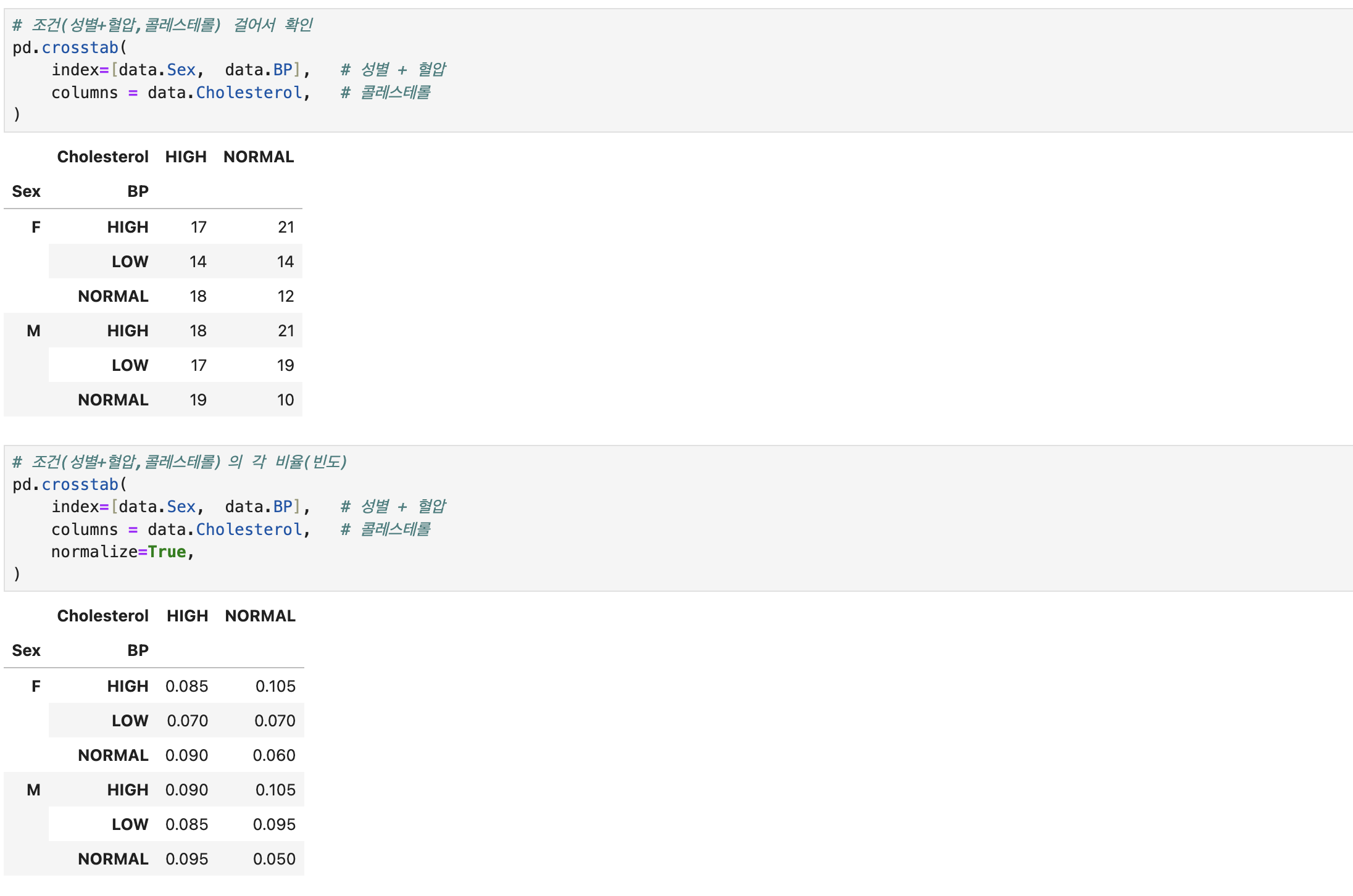
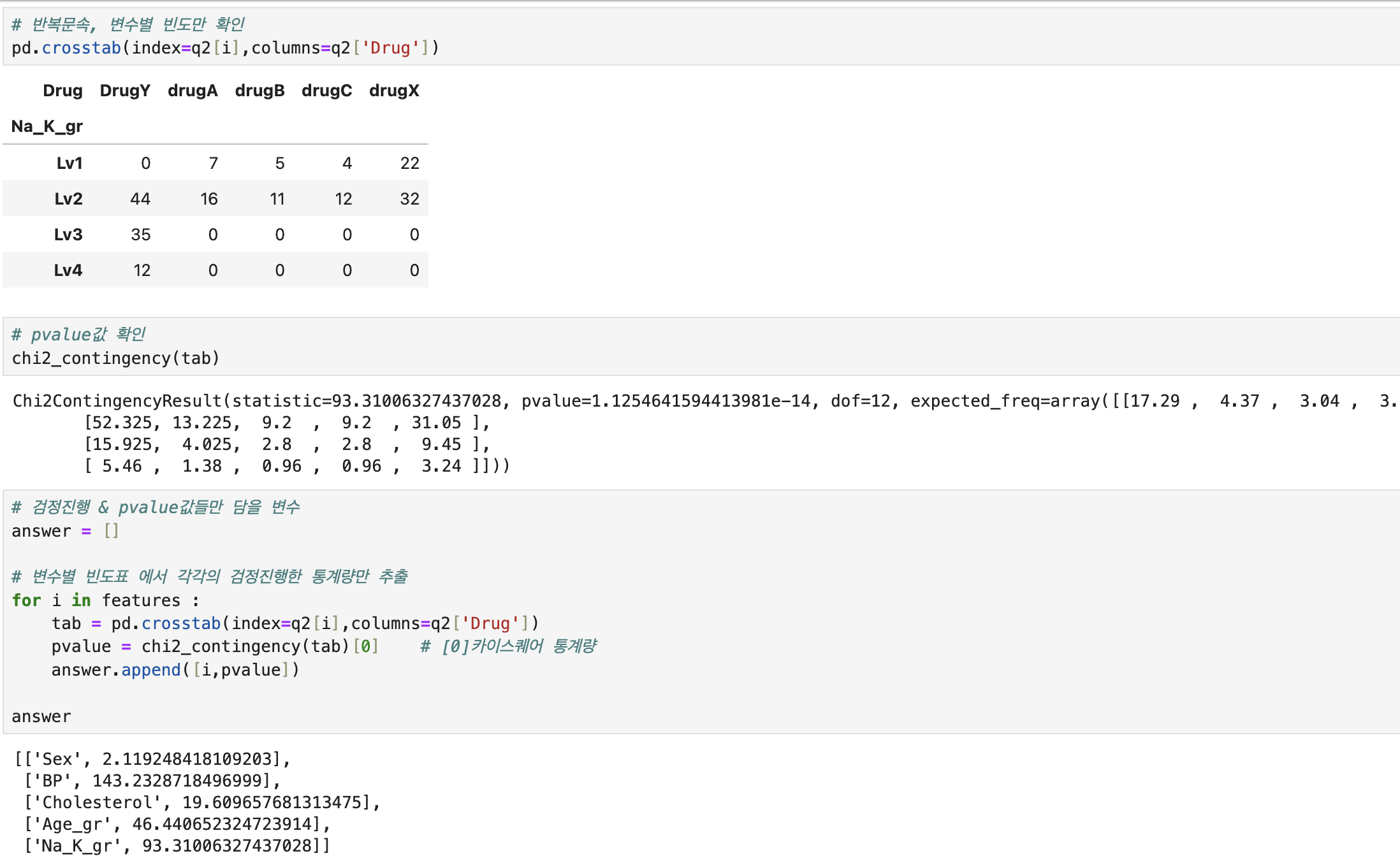
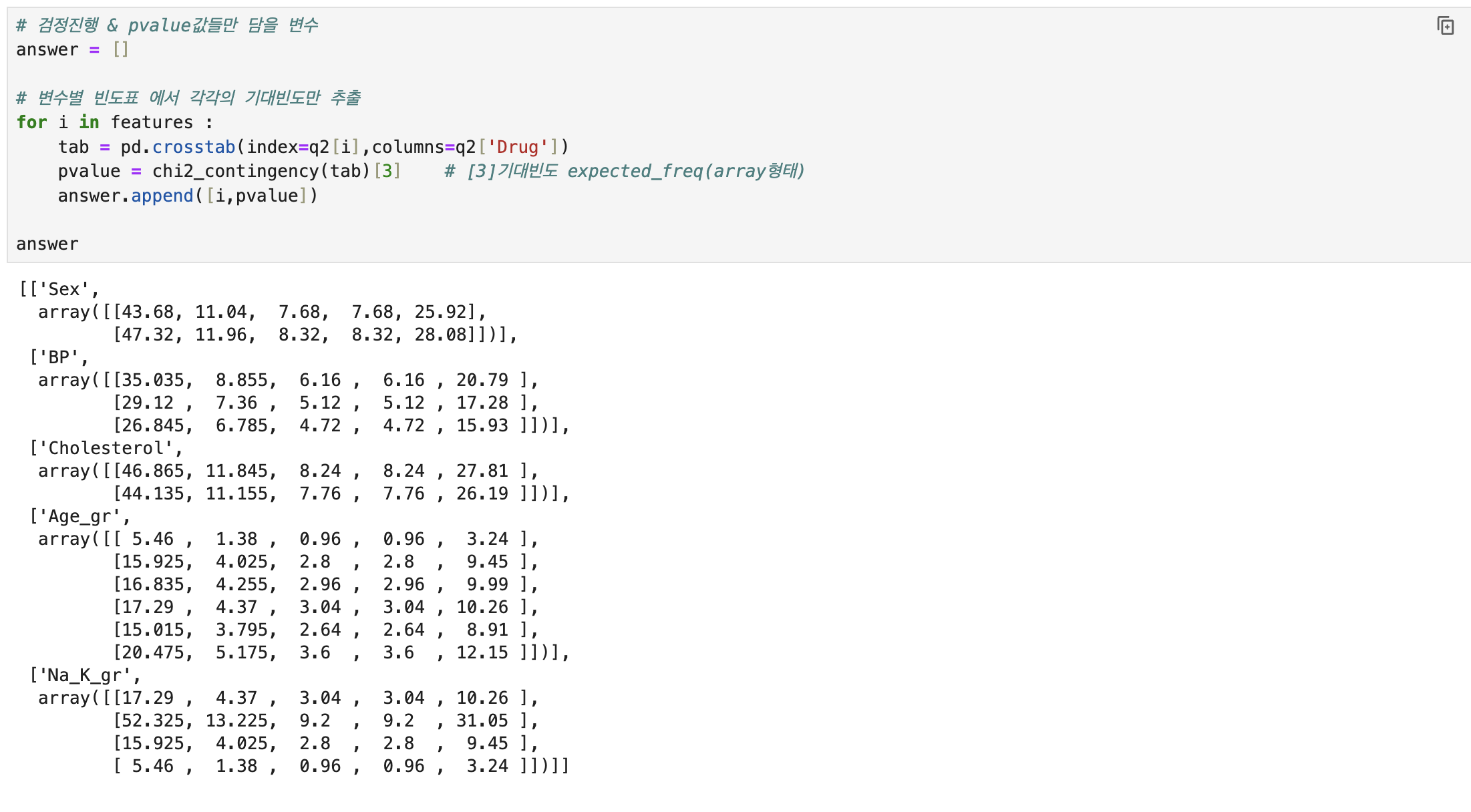
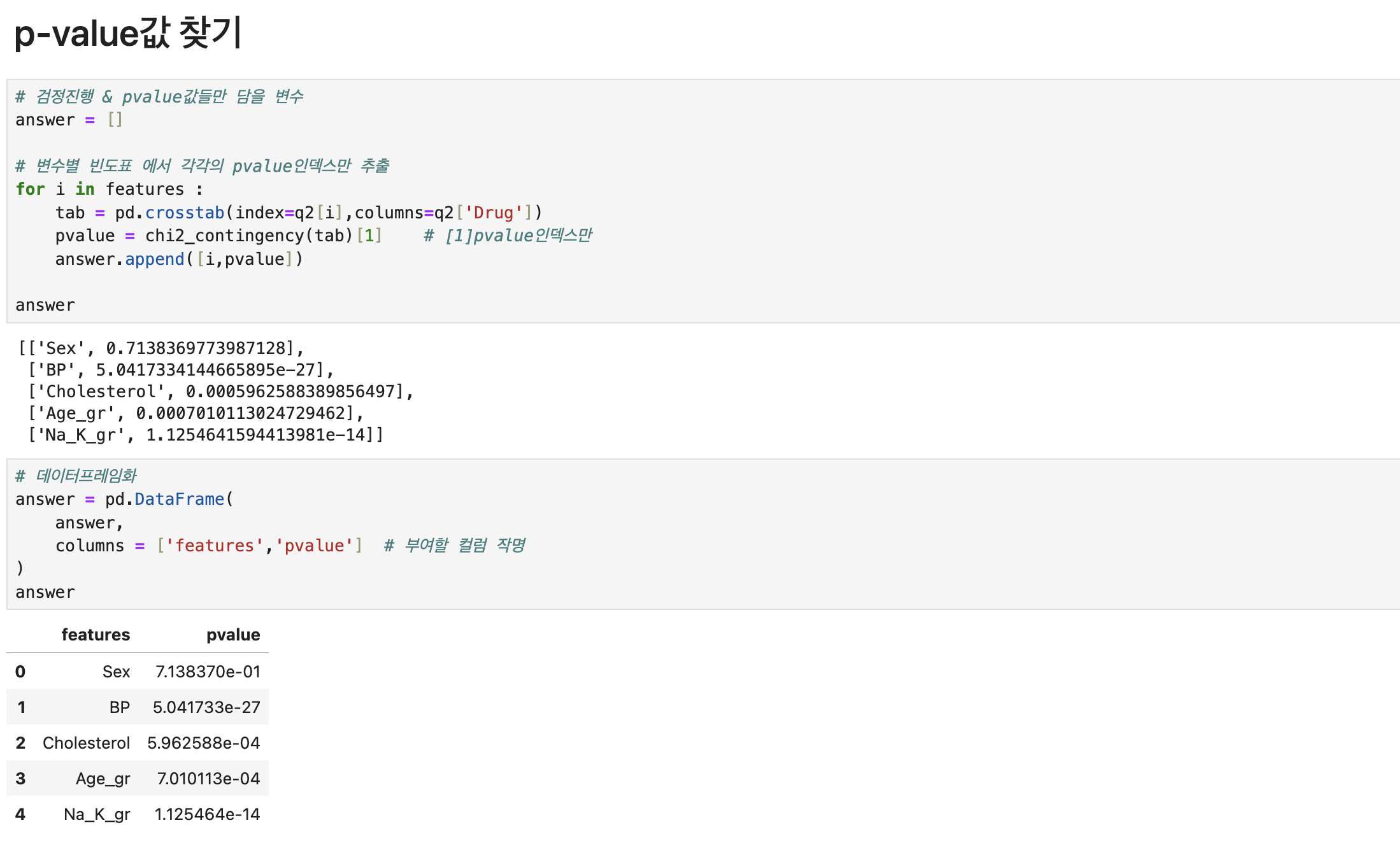
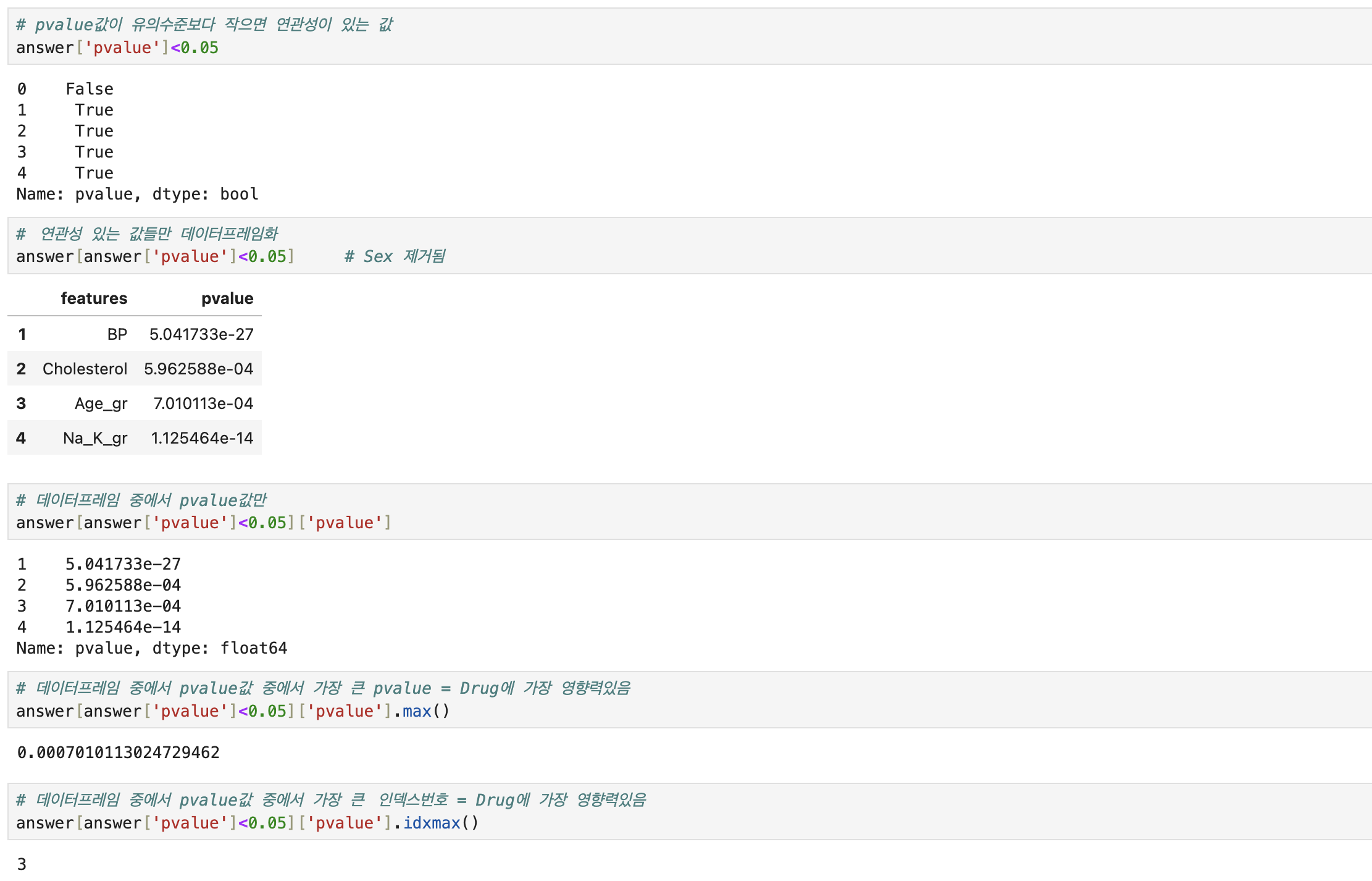
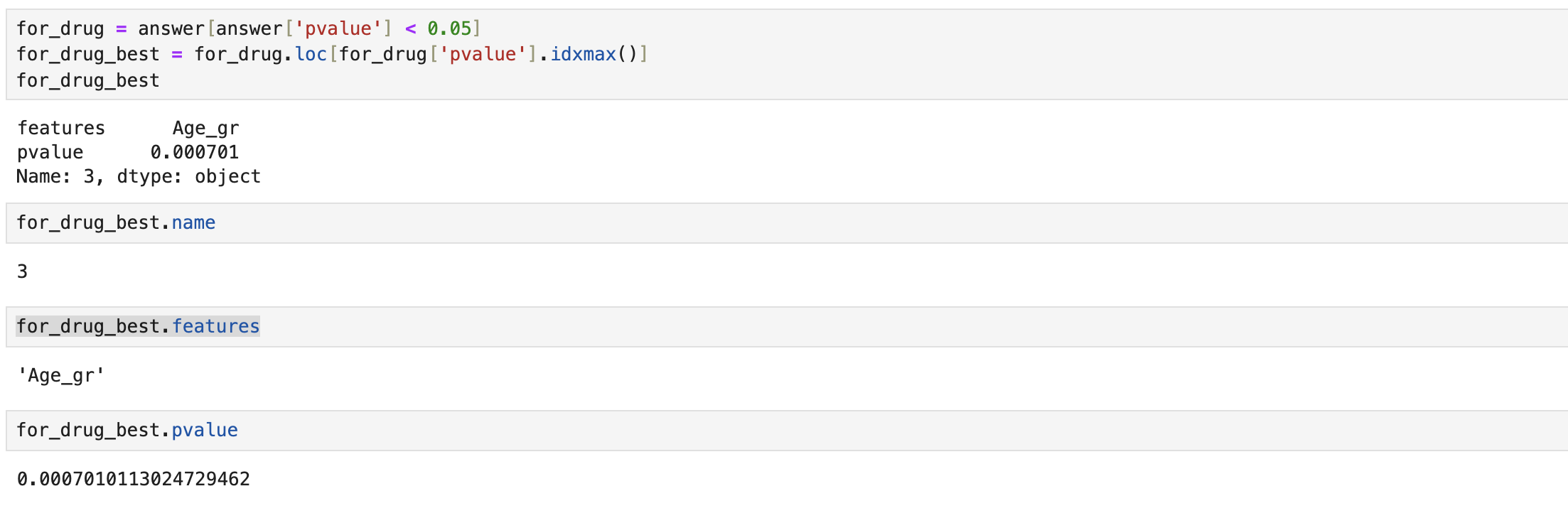
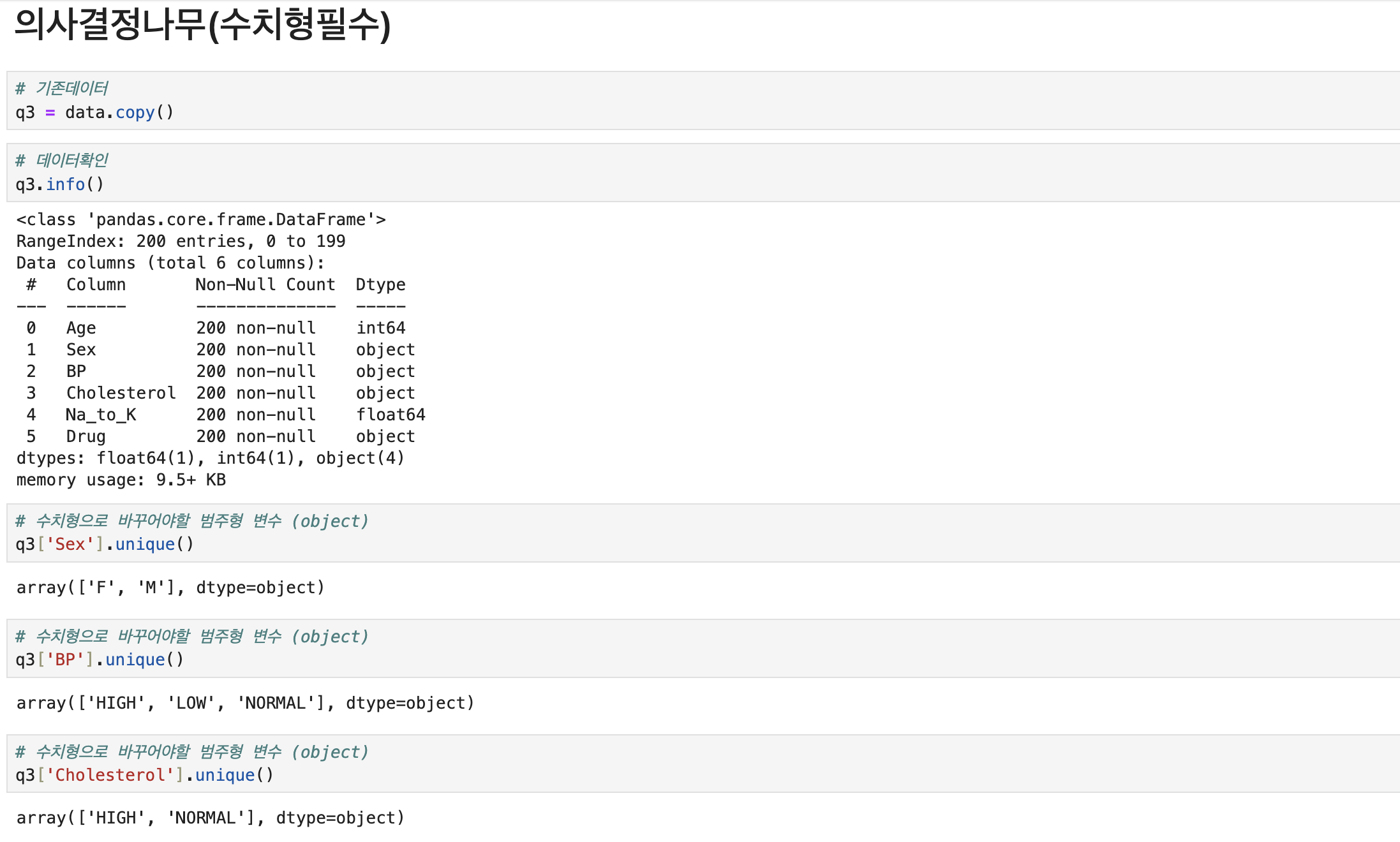
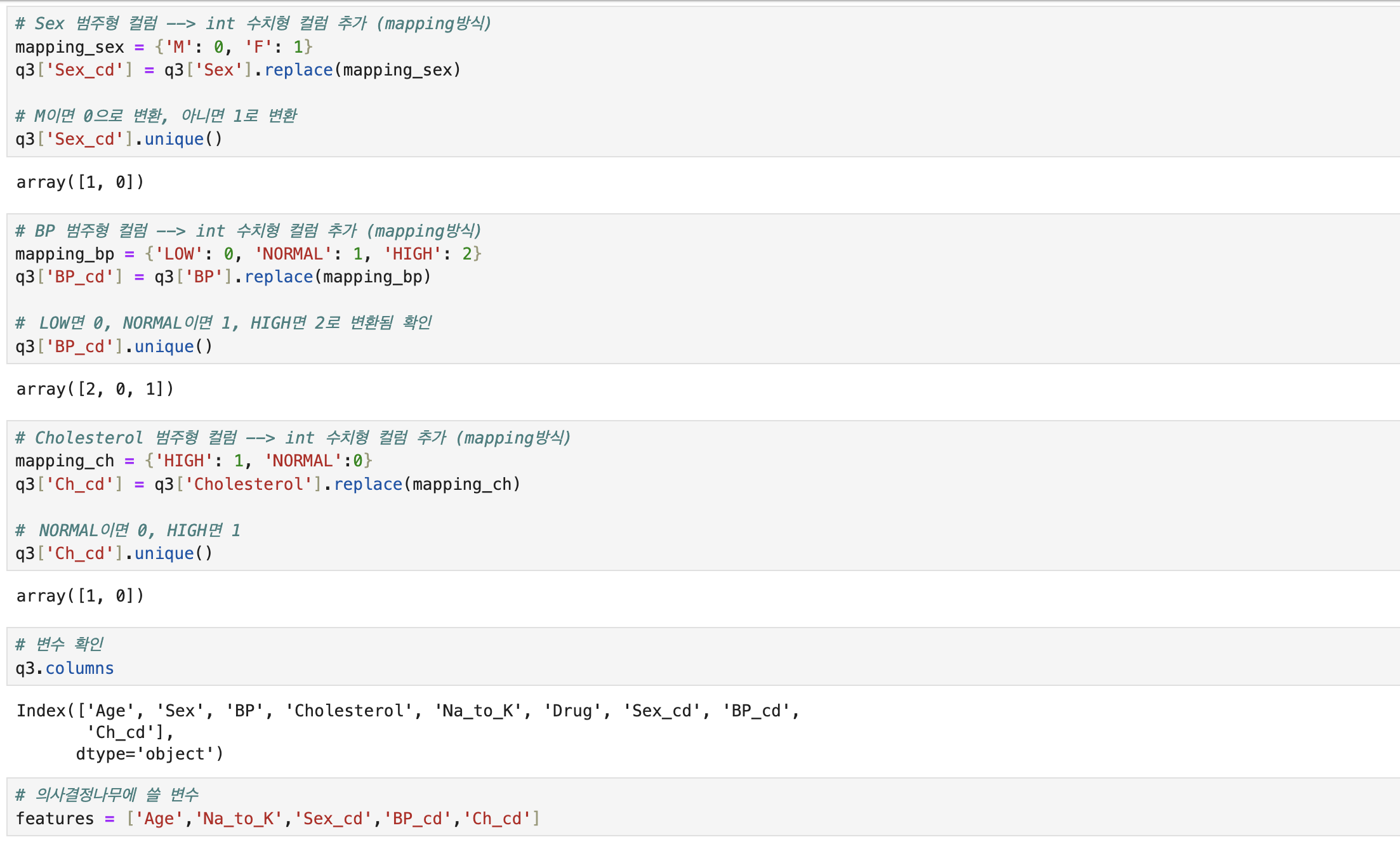
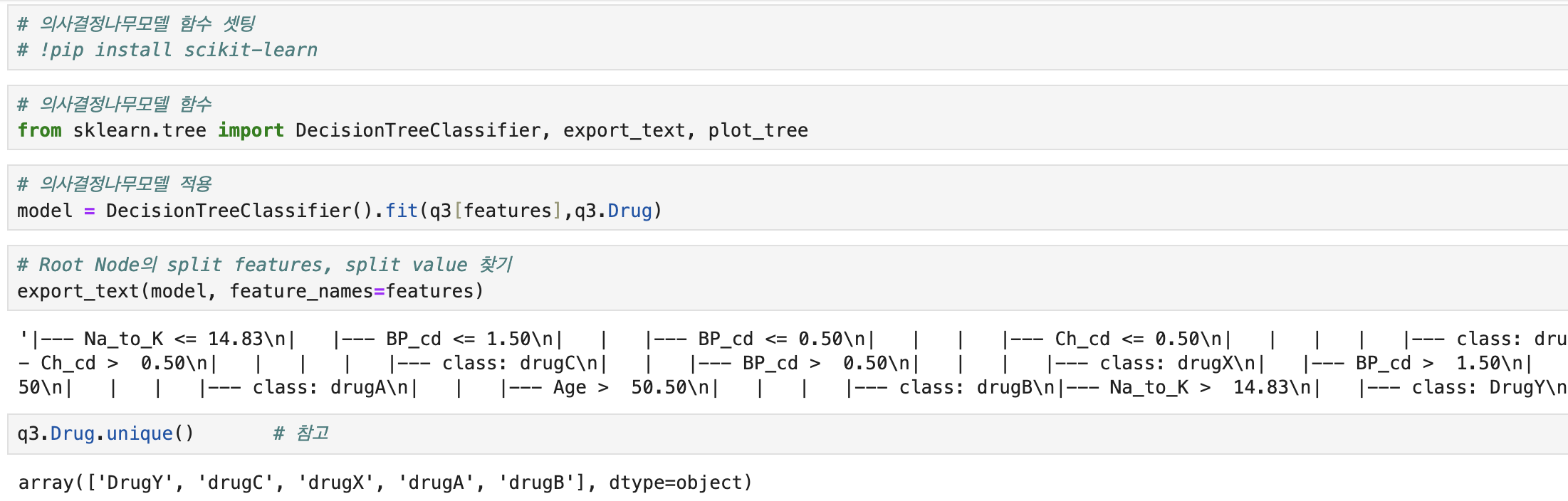
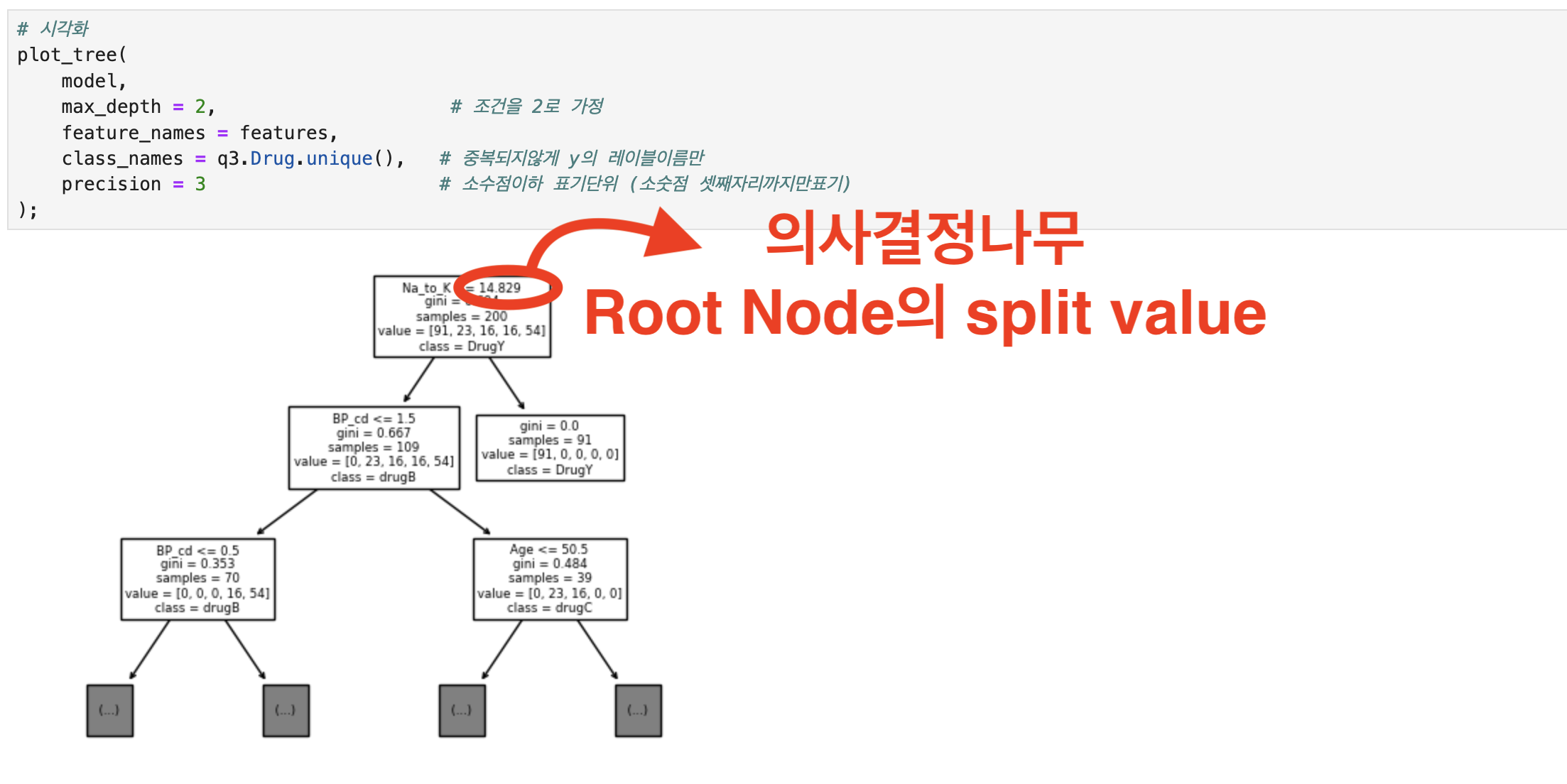
# 필요함수 import pandas as pd # 불러오기 data = pd.read_csv('./Dataset/Dataset_02.csv') # 각 조건별 환자의 전체대비 비율 q1=\ pd.crosstab( index=[data.Sex, data.BP], # 성별 + 혈압 columns = data.Cholesterol, # 콜레스테롤 normalize=True, ) # 해당 조건의 전체대비 비율 q1.loc[('F', 'HIGH'), 'NORMAL'] # 문제2를 위해 원본 복사 q2 = data.copy() # 컬럼생성(np.where절)위한 함수 import numpy as np # 수치형 컬럼--> 범주형 컬럼으로 생성 q2['Age_gr'] = np.where(q2.Age < 20 , 10 , # Age가 20미만이면 10, 아니면 np.where(q2.Age < 30 , 20 , np.where(q2.Age < 40 , 30 , np.where(q2.Age < 50 , 40 , np.where(q2.Age < 60 , 50 , 60 ))))) q2['Na_K_gr'] = np.where(q2.Na_to_K <= 10 , 'Lv1', # Na_to_k가 10이하면 'Lv1'로 np.where(q2.Na_to_K <= 20 , 'Lv2', np.where(q2.Na_to_K <= 30 , 'Lv3', 'Lv4' ))) # 범주형타입의 독립성 검정(카이스퀘어) 함수 from scipy.stats import chi2_contingency # 독립성 검정(카이스퀘어) 위한 변수x들 선택 features = ['Sex','BP','Cholesterol','Age_gr','Na_K_gr'] # 반복문 진행해서, 검정진행 & pvalue값들만 담을 변수 answer = [] # 반복문 진행해서, 변수별 빈도표 에서 각각의 pvalue인덱스만 추출 for i in features : tab = pd.crosstab(index=q2[i],columns=q2['Drug']) pvalue = chi2_contingency(tab)[1] # [1]pvalue인덱스만 answer.append([i,pvalue]) # 각 변수x들과 pvalue값을 데이터프레임화 answer = pd.DataFrame( answer, columns = ['features','pvalue'] # 부여할 컬럼 작명 ) # 유의수준(0.05)보다 작은 pvalue값 중,가장 큰 pvalue answer[answer['pvalue']<0.05]['pvalue'].max() # Drug에 가장 영향력 있는 변수와 p-value값 for_drug = answer[answer['pvalue'] < 0.05] for_drug_best = for_drug.loc[for_drug['pvalue'].idxmax()] print(f'Drug타입에 영향력을 많이 미치는 변수는 {for_drug_best.features}이고, p-value값은 {for_drug_best.pvalue:.5f}입니다.') # 문제3를 위해 원본 복사 q3 = data.copy() # 의사결정나무 수행을 위해 범주형 컬럼--> 수치형 컬럼으로 생성 (mapping) mapping_sex = {'M': 0, 'F': 1} q3['Sex_cd'] = q3['Sex'].replace(mapping_sex) mapping_bp = {'LOW': 0, 'NORMAL': 1, 'HIGH': 2} q3['BP_cd'] = q3['BP'].replace(mapping_bp) mapping_ch = {'HIGH': 1, 'NORMAL':0} q3['Ch_cd'] = q3['Cholesterol'].replace(mapping_ch) # 의사결정나무모델 함수 셋팅 # !pip install scikit-learn # 의사결정나무모델 함수 from sklearn.tree import DecisionTreeClassifier, plot_tree # 의사결정나무에 쓸 변수 features = ['Age','Na_to_K','Sex_cd','BP_cd','Ch_cd'] # 의사결정나무모델 적용 model = DecisionTreeClassifier().fit(q3[features],q3.Drug) # 의사결정나무모델 시각화 plot_tree( model, max_depth = 2, # 조건을 2로 가정 feature_names = features, class_names = q3.Drug.unique(), # 중복되지않게 y의 레이블이름만 precision = 3 # 소수점이하 표기단위 (소숫점 셋째자리까지만표기) ); print(f'의사결정나무 Root Noded의 split value는 14.829입니다.')
풀이과정




답안정리
역시 공부는 손으로 적어가야 머릿속에서 정리가 되는 것 같습니다.

| 문제 | 순서 | 코드 |
|---|---|---|
| 한꺼번에설치 | !pip install -r requirements.txt | |
| 필요함수 | import pandas as pd | |
| 불러오기 | data = pd.read_csv('Dataset_02.csv') | |
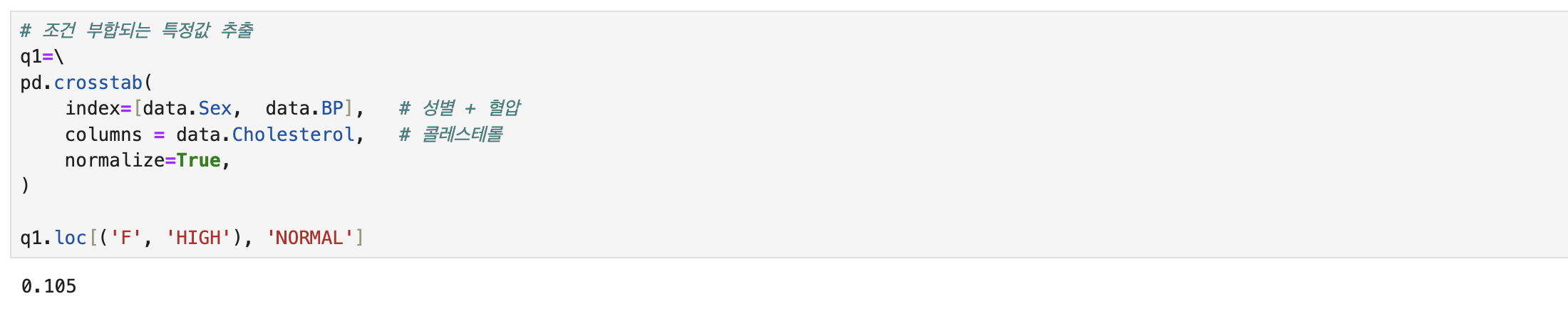
| 1 | 여성으로 혈압High,콜레스테롤Normal인 환자의 전체 대비 비율을 소숫점 세번째자리까지 구하라(반올림) | |
| 전체대비비율 | q1=pd.crosstab( index=[data.Sex, data.BP], columns = data.Cholesterol normalize=True,) | |
| 해당 조건값만 | q1.loc[('F', 'HIGH'), 'NORMAL'] | |
| 2 | Drug타입에 영향을 미치는지 확인하기위하여, 독립성 검정 수행 후 가장 연관성이 큰 p-value값의 소숫점 다섯번째자리까지 구하라 | |
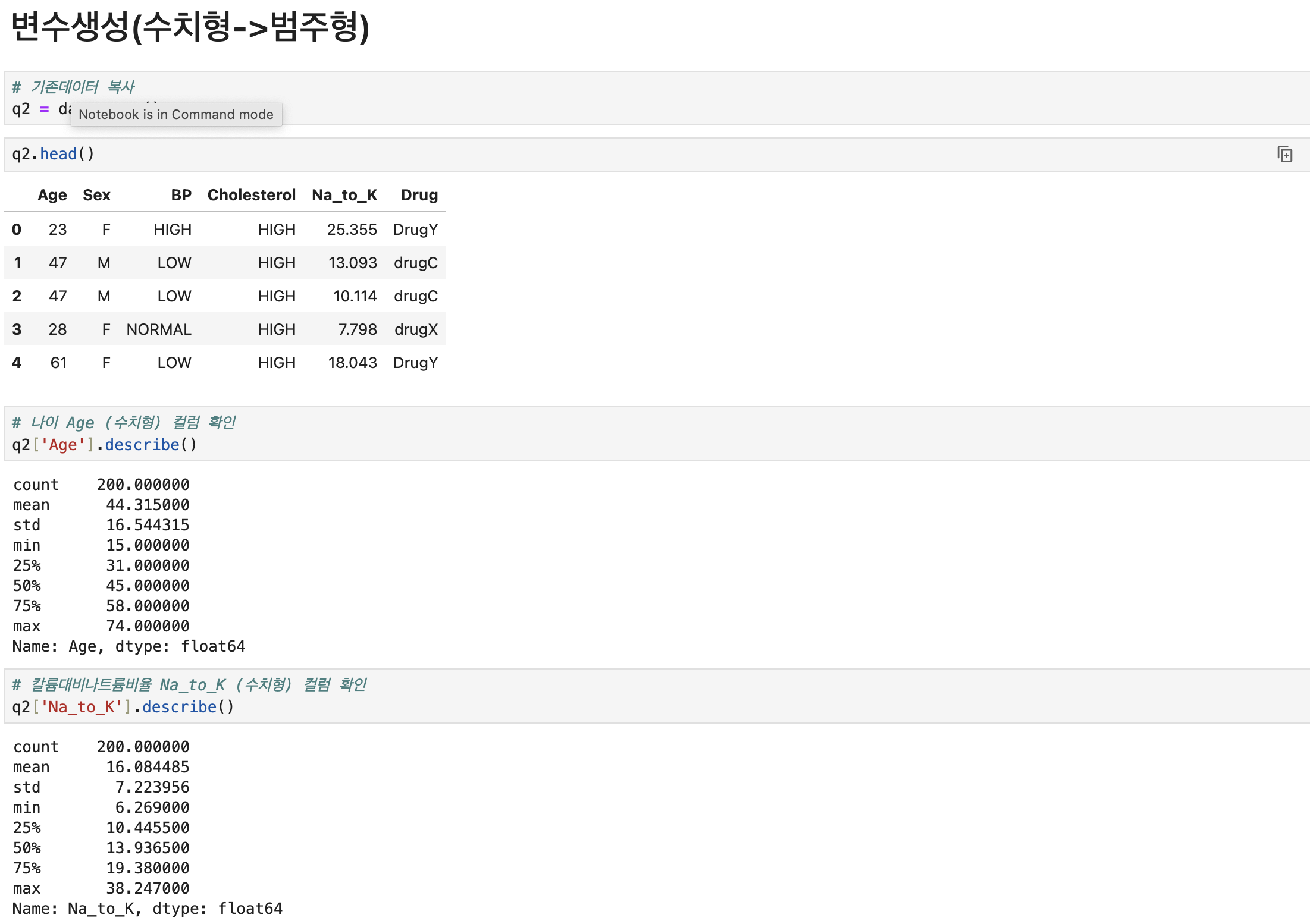
| 원본복사 | q2 = data.copy() | |
| 컬럼생성함수 np.where절 | import numpy as np | |
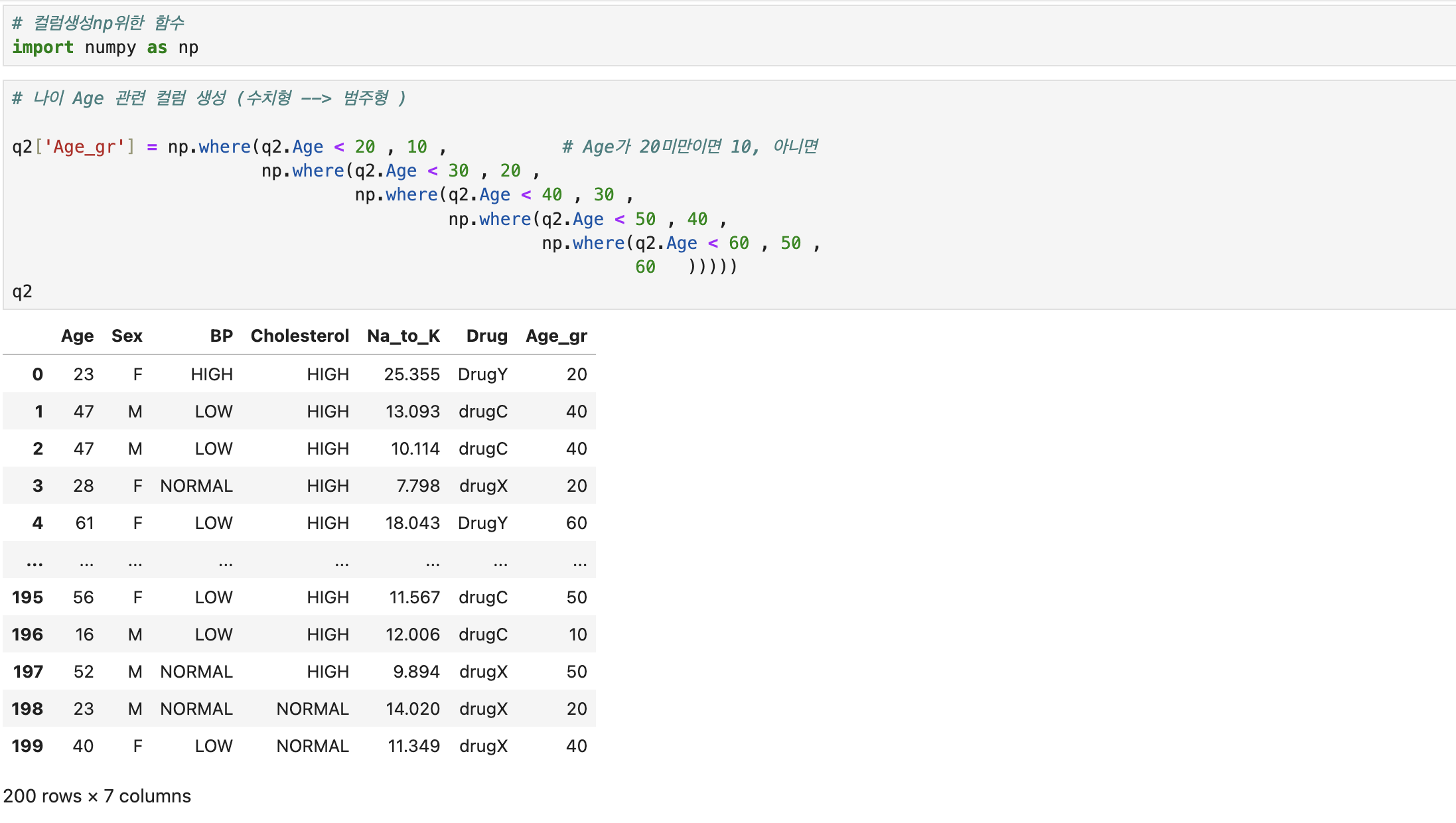
| 컬럼생성 수치형->범주형 | q2['Age_gr'] = np.where(q2.Age < 20, 10, np.where(q2.Age < 30, 20, np.where(q2.Age < 40, 30, np.where(q2.Age < 50, 40, np.where(q2.Age < 60, 50 ,60))))) q2['Na_k_gr'] = np.where(q2.Na_to_k <= 10, 'Lv1', np.where(q2.Na_to_k <= 20, 'Lv2', np.where(q2.Na_to_k <= 30, 'Lv3', 'Lv4'))) | |
| 독립성검정 설치 | !pip install scipy | |
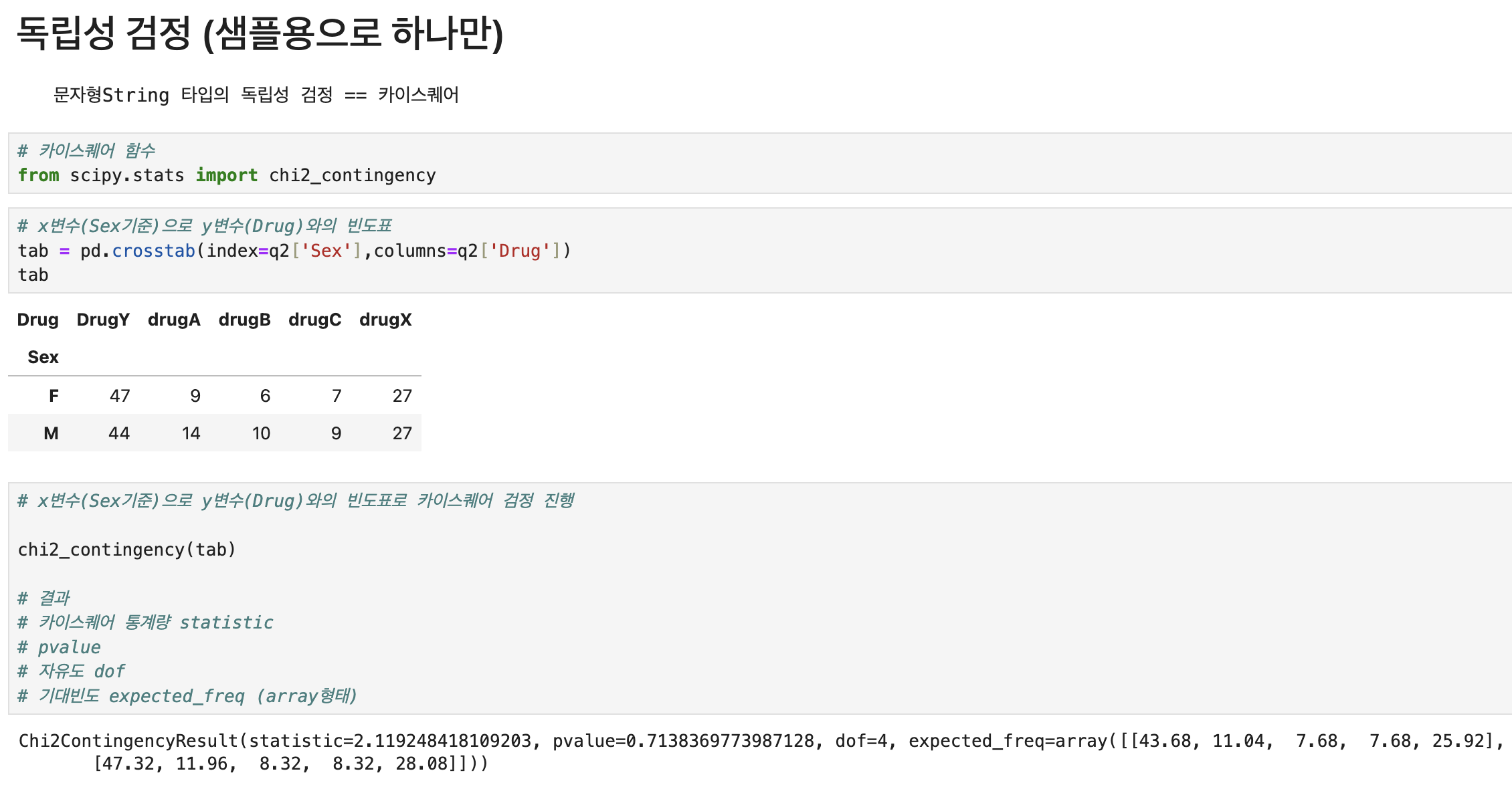
| 독립성검정 함수 | from scipy.stats import chi2_contingency | |
| 독립성검정 변수 | features = ['Sex','BP','Cholesterol','Age_gr','Na_K_gr'] | |
| 변수x들 반복문 빈도표와 pvalue인덱스 | answer=[] for i in features : tab = pd.crosstab(index=q2[i],columns=q2['Drug']) pvalue = chi2_contingency(tab)[1] answer.append([i,pvalue]) | |
| 데이터프레임화 | answer = pd.DataFrame( answer, columns = ['features','pvalue']) | |
| 유의수준이내, 가장 큰 pvalue | answer[answer['pvalue']<0.05]['pvalue'].max() | |
| 가장 영향력 있는 변수와 p-value값 | for_drug = answer[answer['pvalue'] < 0.05] for_drug_best = for_drug.loc[for_drug['pvalue'].idxmax()] | |
| 3 | 의사결정나무를 수행하여,Root Node의 split value의 소숫점 셋째 자리까지 구하라(반올림) | |
| 원본복사 | q3 = data.copy() | |
| 수치형mapping | mapping_sex = {'M': 0, 'F': 1} q3['Sex_cd'] = q3['Sex'].replace(mapping_sex) mapping_bp = {'LOW': 0, 'NORMAL': 1, 'HIGH': 2} q3['BP_cd'] = q3['BP'].replace(mapping_bp) mapping_ch = {'HIGH': 1, 'NORMAL':0} q3['Ch_cd'] = q3['Cholesterol'].replace(mapping_ch) | |
| 함수 셋팅 | !pip install scikit-learn | |
| 함수 | from sklearn.tree import DecisionTreeClassifier, plot_tree | |
| 필요 변수 | features = ['Age','Na_to_K','Sex_cd','BP_cd','Ch_cd'] | |
| 모델적용 | model = DecisionTreeClassifier().fit(q3[features],q3.Drug) | |
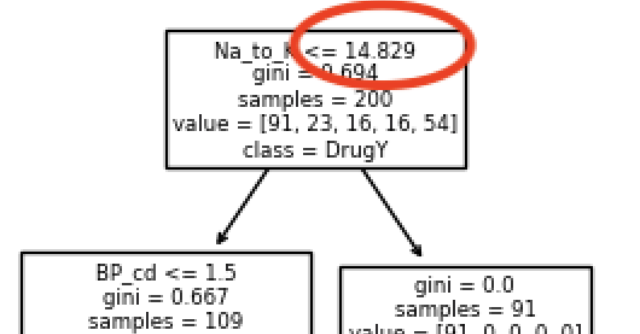
| 시각화 | plot_tree( model, max_depth = 2, feature_names = features, class_names = q3.Drug.unique(), precision = 3) # 소수점이하 표기단위 (소숫점 셋째자리까지만표기)); | |
| Root Node의 split value는 14.829입니다. | ||
 |
이대로 외워라
# 처음 설치 # !pip install -r requirements.txt # 불러오기 import pandas as pd data = pd.read_csv('./Dataset/Dataset_02.csv') # 데이터확인 data.info() print(data.head(2)) ########################## 여성으로서 혈압HIGHT, 콜레스테롤NORMAL 환자의 전체대비비율을 구하라 # 전체대비비율표 q1 = pd.crosstab( index = [data.Sex,data.BP],columns=data.Cholesterol, normalize=True) print(q1) # 해당 조건값만 print(f'\n해당 환자의 전체대비비율은 {q1.loc[("F", "HIGH"), "NORMAL"]}입니다.\n') ########################## Drug타입관련 영향력확인위해, 독립성검정 후 가장 연관성이 큰 p-value를 구하라 # 원본복사 q2 = data.copy() # 수치형 컬럼 --> 범주형(그룹형)컬럼으로 추가 import numpy as np q2['Age_gr'] = np.where(q2.Age < 20, 10 , np.where(q2.Age < 30, 20, np.where(q2.Age < 40 , 30 , np.where(q2.Age < 50 , 40 , np.where(q2.Age < 60, 50 , 60))))) q2['Na_k_gr'] = np.where(q2.Na_to_K <= 10, 'Lv1', np.where(q2.Na_to_K <= 20, 'Lv2', np.where(q2.Na_to_K <=30, 'Lv3','Lv4'))) print(q2.columns) # 독립성검증(카이스퀘어: 범주형그룹형 타입을 검증)에 필요한 변수 features = ['Sex', 'BP', 'Cholesterol', 'Age_gr','Na_k_gr'] # 독립성검증(카이스퀘어)함수 from scipy.stats import chi2_contingency # 독립성검증(카이스퀘어)진행 : 각 변수들의 빈도표crosstab과 pvalue추출 answer=[] for i in features : tab = pd.crosstab(index=q2[i],columns=q2['Drug']) pvalue= chi2_contingency(tab)[1] answer.append([i,pvalue]) answer = pd.DataFrame(answer, columns = ['features','pvalue']) # 유의수준(0.05기준)보다 작은 변수(연관성이있음)중에 pvalue값이 가장 큰 값 # answer['pvalue']<0.05 하면 True로 나옴 = 연관성이 있음 for_drug = answer[answer['pvalue']<0.05] for_drug_max=for_drug.loc[for_drug['pvalue'].idxmax()] print(f'\n유의수준0.05보다 작은 변수들과 pvalue값\n{for_drug}\n') print(f'그 중에서 영향력(pvalue값)이 가장 큰 변수와 pvalue값\n{for_drug_max}\n') answer[answer['pvalue']<0.05]['pvalue'].max() ########################## 의사결정나무를 수행하여, Root Node의 split value를 구하라 # 원본복사 q3 = data.copy() # 범주형(그룹형) 컬럼 --> 수치형 컬럼으로 추가 mapping_sex = {'M': 0, 'F': 1} q3['Sex_cd'] = q3['Sex'].replace(mapping_sex) mapping_bp = {'LOW': 0, 'NORMAL': 1, 'HIGH': 2} q3['BP_cd'] = q3['BP'].replace(mapping_bp) mapping_ch = {'HIGH': 1, 'NORMAL':0} q3['Ch_cd'] = q3['Cholesterol'].replace(mapping_ch) print(q3.columns) # 의사결정나무 수행 및 시각화 from sklearn.tree import DecisionTreeClassifier, plot_tree features = ['Age','Na_to_K','Sex_cd','BP_cd','Ch_cd'] model = DecisionTreeClassifier().fit(q3[features],q3.Drug) plot_tree( model, max_depth=2, # 2로 가정 feature_names=features, #class_names=q3.Drug.unique(), # error나는경우 class_names=q3.Drug.unique().tolist(), precision=3 # 소숫점 3자리까지 ); print(f'\nRoot Node의 split value는 14.829입니다.')


이대로 풀어봐라
# 처음 설치 (처음 할때는 주석 풀고 진행)
# 불러오기
# 데이터확인
########################## 여성으로서 혈압HIGHT, 콜레스테롤NORMAL 환자의 전체대비비율을 구하라
########################## Drug타입(y)와의 영향력 확인위해, 각 x변수들과의 독립성검정 후 가장 연관성이 큰 p-value를 구하라
########################## 의사결정나무를 수행하여, Root Node의 split value를 구하라
왜 독립성 검정을 해야해?
독립성 검정은 두 가지 범주형 변수 간의 관계를 평가하는 통계적인 방법 중 하나입니다. 여기서는 'Drug' 타입과 다른 범주형 변수들 간의 관계를 검정합니다.
Drug(y)와 x들 하나하나씩 반복문해서 검정합니다.
첫번째, Drug와 Age
두번째, Drug와 Na_to_K
세번째, Drug와 Sex_cd
...이래서 반복문을 진행하는겁니다.독립성 검정을 통해 두 범주형 변수 간에 통계적으로 유의한 관계가 있는지 확인할 수 있습니다.
즉, 한 변수가 다른 변수에 영향을 미치는지를 확인할 수 있습니다.변수 간의 상호작용을 이해하고 향후 분석 방향을 결정하는 데에 유용한 통계적 도구 중 하나입니다.
