사용 라이브러리
모델 : scipy.cluster.AgglomerativeClustering
거리계산 : scipy.cluster.hierarchy.linkage
시각화 : scipy.cluster.hierarchy.dendrogram
평가 : sklearn.metrics.silhouette_score , silhouette_samples, yellowbrick.cluster.KElbowVisualizer
계층적 군집은 sklearn이 아님
계층적 군집 (1)
계층적 군집 분석은 데이터와 데이터, 군집과 데이터, 군집과 군집 간의 거리에 대한 유사도로 군집을 형성
Hierarchical Clustering
계층적 군집 분석은 데이터와 데이터, 군집과 데이터, 군집과 군집 간의 거리에 대한 유사도로 군집을 형성
동작하는 방식
두 점 사이를 가까운 것끼리 합침. 이 과정을 반복하여 전체가 하나의 클러스터에 속할 때까지 반복
이 과정에 대한 시각화 결과물이 덴드로그램 형태로 나타남
어떻게 두 점 사이의 거리를 결정할지에 대한 측정 방식이 필요함
유클리디안 거리 측정(직선거리), 맨하탄 거리 측정 방식(블록거리)
군집간의 거리를 구하는 방법
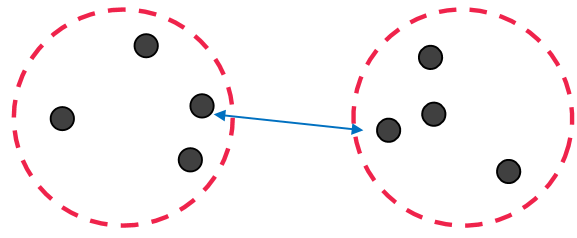
1. Min(Single Link)
군집과 군집의 거리를 구할 때 가장 최소 거리인 것을 유사도로 측정하는 방식
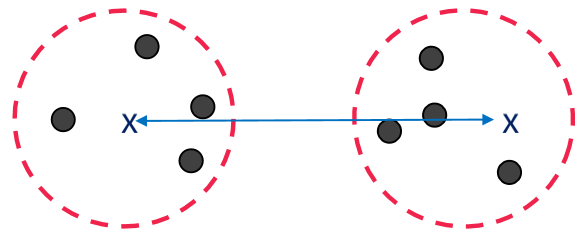
2. Max(Complete Link)
군집과 군집의 거리를 구할 때 가장 최대 거리인 것을 유사도로 측정하는 방식
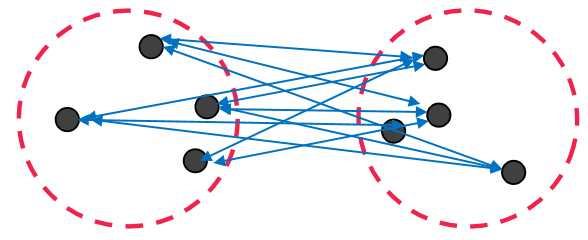
3. Average Link
군집과 군집의 거리를 구할 때 거리의 평균을 구해 유사도로 측정하는 방식
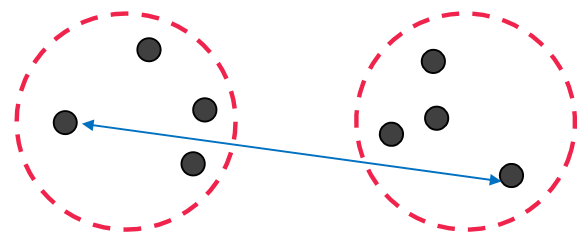
4. Centroids
군집과 군집의 거리를 구할 때 데이터의 중심점 거리를 유사도로 측정하는 방식
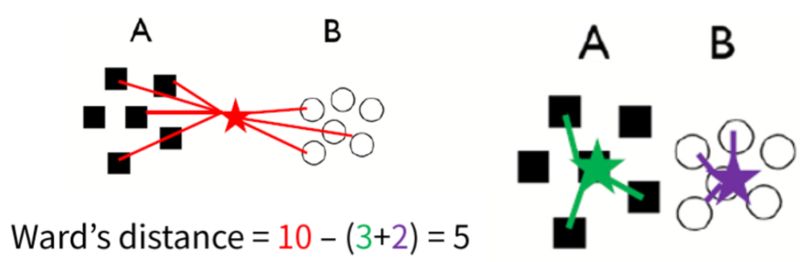
5. Ward's method
두 개의 군집이 병합되었을 때 증가하는 변동성의 양으로 유사도를 측정하는 방식
변동성
두 군집의 중앙값과 두 군집에 있는 모든 데이터와의 거리의 합에서 군집간에 형성되는 거리를 뺀 값
세부 군집 알고리즘으로 Agglomerative Hierarchical Clustering(sklearn을 제공), Divisive Hierarchical Clustering 이 존재
계층 군집 수행
method 파라미터
군집간의 거리를 구하는 방법
single(기본 값), complete, average, weighted, centroid 중 선택
참고 : https://docs.scipy.org/doc/scipy/reference/generated/scipy.cluster.hierarchy.linkage.html
metric 파라미터
scipy.spatial.distance.pdist 클래스의 인스턴스
euclidean(직선): metric의 기본이며 점 사이의직선 거리 측정cityblock: 점 사이의맨하탄 거리 측정(블록 형태)seuclidean: 표준화된 유클리디안sqeuclidean: 제곱된 유클리디안cosine: 코사인 거리 계산- 콜백함수 지정 가능
- linkage : 거리 측정 방법 지정
- 기본 값 : ward
- 그 외 값 : ‘ward’, ‘complete’, ‘average’, ‘single’
군집 시각화
scipy 패키지에서 제공하는 dendrogram을 사용
dendrogram(lnk,
orientation='top', #방향 : top(기본값), bottom, left, right
labels=range(1,11), #라벨 인덱스
# count_sort : 각 노드 n에 대해 두 하위 링크가 표시되는 순서
# False : 아무것도 안함(기본값)
# ascending : 클러스터 원본 개체수가 가장 적은 하위 객체가 먼저 출력
# descending : 클러스터 원본 개체수가 가장 많은 하위 객체가 먼저 출력
count_sort="ascending",
# distance_sort : 각 노드 n에 대해 두 하위 링크가 표시되는 순서
# False : 아무것도 안함(기본값)
# ascending : 직계 자손의 사이의 거리가 최소인 하위 객체가 먼저 출력
# descending : 직계 자손의 사이의 거리가 최대인 하위 객체가 먼저 출력
distance_sort="ascending",
show_leaf_counts=True, #True : 맨 아래 노드에 속한 개체 수 표시(k>1 경우만)
)병합군집(sklearn)
학습모델 구현
AgglomerativeClustering(n_clusters=2, #찾아야 할 클러스터 수
# 클러스터 간 거리 측정 방법(euclidean, cityblock, manhattan, cosine)
# affinity 파라미터는 sklearn 1.4.0 버전부터 사용 불가
metric="euclidean",
# 거리측정 방법
# 'ward'(기본값), 'complete', 'average', 'single'
linkage="single",
# 노드간의 거리 계산값을 저장할지 여부(시각화를 위해서는 True를 필수 적용)
compute_distances=True
)노드간의 거리 확인
학습 모델 객체 생성 과정에서 반드시 compute_distances=True로 설정돼야만 한다.
군집결과 시각화
# scipy는 아래의 과정을 자동으로 수행, sklearn은 수동으로 설정(공식으로 판단할 것)
counts = np.zeros(clustering.children_.shape[0]) #zeros 모든 원소가 0인 요소
n_samples = len(clustering.labels_)
# labels_ : 각 입력 데이터의 그룹 번호
# 자식 노드를 반복하면서 점을 찍어준다(자식이 몇개 있는지 확인하는 과정)
for i, merge in enumerate(clustering.children_):
current_count = 0
for child_idx in merge:
if child_idx < n_samples:
current_count += 1 # leaf node
else:
current_count += counts[child_idx - n_samples]
counts[i] = current_count
# 자식 노드의 수를 저장하는 배열
linkage_matrix = np.column_stack(
[clustering.children_, clustering.distances_, counts]
).astype(float)
# plt를 설정하지 않으면 쓸데 없는 값도 함께 추출됨
# dendrogram(linkage_matrix)
plt.figure(figsize=(10,6))
dendrogram(linkage_matrix)
plt.show()
plt.close()