
안녕하세요! 이번 velog는 제가 이번학기 머신러닝을 이용해 뇌파 측정하는 프로젝트에 참여하고 있어서 배운것들 까먹지 않게 기록하는 velog입니다.
이번 시간 목표 : mne python과 .mat파일을 이용해 ERD측정하기
ERD?
백엔드에서 사용하는 Entity Relationship Diagram? 😵 아닙니다 ㅎ
Event Related Desynchronization으로 특정 작업을 수행할 때 움직임과 관련된 뇌파의 활동이 억제되는 현상을 ERD라고 합니다.
뇌파란?
Encephal(뇌) 안의 신경활동들에 의한 Electrical(전기)적 신호를 Gram(기록)한 것 -> Electro Encephal Gram으로 줄여서 EEG라고 합니다.
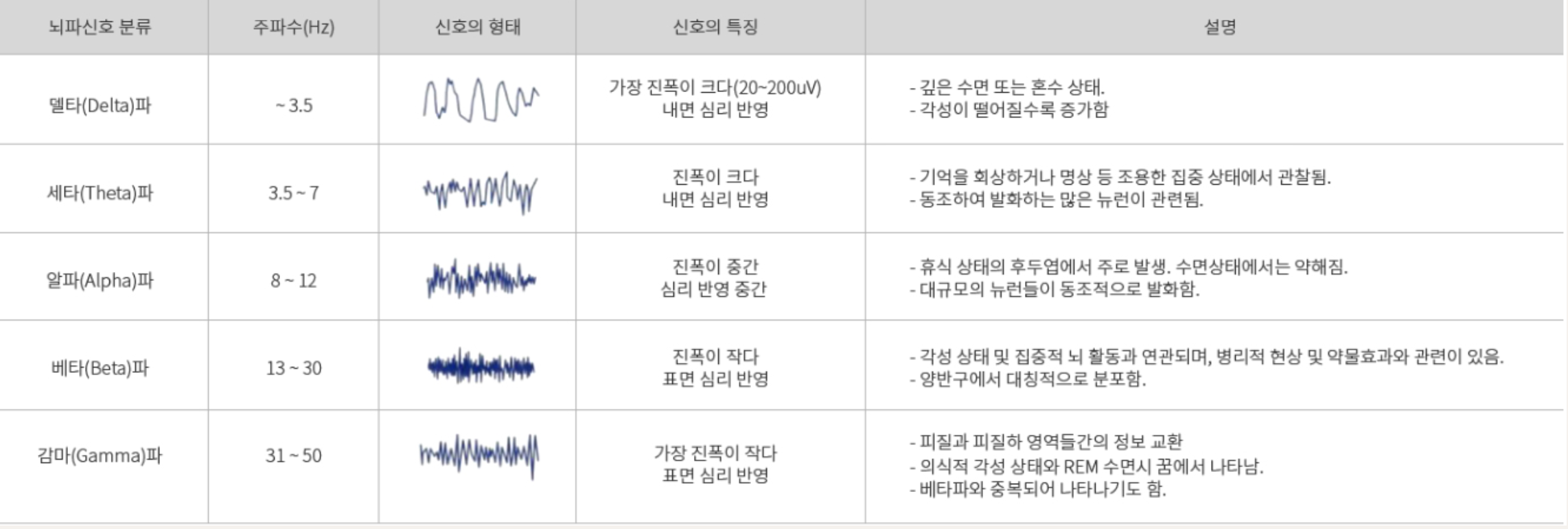
뇌파의 종류로는 아래 5가지가 있습니다.
mne-python?
뇌파와 같은 생체신호를 다루기 용이하게 지원하는 외부 Library
기초 개념들
- Raw Data : 습득한 뇌 신호에 아무런 작업하지 않은 데이타
- Event : 사건 / 실험이 일어났을 때 특정 시간 지점
- Ephoch : 실험에서 특정 시간이나 자극에 대해 뇌는 아주 짧게 반응하는데, 이 짧은 반응을 기록한것
code
import mne #mne python 사용하기 위해
import scipy.io #입출력 특히 matlab파일 사용하기 위해
import numpy as np #python의 대규모 수학 계산 도와줌
import matplotlib.pyplot as plt #데이타를 그래프로 시각화.mat 파일 raw 객체로 변환시키기
mne에서 데이타 작업하기 위해서는 raw 객체로 만들어줘야 합니다.
# mat file 불러오기
mat = scipy.io.loadmat("GIST_MI/s01.mat")# mat 구조 살펴보기
for i in mat:
print(i)
print()
# eeg데이타 추출하기
fields = list(mat['eeg'][0].dtype.fields.keys())
print("fields:", fields)raw 객체를 생성하기 위해 raw 안에 뭐가 들어갈지 info 객체가 필요해서 info 객체 만들기 -> 채널 이름과 채널 유형이 필요함
#eeg 채널이름 정의
eeg_ch_names = [
"Fp1", "AF7", "AF3", "F1", "F3", "F5", "F7", "FT7", "FC5", "FC3", "FC1",
"C1", "C3", "C5", "T7", "TP7", "CP5", "CP3", "CP1", "P1", "P3", "P5", "P7",
"P9", "PO7", "PO3", "O1", "Iz", "Oz", "POz", "Pz", "CPz", "Fpz", "Fp2",
"AF8", "AF4", "AFz", "Fz", "F2", "F4", "F6", "F8", "FT8", "FC6", "FC4",
"FC2", "FCz", "Cz", "C2", "C4", "C6", "T8", "TP8", "CP6", "CP4", "CP2",
"P2", "P4", "P6", "P8", "P10", "PO8", "PO4", "O2",
]
#emg 채널이름 정의
emg_ch_names = ["EMG1", "EMG2", "EMG3", "EMG4"]
#stim이라는 채널과 eeg,emg 채널들 결합
ch_names = eeg_ch_names + emg_ch_names + ["Stim"]
#채널 유형 정의 -> eeg채널 64개, emg채널 4개, stim채널 1개
ch_types = ["eeg"] * 64 + ["emg"] * 4 + ["stim"]stim channel?
EEG,EMG에서 신호를 받아 기록하는게 아니라 Event를 기록하는 채널 -> Stim Channel을 통해 언제 event가 발생했는지 알 수 있다.
제가 이번 캠프때 배운 논문에서는 imagery_event와 movement_event가 Stim channel역할을 합니다.
srate = mat['eeg'][0]['srate'][0][0] #eeg구조체에서 sample rate추출
# info 생성
info = mne.create_info(ch_names=ch_names, sfreq=srate, ch_types=ch_types)
# Stim channel 붙이기
left = np.vstack((mat['eeg'][0]['imagery_left'][0], mat['eeg'][0]['imagery_event'][0]))
right = np.vstack((mat['eeg'][0]['imagery_right'][0], mat['eeg'][0]['imagery_event'][0]))위에서 생성한 left와 right으로 raw 객체 생성!!
# Raw 생성
lr = mne.io.RawArray(left, info=info)
rr = mne.io.RawArray(right, info=info)
raw = [lr, rr]LEFT = 0
RIGHT = 1Reference
이 raw data 바로 사용하냐? -> NO!!
뇌파 측정을 할 때 두피, 머리카락 등 외부 요소들에 의해 영향을 받을 수도 있어서 이 영향들을 최소화하는 과정이 필요한데, 이 작업을 Reference라고 합니다.
mne.set_eeg_reference(raw[LEFT], ch_type='eeg')
mne.set_eeg_reference(raw[RIGHT], ch_type='eeg')Raw 객체 살펴보기
print("Raw의 길이:", len(raw[LEFT]))
print("초로 환산하면:", len(raw[LEFT]) / srate)
print("n_imagery_trials로 나누면:", len(raw[LEFT]) / srate / mat['eeg'][0]['n_imagery_trials'][0][0])위 코드 실행시키면
Raw의 길이: 358400
초로 환산하면: [700.]
n_imagery_trials로 나누면: [7.]이런 결과가 나오는데, 이것은 raw 배열에 7초짜리 실험이 100개가 담겼다는 뜻입니다.
Epoching
연속적인 100개의 실험 2차원으로 재구성하는 작업 -> 실험들 event 로 나누기 때문에 Epoch 을 생성하기 위해서는 Event 가 필요하다.
# events 생성
le = mne.find_events(raw[LEFT], stim_channel='Stim')
re = mne.find_events(raw[RIGHT], stim_channel='Stim')
events = [le, re]Raw데이타 plot하기
plot? -> EEG 분석에서 raw를 plot한다는 것은 EEG 데이타에서 기록된 time-series 데이타를 시각화 한다는 뜻입니다.
raw_data = raw[LEFT].get_data()
plt.plot(raw_data[0])
plt.show()이 코드에서는 첫번째 채널에서 eeg 데이타를 시각화 한다고 생각하시면 됩니다.

실행시키면 이런 그래프로 나오고, x축 : 시간 , y축 : eeg 신호의 진폭입니다.
Baseline correction
앞에서 말했듯이 위 그래프는 100개의 실험을 연속적으로 나타낸 것이고, 이를 100개로 나눌 때 기준점이 필요합니다. 그 기준점을 baseline 이라고 하고 이 기준점으로 다시 나누는 작업을 baseline correction 이라고 합니다.
# Epochs 생성
le = mne.Epochs(raw[LEFT], events[LEFT], tmin=-1.999, tmax=5, baseline=(-0.5, 0))
re = mne.Epochs(raw[RIGHT], events[RIGHT], tmin=-1.999, tmax=5, baseline=(-0.5, 0))
epochs = [le, re]생성한 Epochs 중에서 c3과, c4지점의 Epoch만 관찰하기 때문에 다시 한번 filter
epochs[LEFT].load_data()
epochs[RIGHT].load_data()
epochs[LEFT].pick(picks=['C3', 'C4'])
epochs[RIGHT].pick(picks=['C3', 'C4'])C3 = 0
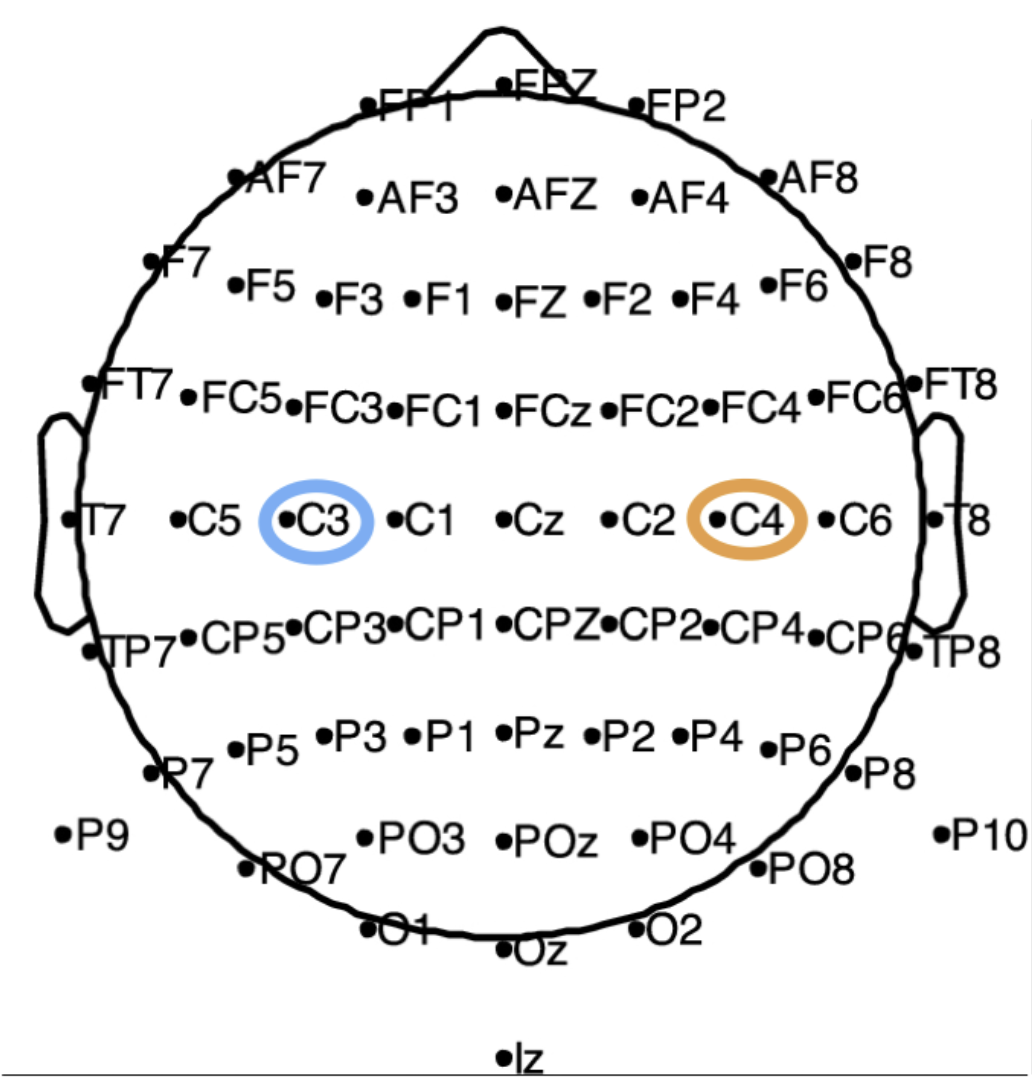
C4 = 1C3? C4?
뇌의 특정 지점입니다.
Epoching한 데이타 다시 plot
print(epochs[LEFT].get_data().shape)실행시키면 (100, 2, 3584)이 나오는데 100 - Epoch의 갯수, 2 - EEG 채널의 갯수, 3584 - Epoch 당 들어있는 데이타의 갯수를 뜻합니다.
Left와 Right 나눠서 c3지점과 c4지점의 EEG 관찰하기
data = [epochs[LEFT].get_data(), epochs[RIGHT].get_data()]
fig, axes = plt.subplots(nrows=2, ncols=1, figsize=(8, 8))
axes[0].plot(data[LEFT][0][C3], label='C3')
axes[0].plot(data[LEFT][0][C4], label='C4')
axes[0].set_title("Left")
axes[0].legend()
axes[1].plot(data[RIGHT][0][C3], label='C3')
axes[1].plot(data[RIGHT][0][C4], label='C4')
axes[1].set_title("Right")
axes[1].legend()
plt.show()Run->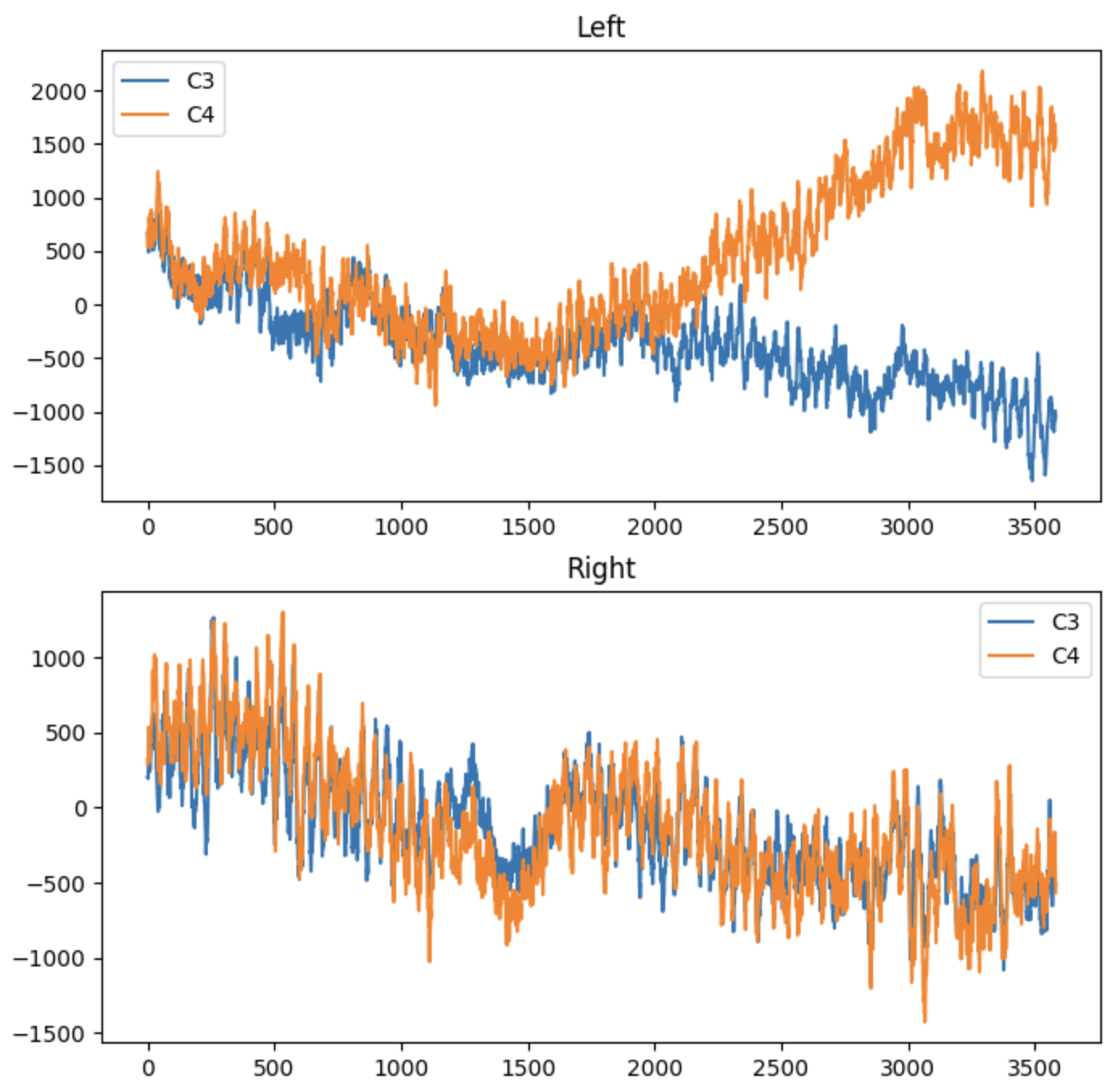
BandPass Filtering
ERD는 주로 알파파 영역에서 발생하기 때문에 관찰한 raw data를 알파파(Alpha wave) 대역으로 필터링 시켜야 합니다.
le = [] #Left에 해당하는 epochs 저장하기 위해
re = []
for i in range(0, len(epochs[LEFT])): #Left에 있는 epoch들 순회
#Left에 있는 epoch들 알파파로 필터링 후 le에 추가
le.append(epochs[LEFT][i].filter(l_freq=8, h_freq=13))
re.append(epochs[RIGHT][i].filter(l_freq=8, h_freq=13))
epochs = [le, re]알파파로 필터링 한 Epoch들 plot하기
#Left와 Right에 대한 데이타 시각화
data = [epochs[LEFT][0].get_data(), epochs[RIGHT][0].get_data()]
fig, axes = plt.subplots(nrows=2, ncols=1, figsize=(8, 8))
axes[0].plot(data[LEFT][0][C3], label='C3')
axes[0].plot(data[LEFT][0][C4], label='C4')
axes[0].set_title("Left")
axes[0].legend()
axes[1].plot(data[RIGHT][0][C3], label='C3')
axes[1].plot(data[RIGHT][0][C4], label='C4')
axes[1].set_title("Right")
axes[1].legend()
plt.show()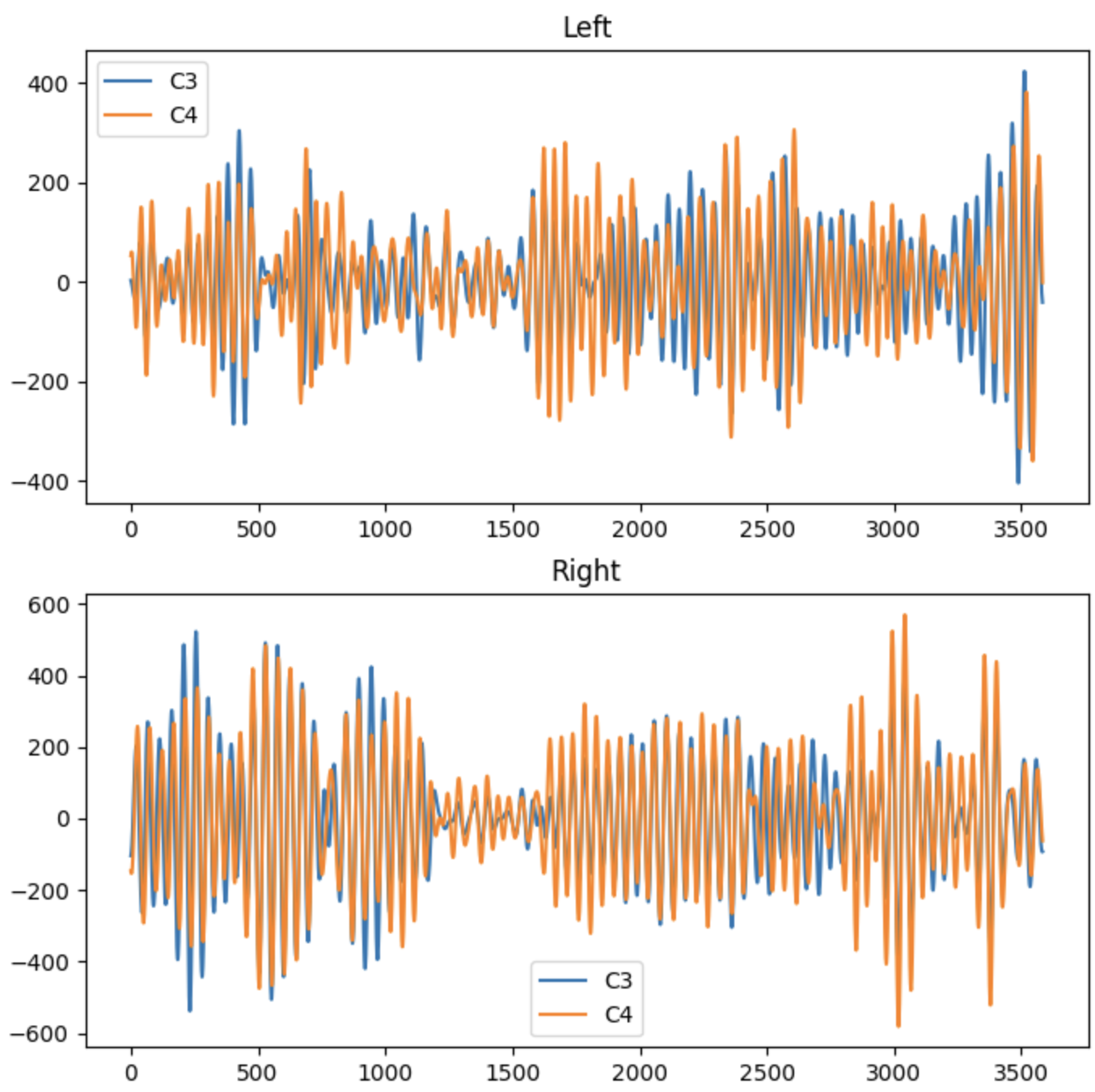
위 사진은 EEG의 두 채널 c3과 c4를 라벨링한 조건으로 시각화해서 변화를 볼 수 있게 한것입니다. -> y축이 뇌의 진폭으로 뇌 활동의 강도를 측정합니다.
실험 데이타 평균 구하기
평균을 구하려면 우선 데이타를 절댓값으로 바꿔야 합니다.
# 모든 데이타들 data배열에 넣기
ld = []
rd = []
for i in range(0, len(epochs[LEFT])):
ld.append(epochs[LEFT][i].get_data()[0])
rd.append(epochs[RIGHT][i].get_data()[0])
data = [ld, rd]
#데이타에 있는 값들 절댓값 반환
data = [np.abs(data[LEFT]), np.abs(data[RIGHT])]
# 예시 데이터 plot
fig, axes = plt.subplots(nrows=2, ncols=1, figsize=(8, 8))
axes[0].plot(data[LEFT][0][C3], label='C3')
axes[0].plot(data[LEFT][0][C4], label='C4')
axes[0].set_title("Left")
axes[0].legend()
axes[1].plot(data[RIGHT][0][C3], label='C3')
axes[1].plot(data[RIGHT][0][C4], label='C4')
axes[1].set_title("Right")
axes[1].legend()
plt.show()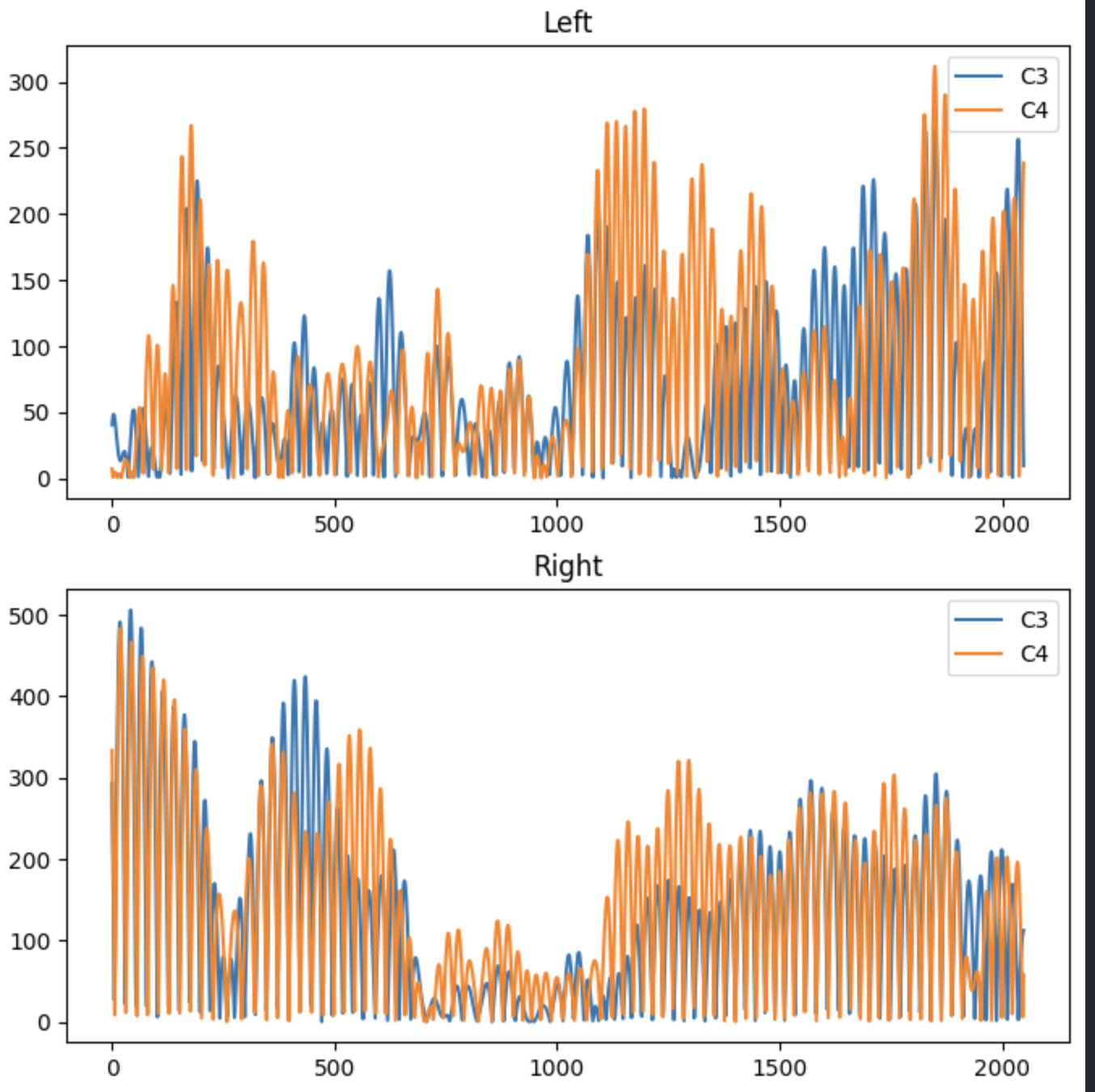
#평균 구하기
data = [np.mean(data[LEFT], axis=0), np.mean(data[RIGHT], axis=0)]
# 예시 데이터 plot
fig, axes = plt.subplots(nrows=3, ncols=1, figsize=(8, 15))
axes[0].plot(data[LEFT][C3], label='C3')
axes[0].plot(data[LEFT][C4], label='C4')
axes[0].set_title("Left")
axes[0].legend()
axes[1].plot(data[RIGHT][C3], label='C3')
axes[1].plot(data[RIGHT][C4], label='C4')
axes[1].set_title("Right")
axes[1].legend()
axes[2].plot(data[LEFT][C3] - data[RIGHT][C3], label='C3')
axes[2].plot(data[LEFT][C4] - data[RIGHT][C4], label='C4')
axes[2].set_title("Left - Right")
axes[2].legend()
plt.show()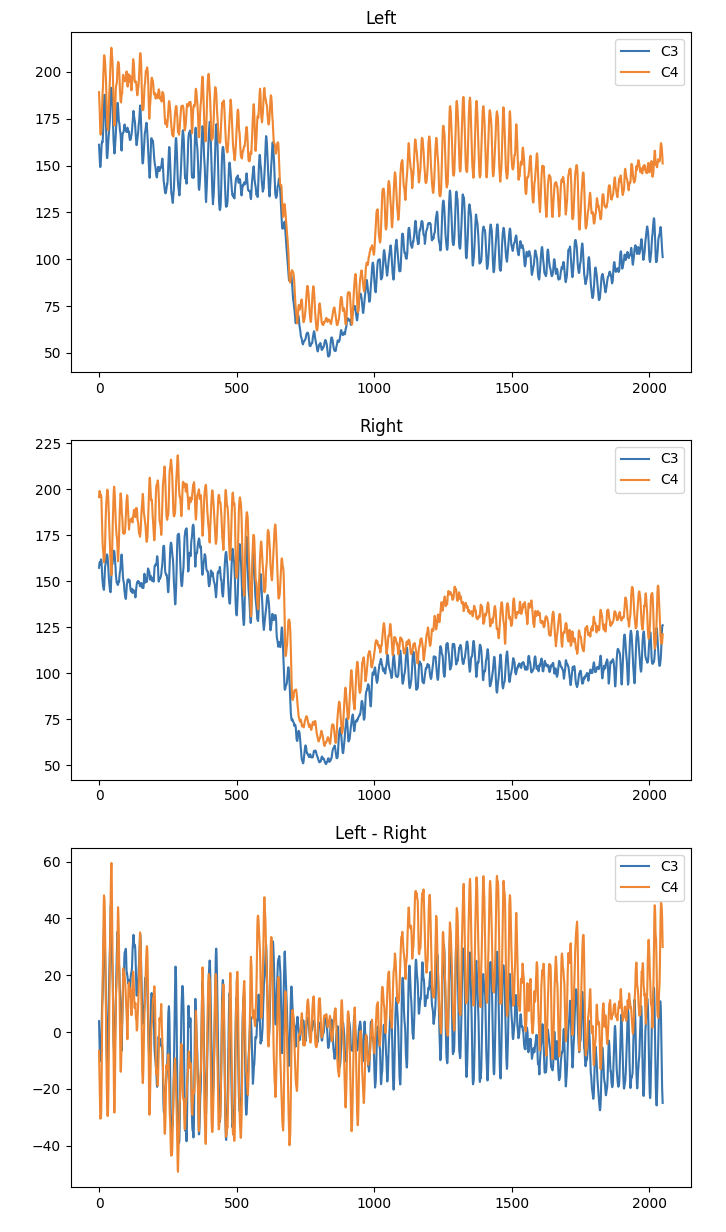
확인할 수 있는 것
- Left와 Right에서 어떤 차이점을 발생시키는지
- C3과 C4의 차이점을 발견해서 뇌의 각 지점마다 어떤 차이점이 있는지 확인할 수 있다.
- 신호 패턴을 분석해 특정 자극이 뇌파 패턴과 관련 있는지 확인할 수 있다.
ERD 구하기
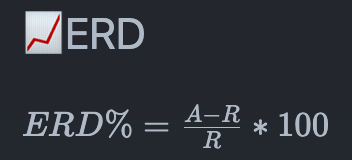
A : 활동중인 상태에서 측정한 뇌파의 전력
R : 작업하고 있지 않을 때 뇌파 상태
A - R < 0 : 뇌가 덜 활동하고 있다
(A - R) / R : 얼마나 뇌 활동 감소했는지 비율로 만듦
*100 : 백분율로 만든다
ERD Code
R = [np.mean(data[LEFT][:, :513], axis=1).reshape(2,1), np.mean(data[RIGHT][:, :513], axis=1).reshape(2,1)]
ERD = [(data[LEFT] - R[LEFT]) / R[LEFT] * 100, (data[RIGHT] - R[RIGHT]) / R[RIGHT] * 100]
# 예시 데이터 plot
fig, axes = plt.subplots(nrows=3, ncols=1, figsize=(8, 15))
axes[0].plot((np.arange(len(ERD[LEFT][C3])) - 512) / 512, ERD[LEFT][C3], label='C3')
axes[0].plot((np.arange(len(ERD[LEFT][C4])) - 512) / 512, ERD[LEFT][C4], label='C4')
axes[0].set_title("Left")
axes[0].set_xlabel("Times")
axes[0].set_ylabel("ERD%")
axes[0].legend()
axes[1].plot((np.arange(len(ERD[RIGHT][C3])) - 512) / 512, ERD[RIGHT][C3], label='C3')
axes[1].plot((np.arange(len(ERD[RIGHT][C4])) - 512) / 512, ERD[RIGHT][C4], label='C4')
axes[1].set_title("Right")
axes[1].set_xlabel("Times")
axes[1].set_ylabel("ERD%")
axes[1].legend()
axes[2].plot((np.arange(len(ERD[RIGHT][C3])) - 512) / 512, ERD[LEFT][C3] - ERD[RIGHT][C3], label='C3')
axes[2].plot((np.arange(len(ERD[RIGHT][C3])) - 512) / 512, ERD[LEFT][C4] - ERD[RIGHT][C4], label='C4')
axes[2].set_title("Left - Right")
axes[2].set_xlabel("Times")
axes[2].set_ylabel("ERD%")
axes[2].legend()
for ax in axes:
ax.axvline(x=0, color='red', linestyle='--')
plt.show()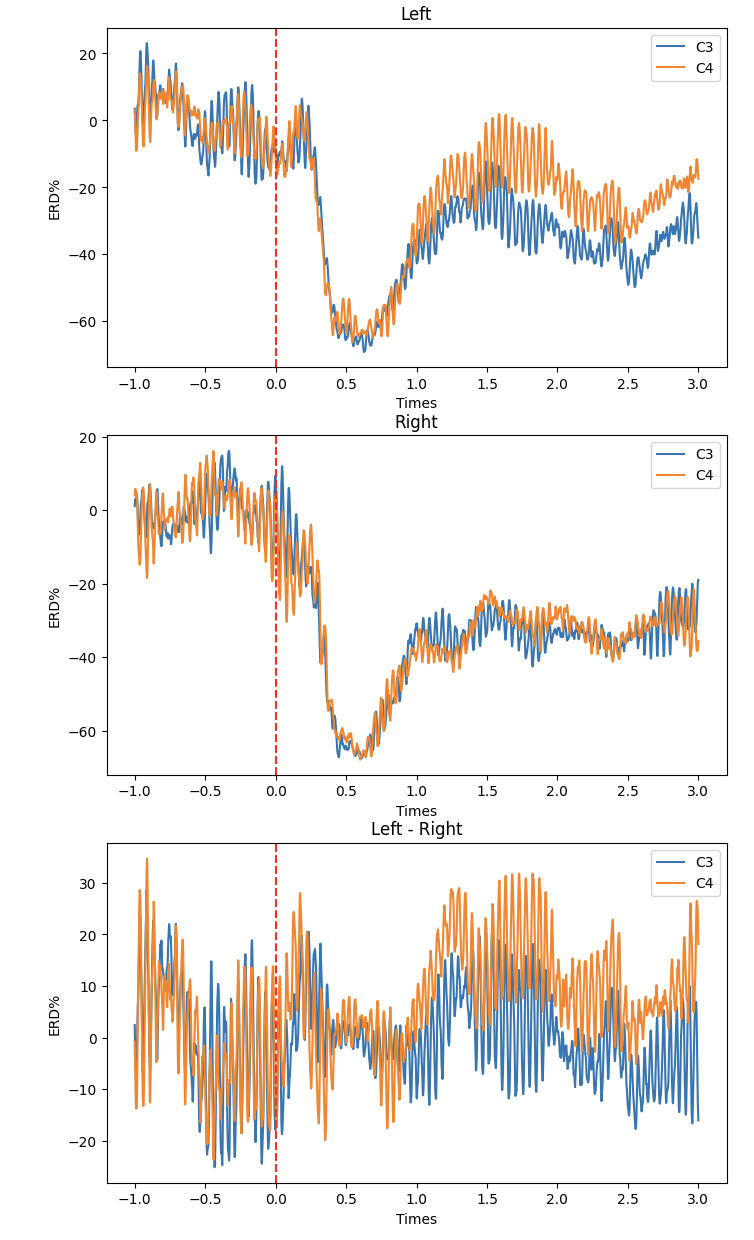
위 그래프로 -1~3s 신호를 평균으로 그래프를 그려서 Left의 경우 C4가 약하고, Right는 C3이 약하다는 것을 알수 있습니다.
위 방법대로 하면 사람 한명 .mat파일로 된 데이타를 raw객체로 만든 후 ERD로 보는 프로세스 끝입니다!

안녕하세요 컴공과 진학 희망하는 고등학교3학년인데요! 이번에 bci관련 발표를 준비하게 되어서 뇌파분석하는 방법을 찾던 중 들리게 되었습니당 혹시 궁금한 거 여쭤봐도 될까요?