DNA
- 티어 : Silver 5
- 시간 제한 : 2 초
- 메모리 제한 : 128 MB
- 알고리즘 분류 : 구현, 문자열, 브루트포스 알고리즘
문제
DNA란 어떤 유전물질을 구성하는 분자이다. 이 DNA는 서로 다른 4가지의 뉴클레오티드로 이루어져 있다(Adenine, Thymine, Guanine, Cytosine). 우리는 어떤 DNA의 물질을 표현할 때, 이 DNA를 이루는 뉴클레오티드의 첫글자를 따서 표현한다. 만약에 Thymine-Adenine-Adenine-Cytosine-Thymine-Guanine-Cytosine-Cytosine-Guanine-Adenine-Thymine로 이루어진 DNA가 있다고 하면, “TAACTGCCGAT”로 표현할 수 있다. 그리고 Hamming Distance란 길이가 같은 두 DNA가 있을 때, 각 위치의 뉴클오티드 문자가 다른 것의 개수이다. 만약에 “AGCAT"와 ”GGAAT"는 첫 번째 글자와 세 번째 글자가 다르므로 Hamming Distance는 2이다.
우리가 할 일은 다음과 같다. N개의 길이 M인 DNA s1, s2, ..., sn가 주어져 있을 때 Hamming Distance의 합이 가장 작은 DNA s를 구하는 것이다. 즉, s와 s1의 Hamming Distance + s와 s2의 Hamming Distance + s와 s3의 Hamming Distance ... 의 합이 최소가 된다는 의미이다.
입력
첫 줄에 DNA의 수 N과 문자열의 길이 M이 주어진다. 그리고 둘째 줄부터 N+1번째 줄까지 N개의 DNA가 주어진다. N은 1,000보다 작거나 같은 자연수이고, M은 50보다 작거나 같은 자연수이다.
출력
첫째 줄에 Hamming Distance의 합이 가장 작은 DNA 를 출력하고, 둘째 줄에는 그 Hamming Distance의 합을 출력하시오. 그러한 DNA가 여러 개 있을 때에는 사전순으로 가장 앞서는 것을 출력한다.
예제 입출력
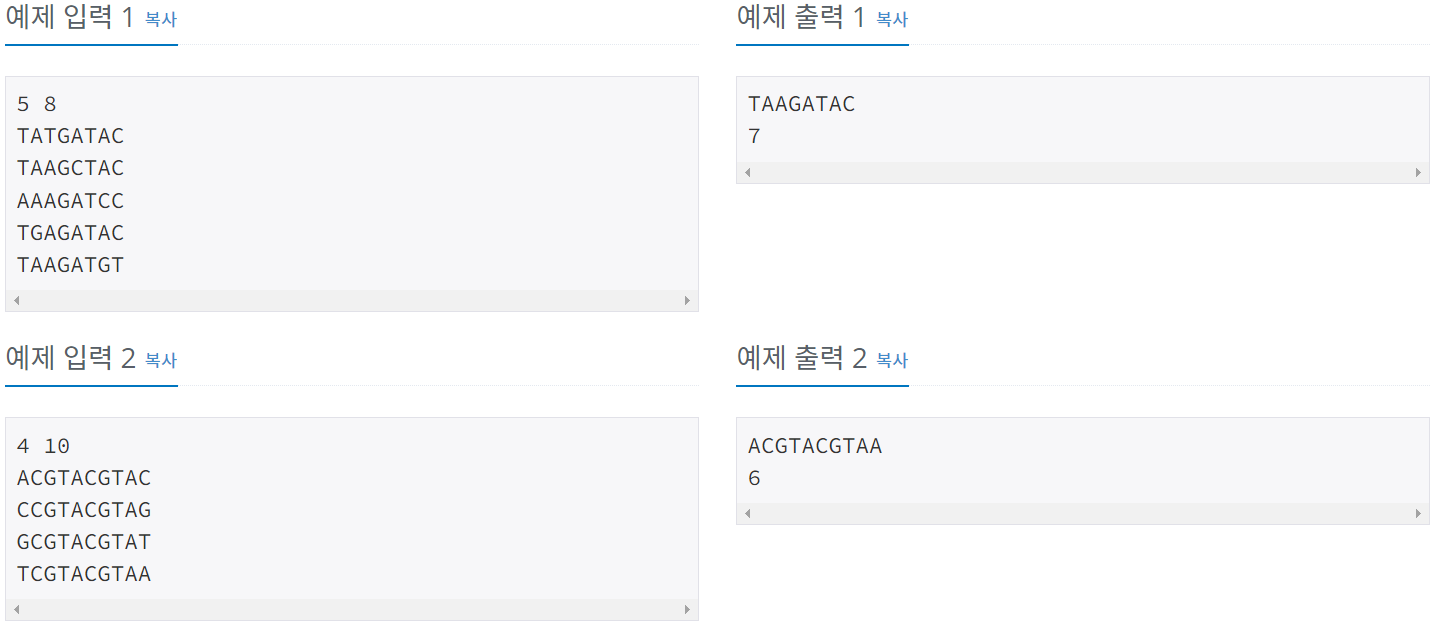
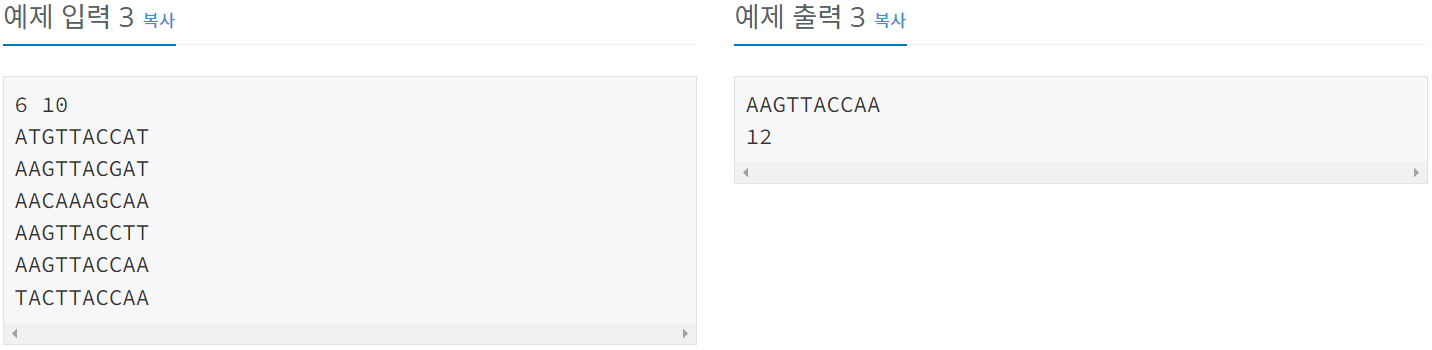
Algorithm
1. 리스트에 각 DNA를 문자열 형태로 저장
2. DNA의 각 INDEX 글자를 보면서 Dictionary에 해당 글자가 몇 번씩 나왔는지 Check
3. 가장 빈도가 높은 글자를 S에 추가
4. N - 빈도를 answer에 추가 : Hamming Distance
5. index 증가시키면서 2~4 반복 Code
# 입력
N, M = map(int, input().split())
DNA = []
for i in range(N):
DNA.append(input())
answer = [] # Hamming Distance의 합이 가장 작은 DNA를 저장할 List
count = 0 # Hamming Distance의 합
for i in range(M): # 문자열의 길이
frequency = {} # 각 문자의 빈도를 저장할 Dictionary
for j in range(N): # DNA 수
if DNA[j][i] not in frequency: # j번째 index에 있는 DNA가 frequency에 없으면
frequency[DNA[j][i]] = 1 # 생성
else: # 있으면
frequency[DNA[j][i]] += 1 # 빈도수 증가
# 최빈값 찾기
max_val = 0 # 최빈값을 저장할 변수
max_key = '' # 최빈값에 해당하는 DNA 저장할 변수
for key, value in frequency.items(): # key와 value를 확인하면서
if value > max_val: # 빈도가 높은 뉴클리오티드 문자와 빈도 저장
max_val = value
max_key = key
elif value == max_val: # 동일한 빈도를 갖는 뉴클리오티드 문자가 있다면 사전 순으로 앞서는 것 저장
if ord(max_key) > ord(key):
max_key = key
# 최빈값을 갖는 DNA와 N - 최빈값(Hamming Distance) 저장
answer.append(max_key)
count += N - max_val
print(''.join(answer)) # List를 문자열로 출력
print(count)메모리: 30864 KB
시간: 120 ms
