scikit-learn 개요
대표적인 Python Machine Learning(ML) Framework 중 하나다.
주의사항
- scikit-learn은 문자열 값을 처리할 수 없기 때문에 정수형으로 변경해야 한다.
2. 주요 Module
2.1. Data
- sklearn.datasets
scikit-laern에 내장되어 있어 외부에서 불러올 필요 없는 머신러닝 학습이 가능한 datasets를 제공한다.
load_iris
Iris(붓꽃)와 관련된, 분류를 위한 datasets
load_digits
이미지 픽셀과 관련된, 분류를 위한 datasets
load_breast_cancer
위스콘신 유방암과 관련된, 분류를 위한 datasets
load_diabets
당뇨병 관련된, 회귀를 위한 datasets
load_boston
미국 보스턴 집값과 관련된, 회귀를 위한 datasets
2.2.Feature
-
sklearn.preprocessing
데이터 전처리에 필요한 기능을 제공한다. 우선 결측치를 확인하고 적절한 값으로 대체하거나 삭제해야 한다. 일반적으로 평균 값을 사용하나 데이터를 왜곡할 가능성이 존재하기 때문에 적절한 대체값을 사용해야 한다. 또한 대부분의 값과 차이가 큰 값은 이상치로 처리하여 삭제할 것인지 그대로 포함시킬 것인지 등을 결정해야 한다.
Encoding
scikit_learn 데이터는 문자열을 처리할 수 없다. 따라서 문자열을 숫자로 바꾸는 전처리 작업이 필요하다. 불필요한 Feature는 삭제하고 필요한 Feature는 Feature vectorization(벡터화)해야 한다.
머신러닝에서 사용하는 대표적인 인코딩 방식은 Label encoding(레이블 이코딩)과 One Hot encoding(원 핫 인코딩) 방식이 있다.-
LabelEncoder
Category형 Feature를 숫자로 변환하는 것을 의미한다. 예컨대 iris datasets에서 target 값인['setosa' 'versicolor' 'virginica']를 [0, 1, 2]로 변환하는 방식이다.
그러나 Label encoding의 경우 숫자의 크고 적음이 머신러닝 학습에 영향을 끼칠 수 있다. 즉 단순 정수 값이 순서 또는 중요도로 작용할 수 있다. 따라서 선형회귀 등의 알고리즘에는 적용하면 안 된다. 반대로 Tree 계열의 알고리즘은 숫자의 특성을 반영하지 않으므로 Label encoding을 해도 학습에 영향이 없다.예제 코드
# 예제 데이터 exam = ["setosa", "versicolor", "virginica"] # LabelEncoder object 생성 l_encode = LabelEncoder() # fit을 통해 인코딩 수행 l_encode.fit(exam) # 인코딩 변환값을 label에 저장 label = l_encode.transform(exam) print("Label encoding value =", label) ## Label encoding Value = [0 1 2] # LabelEncoder는 transform의 결과로 numpy type을 반환한다. print("LabelEncoder transform 결과 type:", type(label)) ## LabelEncoder transform 결과 type: <class 'numpy.ndarray'> # 인코딩 된 값의 원본 값 확인 print("Original value =", l_encode.classes_) ## Original value = ['setosa' 'versicolor' 'virginica'] # Decoding print("Decoding([2, 2, 1, 0, 0]) =", l_encode.inverse_transform([2, 2, 1, 0, 0])) ## Decoding([2, 2, 1, 0, 0]) = ['virginica' 'virginica' 'versicolor' 'setosa' 'setosa']
-
OneHotEncoder
OneHotEncoder는 사용 전 문자열 값을 정수형 값으로 변환해야 한다. 따라서LabelEncoder와 함께 사용한다. 동시에OneHotEncoder입력 값은 2차원 데이터여야 한다. 최종적으로 2차원 행렬을 만들어서 columns 값에 해당하는 경우 1을 표시하고 나머지는 0을 표시한다.
그러나, 데이터가 많아질 수록 차원이 매우 증가할 수 있다는 한계점을 가진다.
반환되는 결과setosa versicolor virginica virginica 0 0 1 virginica 0 0 1 versicolor 0 1 0 setosa 1 0 0 setosa 1 0 0 OneHotEncoder(*, categories='auto', drop=None, sparse=True, dtype=<class 'numpy.float64'>, handle_unknown='error' )Method
fit(y)transform(y)inverse_transform(y)fit_transform(y)
fit과 transform 동시에 수행get_params(deep=True)set_params(**params)
예제 코드
# 예제 데이터 exam = ['virginica', 'virginica', 'versicolor', 'setosa', 'setosa'] # LabelEncoder object 생성 l_encode = LabelEncoder() # fit을 통해 인코딩 수행 l_encode.fit(exam) # 인코딩 변환값을 label에 저장 label = l_encode.transform(exam) print("Label encoding value =", label) ## Label encoding Value = [2 2 1 0 0] ############# One Hot Encoding ############# # LabelEncoder의 결과를 2차원 데이터로 변환 # # LabelEncoder는 transform의 결과로 numpy type을 반환하기 때문에 numpy.reshape()를 활용해 2차원으로 변환한다. # x는 1개 값만 가지게 만들고, y값은 그에 맞춰 할당한다. label = label.reshape([-1, 1]) print(label) ## [[2] ## [2] ## [1] ## [0] ## [0]] # OneHotEncoder object 생성 oh_encode = OneHotEncoder() # fit을 통해 인코딩 수행 oh_encode.fit(label) # 인코딩 변환값을 oh_label에 저장 # rows index와 1의 columns index를 반환한다. oh_label = oh_encode.transform(label) print(oh_label) ## (0, 2) 1.0 ## (1, 2) 1.0 ## (2, 1) 1.0 ## (3, 0) 1.0 ## (4, 0) 1.0 # oh_label의 type print(type(oh_label)) ## <class 'scipy.sparse.csr.csr_matrix'> # oh_label을 2차원으로 변환 print(oh_label.toarray()) ## [[0. 0. 1.] ## [0. 0. 1.] ## [0. 1. 0.] ## [1. 0. 0.] ## [1. 0. 0.]] # oh_label의 2차원 형태 print(oh_label.shape) ## (5, 3)
pandas.get_dummies
pandas는 더 쉬운 방법으로 One Hot Encodoing 변환을 지원한다.
pandas.get_dummies(data, prefix=None, prefix_sep='_', dummy_na=False, columns=None, sparse=False, drop_first=False, dtype=None )
예제 코드
# 예제 데이터 exam = ['virginica', 'virginica', 'versicolor', 'setosa', 'setosa'] # DataFrame으로 변환 df = pd.DataFrame({"iris": exam}) # OneHotEcondeing 형태로 변환 pd.get_dummies(df)
-
-
sklearn.preprocessing
Feature Scaling
Feature 변수의 값 범위를 일정한 수준으로 맞추는 것을 의미한다. 예컨대 자동차와 관련된 datasets에서 Feature로 [연비, 가격, 크기, 0-100] 등이 있다면 서로 다른 단위와 범위를 가진다.
* 주의사항
training datasets과 test datasets을 나누어서 Feature Scaling을 할 때는 주의해야 할 점이 존재한다. training datasets은fit()을 통해 데이터 변환을 위한 기준을 설정하고(표준화, 정규화 등), transform()을 통해 적용한다. 그런데 test datasets을fit()하게 되면 test datasets 만의 기준을 가지고 데이터가 변환된다. 따라서 test datasets은 training datasets에서fit()해놓은 것을 그대로 사용하여transform()만 하면 된다.
따라서 전체 datasets에 먼저 Feature Scaling을 적용한 후 datasets을 분리하는 것이 바람직하다.
1) 표준화(Standardization)
Feature 각각을 평균이 0이고 분산이 1인 가우시안 정규 분포 값으로 변환하는 것을 의미한다. Feature 의 i번 째 데이터가 일 때 표준화한 는 이다. 즉 평균에서 떨어진 거리에서 표준편차를 나눈 것이다.2) 정규화(Normalization)
Feature 각각을 최소값이 0이고 최대값이 1인 값으로 변환하는 것을 의미한다. Feature 의 i번 째 데이터가 일 때 표준화한 는 이다. 즉 min----------max의 일직선 상의 데이터 집합에서 의 위치를 계산한 값이라고 할 수 있다.
그러나, scikit learn에서 제공하는 Normalizer 모듈은 위에서 언급한 정규화와 차이가 존재한다. scikit learn의 Normalizer 모듈은 선형대수에서의 정규화 개념이 적용되어, 개별 vector의 크기를 맞추기 위해 변환한다. 따라서 개별 vector를 Feature vector의 크기로 나눈다. 예컨대 세 개의 Feature 가 있으면, 정규화한 s는 세 개의 Feature 의 합으로 나눈다. . 추가로 선형대수 개념의 정규화는 벡터 정규화(Vector Normalization)이라고 한다.-
StandardScaler
Feature값을 표준화하여 평균이 0이고, 분산이 1인 가우시안 정규 분포 값으로 변환한다. Support Vector Machine, Linear Regression(선형 회귀), Logistic Regression은 data가 가우시안 분포를 가지고 있다고 가정하고 구현되었기 때문에 가우시안 정규 분포로 데이터 값을 표준화해야 한다.StandardScaler(*, copy=True, with_mean=True, with_std=True )
예제 코드
# iris to DataFrame iris_df = pd.DataFrame(iris.data, columns=iris.feature_names) # iris datasets 내 각 feature의 평균 print("iris features mean:\n", iris_df.mean(), sep="") print("-" * 20, end="\n\n") ## iris features mean: ## sepal length (cm) 5.843333 ## sepal width (cm) 3.057333 ## petal length (cm) 3.758000 ## petal width (cm) 1.199333 ## dtype: float64 # iris datasets 내 각 feature의 분산 print("iris features variance:\n", iris_df.var(), sep="") ## iris features variance: ## sepal length (cm) 0.685694 ## sepal width (cm) 0.189979 ## petal length (cm) 3.116278 ## petal width (cm) 0.581006 ## dtype: float64 # StandardScaler object 생성 st_scaler = StandardScaler() # StandardScaler로 datasets 변환 iris_st_scaler = st_scaler.fit_transform(iris_df) # 변환한 값 출력 # print(iris_scaler) ## 표준화된 numpy.ndarray 반환 # numpy.ndarray 반환된 값 pandas.DataFrame으로 변환 iris_df_st_scaler = pd.DataFrame(iris_st_scaler, columns=iris_df.columns) # iris datasets 내 각 feature의 평균 print("iris features mean:\n", iris_df_st_scaler.mean(), sep="") ## iris features mean: ## sepal length (cm) -1.690315e-15 ## sepal width (cm) -1.842970e-15 ## petal length (cm) -1.698641e-15 ## petal width (cm) -1.409243e-15 ## dtype: float64 # iris datasets 내 각 feature의 분산 print("iris features variance:\n", iris_df_st_scaler.var(), sep="") ## iris features variance: ## sepal length (cm) 1.006711 ## sepal width (cm) 1.006711 ## petal length (cm) 1.006711 ## petal width (cm) 1.006711 ## dtype: float64 -
MinMaxScaler
Feature값을 정규화하여 0과 1사이의 범위 값으로 변환한다. 만약 음수 값이 포함되어 있으면 -1과 1사이의 범위 값으로 변환한다. 데이터의 분포가 가우시안 분포가 아닐 경우 사용할 수 있다.MinMaxScaler(feature_range=0, 1, *, copy=True, clip=False )
예제 코드
# MinMaxScaler object 생성 mm_scaler = MinMaxScaler() # MinMaxScaler로 datasets 변환 iris_mm_scaler = mm_scaler.fit_transform(iris_df) # numpy.ndarray 반환된 값 pandas.DataFrame으로 변환 iris_df_mm_scaler = pd.DataFrame(iris_mm_scaler, columns=iris_df.columns) # iris datasets 내 각 feature의 최솟값 print("iris features min:\n", iris_df_mm_scaler.min(), sep="") ## iris features min: ## sepal length (cm) 0.0 ## sepal width (cm) 0.0 ## petal length (cm) 0.0 ## petal width (cm) 0.0 ## dtype: float64 # iris datasets 내 각 feature의 최댓값 print("iris features max:\n", iris_df_mm_scaler.max(), sep="") ## iris features max: ## sepal length (cm) 1.0 ## sepal width (cm) 1.0 ## petal length (cm) 1.0 ## petal width (cm) 1.0 ## dtype: float64
-
-
sklearn.feature_selection
-
sklearn.feature_extraction
text 또는 image의 벡터화된 feature를 추출한다.
2.3. 차원 축소
- sklearn.decomposition
차원 축소와 관련된 알고리즘을 지원한다.- PCA
- NMF
- Turncated SVD
2.4. 데이터 분리/검증, 파라미터 튜닝
- sklearn.model_selection
교차 검증을 위한 학습용/테스트용 데이터 세트를 분리하고, Grid Search를 통해 최적 파라미터를 추출한다.
training_test_split
datasets를 traininging(학습)할 데이터와 검증에 사용될 Test 데이터로 분리한다.training_test_split(*arrays, # feature datasets와 label datasets 할당 test_size=None, # datasets에서 test로 분류할 데이터의 비율(0.00 ~ 1.00) # None이면, default 값인 0.25(1/4) 할당 training_size=None, # None이면 자동으로 (1 - test_size) 값 할당 random_state=None, # 데이터를 섞을 때 Seed로 사용할 int 값 shuffle=True, stratify=None # 데이터를 나눌 때 target(label)이 일정한 비율로 분류 )- random_state
training datasets와 test datasets로 나누기 전 raw datasets을 shuffle(무작위로 섞음)한다. 이 때 섞는 기준(Seed)를 int값으로 주게 되면, 실행할 때마다 training datasets로 분류되는 데이터와 test datasets로 분류되는 데이터가 고정된다. 모델을 개선하기 위해 고정된 값을 부여하면 개선 정도를 파악하기 용이하다.
- stratify
stratify=None이 아니면 raw datasets를 training datasets와 test datasets로 나눌 때 raw_data의 target(label) 비율에 맞춰 데이터를 나눈다.
예컨대 label이A,B,C가 있을 때n(A) = 45,n(B) = 45,n(C) = 10일 때 stratify가 None인 상태로 실행하게 되면 test datasets에C가 10개 모두 포함될 수 있다. 이렇게 되면C에 대해 학습하지 않았기 때문에 ML모델은 C로 분류할 수 없다.
반면,test_size=0.5일 때 stratify에 target(label)을 할당하면
training datasets에는n(A) = 23,n(B) = 22,n(C) = 5
test datasets에는n(A) = 22,n(B) = 23,n(C) = 5로 나뉘게 된다.
*n(K)는K의 개수를 의미한다.
- random_state
KFold
training datasets를 K개의 fold로 나눈 후 K-1개는 training datasets로 사용하고, 1개의 fold는 validation dataset를 사용해 Cross Validation(교차 검증)하는 방법이다. 이 때 K개의 fold는 한 번씩 번갈아가면서 validation dataset이 되기 때문에 검증은 K번 수행된다.
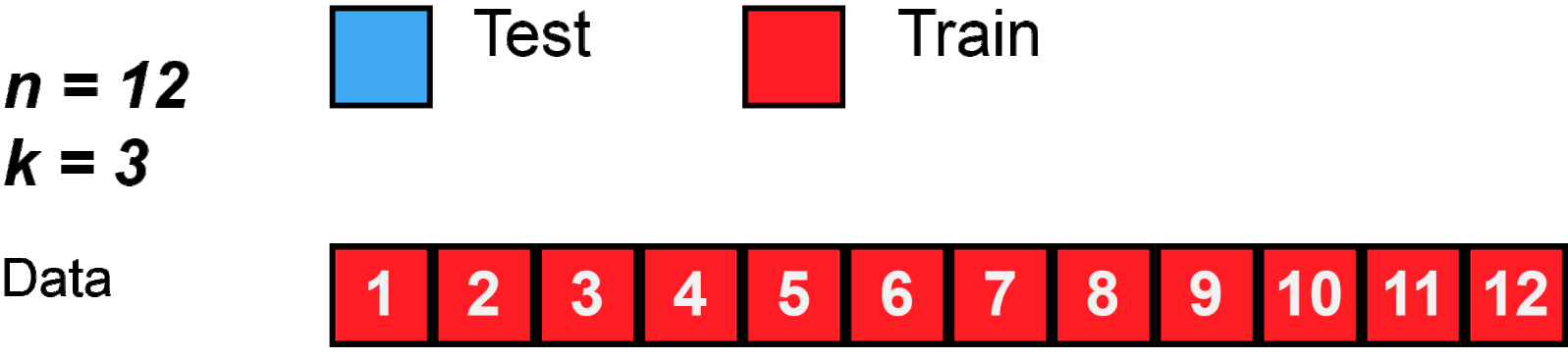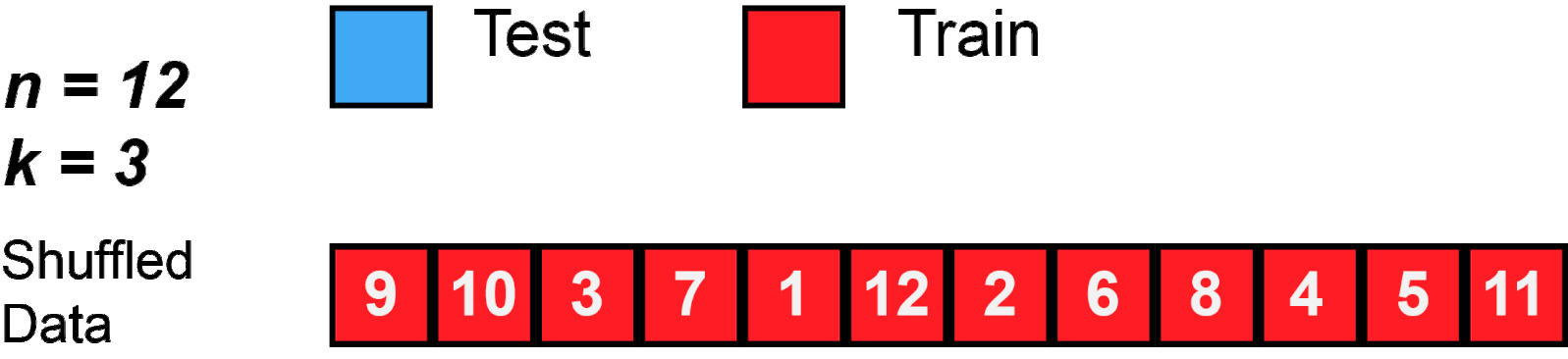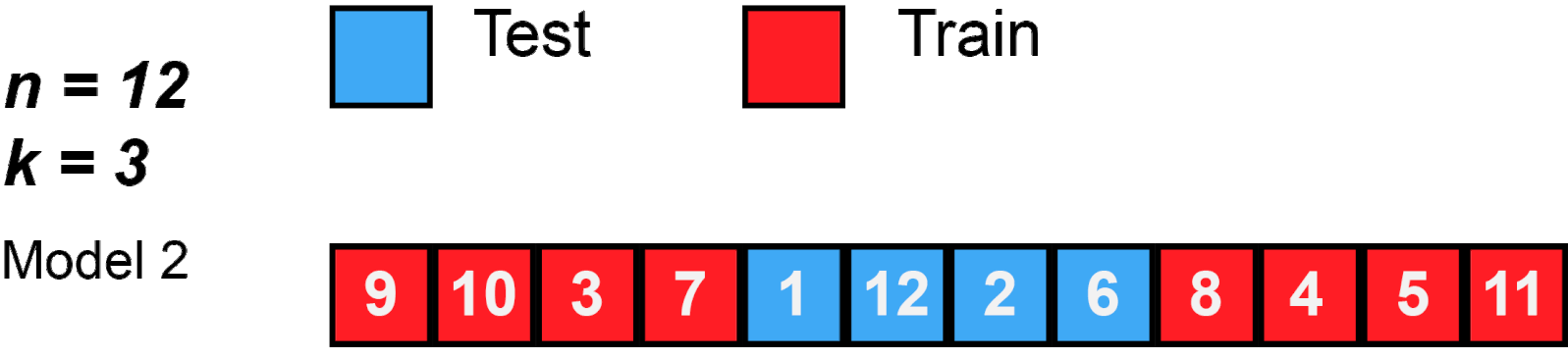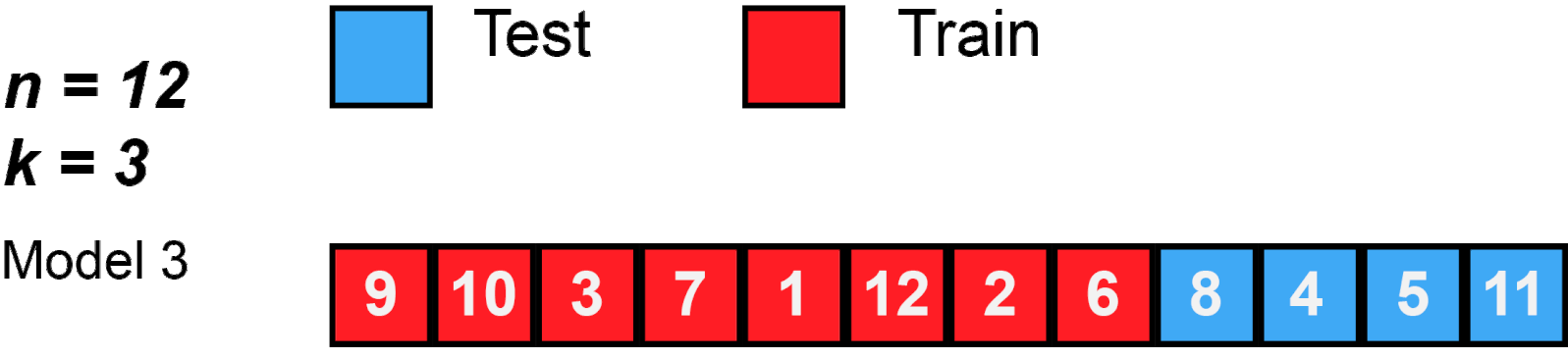
KFold([n_splits=5, # fold로 나눌 개수 *, shuffle=False, random_state=None] )
StratifiedKFold
KFold로 fold를 나누면 데이터의 분포가 일정하지 않다. 머신러닝 모델 학습 시 raw datasets의 데이터 분포를 제대로 반영하지 못하기 때문에 신뢰성있는 결과를 얻을 수 없으며, 최악의 경우 하나의 데이터 집단이 완전히 빠져있는 경우가 있을 수 있다.
예컨대 1,000개의 datasets에서 label값이 n(0) = 995, n(1) = 5이고 K가 5이면 어느 한 쪽에만 1인 데이터가 몰려있을 수 있다.
따라서 일반적으로 Classification(분류)에서는 StratifiedKFold를 사용해 데이터의 분포를 일정하게 맞추는 것이 바람직하다.
그러나 Regression(회귀)에서는 StratifiedKFold를 지원하지 않는다. 왜냐하면 회귀의 경우 연속된 값이므로, 데이터 분포를 고려할 필요가 없기 때문이다.StratifiedKFold(n_splits=5, *, shuffle=False, random_state=None )
cross_val_score
cross_val_score는 학습(fit), 예측(predict), 평가(evaluation)를 자체적으로 수행하기 때문에 코드가 간결하다. Classification(분류)에서는 StratifiedKFold 방식을 사용하며, Regression(회귀)에서는 KFold 방식을 사용한다.cross_val_score(estimator, # ML Algorithm Class object X, # Feature datasets y=None, # Label datasets *, groups=None, scoring=None, # 성능 평가 지표 cv=None, # 교차검증(cross-validation) Fold 개수 n_jobs=None, verbose=0, fit_params=None, pre_dispatch='2*n_jobs', error_score=nan )
cross_validate
여러개의 평가 지표를 반환한다.
GridSearchCV
최적의 하이퍼 파라미터 조합을 찾고, 하이퍼 파라미터 값으로 모델을 학습킨 결과값을 반환한다. 이때 parameter값과 cv값의 곱 만큼 계산을 반복하게 된다.GridSearchCV(estimator, # ML Algorithm Class object param_grid, # Estimator 튜닝을 위해 사용할 하이퍼 파라미터 값 *, scoring=None, # 성능 평가 지표 n_jobs=None, refit=True, True이면 도출된 최적 하이퍼 파라미터 값으로 Estimator 재학습 cv=None, # 교차검증(cross-validation) Fold 개수 verbose=0, pre_dispatch='2*n_jobs', ' error_score=nan, return_train_score=False)Attributes
cv_results_
GridSearchCV의 결과를 딕셔너리 형태로 저장한 datasets이다.
best_params_
최적의 하이퍼 파라미터 값
best_score_
최적의 하이퍼 파라미터 값을 적용한 fold 정확도의 평균
best_estimator
최적의 하이퍼 파라미터 값을 적용한 Estimator(ML Algorithm)
2.5. 성능 평가
- sklearn.metrics
분류, 회귀, clustering, Pairwise 등 성능 평가 방법을 제공한다.
accuracy_score
전체 중 맞은 것을 비율인 Accuracy(정확도)를 반환한다.accuracy_score(y_true, # test label y_pred, # ML 알고리즘으로 학습한 모델로 예측한 결과값 *, normalize=True, sample_weight=None)- Precision
- Recall
- ROC-AUC
- RMSE
2.6. ML Algorithm
-
sklearn.ensemble
앙상블 알고리즘을 제공한다.- Random Forest
- AdaBoost(에이다 부스팅, Adaptive Boosting)
- Gradient Boosting
-
sklearn.linear_model
회귀 및 SGD(Stochastic Gradient Descent) 알고리즘을 제공한다.- Linear Regression(선형 회귀)
- Ridge
- Lasso
- Logistic Regression
-
sklearn.naive_bayes
Naive Baye(나이브 베이즈) 알고리즘을 제공한다.- Gaussian Naive Baye(가우시안NB)
- Multinomial Distribution Naive Baye(다항분포NB)
-
sklearn.neighbors
최근접 이웃 알고리즘을 제공한다.- K-NN
-
sklearn.svm
Support Vector Machine(SVM) 알고리즘을 제공한다. -
sklearn.tree
Decision Tree(의사 결정 트리) 알고리즘을 제공한다.
DecisionTreeClassifierDecisionTreeClassifier(*, criterion='gini', splitter='best', max_depth=None, min_samples_split=2, min_samples_leaf=1, min_weight_fraction_leaf=0.0, max_features=None, random_state=None, max_leaf_nodes=None, min_impurity_decrease=0.0, min_impurity_split=None, class_weight=None, ccp_alpha=0.0 )Method
fit
tree 알고리즘으로 머신러닝 모델을 학습시킨다.
fit(X, # training feature datasets y[, # training label sample_weight=None, check_input=True, X_idx_sorted='deprecated'] )predict
학습시킨 모델을 바탕으로X를 예측한다.
predict(X[, # test feature datasets check_input=True])
- sklearn.cluster
Unsupervised Clustering 알고리즘을 제공한다.- K-Means
- Hierarchical
- DBSCAN
2.7. Utility
- sklearn.pipeline
Feature 처리, ML 알고리즘 학습, 예측 등을 묶어서 실행할 수 있는 유틸리티를 제공한다.