IPython에는 Python code의 퍼포먼스, 메모리 사용, 수행 시간등을 편리하게 프로파일링 하게 도와주는 여러 Magic Command들이 있다
- Magic Command가 뭔지 모른다면 아래 글을 참고
Jupyter Notebook에서 Magic Command 사용하기
%time: 주어진 코드를 한 번 실행했을 때 수행시간을 구한다%timeit: 주어진 코드를 여러 번 실행한 후, 그 평균 수행시간을 구한다.%time보다 더 정확하다%prun: 프로파일러와 함께 코드를 실행한다%run -p [prof_opts] filename.py [args to program]: 전체 .py파일을 프로파일러와 함께 실행한다%lprun: 코드 한 라인씩 분석되는 프로파일러와 함께 코드를 실행한다%%heat: 주어진 코드의 시간 사용 비율을 heat map형태로 보여준다%memit: 주어진 코드의 메모리 사용량을 계산한다%mprun: 코드 한 라인씩 분석되는 메모리 프로파일러와 함께 코드를 실행한다
%time 과 %timeit 의 경우 IPython에 기본으로 포함되어있지만, %prun 과 %lprun 은 line_profiler 모듈을, %memit 과 %mprun의 경우 memory_profiler 모듈을 설치해 주어야 한다
코드 수행시간 분석하기 : %time, %timeit
%time 은 코드가 실행되는데 걸리는 시간을 측정해준다
In [13]: %time sum(range(100))
Wall time: 641 µs
Out [13]: 4950%timeit은 주어진 코드를 여러번 실행해보고 그 평균시간을 알려준다
In [14]: %%timeit
a = 1
b = 1
sum = 0
for i in range(10):
sum += a
a,b = b, a+b
2.33 µs ± 798 ns per loop (mean ± std. dev. of 7 runs, 100000 loops each)%timeit 에는 여러 옵션들이 존재한다
- -n : 한 루프당 몇 번 실행할 것인지 지정한다. 지정하지 않으면 알아서 적당히 충분히 정확한 데이터가 나올 정도로 실행함
- -r :
%timeit은 여러번 반복해서 그 평균값과 오차범위를 보여주는데, 몇 번 반복할 것인지를 지정할 수 있다. 지정하지 않으면 기본값으로 7번 반복한다.
-n과 -r이 어떻게 다른가요?
예를들어 %timeit -n 100 -r 3 로 설정을 하면
주어진 코드는 100(loops) * 3(runs)로 총 300번 실행이 된다
즉 1번의 run마다 100번씩 실행을 하는데, 100번중 가장 최상의 결과(the best result)를 저장한다
그리고 총 3번의 run을 통해 best result가 3개 생기고, 그것들의 평균을 구해 표준편차와 함께 보여주게 된다

위 그림을 보면 100번의 loop를 돌려도 단 1번만 run을 했을 경우에는 표준편차값이 없다
반면 1번씩 실행하는것으로 100번의 run을하면 그 100번의 데이터를 통계적으로 분석하는 과정에서 표준편차값이 존재하게 된다
- -t : 시간계산에
time.time모듈을 사용하게 된다. Unix 시스템에서 디폴트값이다 - -c : 시간계산에
time.clock모듈을 사용하게 된다. Windows 시스템에서 디폴트값이다. Unix시스템에서 이 옵션을 주면 Wall time(사람이 인지할 수 있는 시간) 대신 CPU time으로 계산된다 - -p : 소숫점 몇자리까지 표시되는지 설정할 수 있다. 디폴트값은 3이다
- -q : Quiet, 즉 결과값이 출력되지 않는다
- -o : 결과를 TimeitResult객체로 반환한다
In [21]: a = %timeit -o sum(range(100))
2.75 µs ± 199 ns per loop (mean ± std. dev. of 7 runs, 100000 loops each)
In [22]: type(a)
Out [22]: IPython.core.magics.execution.TimeitResult전체 스크립트 프로파일링 하기 : %prun
%prun 은 profile모듈을 사용하여 주어진 코드를 프로파일링한다
참고로 IPython에선 %prun magic command가 아닌, profile.run() 를 통해 프로파일링 하는것이 불가능하다. 왜냐면 profile.run() 은 내부적으로 특정 네임스페이스를 가정해서 실행하는데 IPython에서는 그런 가정을 지원하지 않기 때문이다
%prun 이 어떻게 동작하는지 알기위해 일단 아래와 같은 함수가 있다고 가정해보자
def sum_of_lists(N):
total = 0
for i in range(5):
L = [j ^ (j >> i) for j in range(N)]
total += sum(L)
return total%prun sum_of_lists(10000) 의 결과는 다음과 같다
14 function calls in 0.010 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
5 0.009 0.002 0.009 0.002 <ipython-input-19-f105717832a2>:4(<listcomp>)
5 0.001 0.000 0.001 0.000 {built-in method builtins.sum}
1 0.000 0.000 0.010 0.010 <ipython-input-19-f105717832a2>:1(sum_of_lists)
1 0.000 0.000 0.010 0.010 <string>:1(<module>)
1 0.000 0.000 0.010 0.010 {built-in method builtins.exec}
1 0.000 0.000 0.000 0.000 {method 'disable' of '_lsprof.Profiler' objects}- ncalls : total number of calls. 함수가 몇 번 실행되었는지
- tottime : (내부함수를 제외하고) interpreter가 함수를 수행한 시간
- cumtime : (내부함수를 포함하고) interpreter가 함수를 수행한 시간
- percall : tottime과 cumtime를 각각 실행 횟수(ncalls)로 나눈 값
- filename:lineno(function) : 함수 이름과 파일 번호
위 결과를 조금 더 자세히 들여다 보자면, sum_of_lists함수에서 총 0.010초가 수행이 되었고, 또한 sum_of_lists함수 내부에서 list comprehension과 built-in 함수인 sum()이 5번씩 수행이 되었으며, 각각 수행시간이 0.009초, 0.001초이다. sum_of_lists함수의 tottime 은 0이고 cumtime은 0.010이므로 대부분의 수행은 내부함수인 list comprehension과 sum()에서 이루어졌음을 알 수 있다
%prun역시 여러 옵션을 지원한다
-
-l : 프로파일러를 통해 무엇을 얼마나 출력할지 제한할 수 있다
영역에 어떤 데이터 타입이 오느냐에 따라 제한 내용이 바뀐다
-
string : filename:lineno(function)의 내용을 필터링한다. 예를들어 sum이라는 값을 주면 위 결과값 중 :1(sum_of_lists)과 {built-in method builtins.sum}행만 출력된다
-
int : 결과를 몇 줄 출력할지 제한한다
-
float(0~1) : 전체 분석결과중 몇 %나 출력할지 제한한다. 예를들어 0.4면 내용중 40%만 출력된다
제한 옵션은 여러개 섞어 쓸 수 있다
예를들어
%prun -l sum -l 1 sum_of_lists(10000)의 결과는 다음과 같다
-
14 function calls in 0.006 seconds
Ordered by: internal time
List reduced from 6 to 2 due to restriction <'sum'>
List reduced from 2 to 1 due to restriction <1>
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.001 0.001 0.006 0.006 <ipython-input-19-f105717832a2>:1(sum_of_lists)-
-r : 결과를 출력하는 대신에 pstats.Stats객체의 형태로 반환한다
-
-s : 주어진 key에 따라 결과를 정렬한다. 숫자와 관련된건 내림차순(큰 수가 앞에), 문자와 관련된건 오름차순이다(abc...xyz)
- calls : ncalls 수에따라 정렬한다
- time : tottime 에 따라 정렬한다
- cumulative : cumtime에 따라 정렬한다
- module : filename에 따라 정렬한다(공식문서에file또한 filename으로 정렬이라고 되어있으나, 실제로 옵션 인수로file을 주면 오류가 발생한다)
- pcalls : 재귀(recursion)로 호출된 횟수를 제외한 함수 실행 횟수에 따라 정렬한다
- line : lineno에 따라 정렬한다
- name : function에 따라 정렬한다
- nfl : name/file/line순에 따라 정렬한다
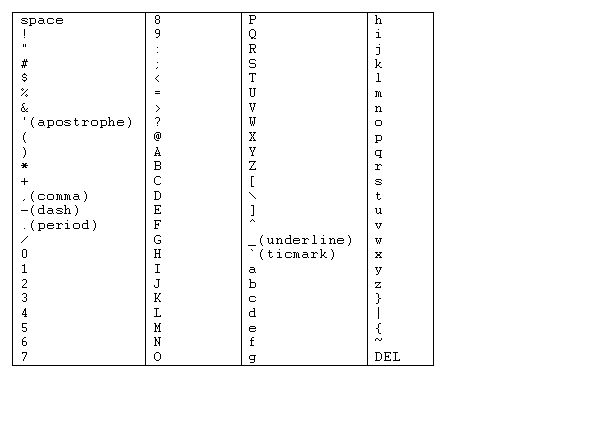
- stdname : filename:lineno(function)열 전체를 하나의 문자열로 간주하고 정렬한다. 아래 표 에 따라 '<'문자의 경우 '{'보다 앞에 오기 때문에, :1(sum_of_lists)가 {built-in method builtins.sum}보다 먼저 오게된다
-
-T : 프로파일 결과를 파일로 저장한다(위치는
C:\Users\계정이름\Documents). 만약 해당 이름의 파일이 이미 있다면 덮어쓴다. 저장하는것과 별개로 프로파일 결과도 여전히 화면에 띄워준다 -
-D : 프로파일 결과를 바이너리 파일로 저장한다
-T옵션과 비교해보자
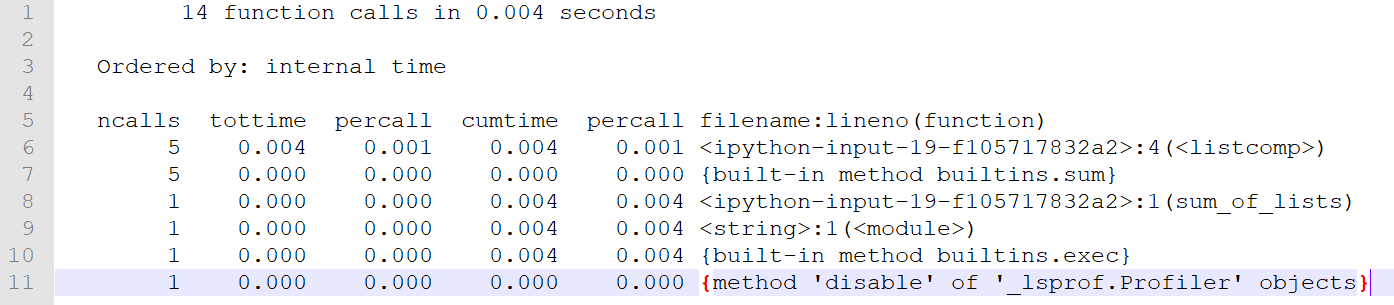
%prun -T profile_test_-T sum_of_lists(10000)로 생성된 profiletest-T파일을 Notepad++로 열었을때 아래와 같이 나온다
반면%prun -D profile_test_-D sum_of_lists(10000)로 생성된 profiletest-D파일을 똑같이 열어보면 아래와 같은것을 볼 수 있다
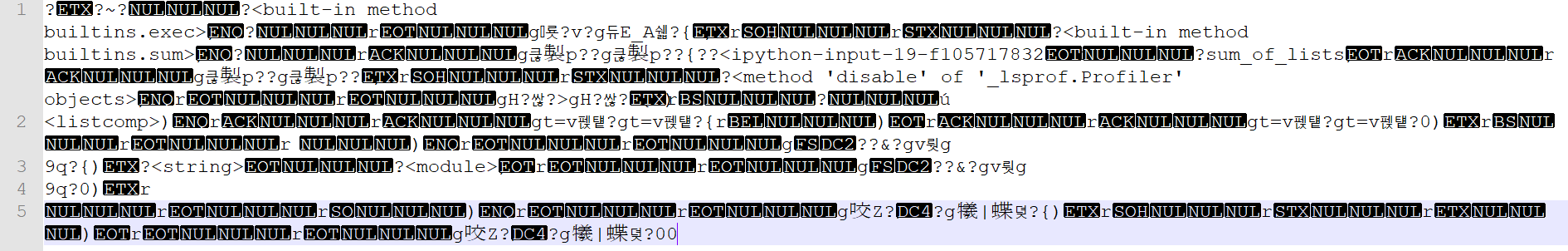
필요에 따라 알아서 Hex editor등으로 사용하면 되겠다 -
-q : 프로파일 결과를 jupyter에서 표시하지 않는다. -T나 -D옵션처럼 프로파일 결과를 다른곳에 저장하는 경우 함께쓰면 유용하다
코드 한 줄씩 프로파일링 하기 : %lprun
%lprun magic command를 사용하기 위해서는 line_profiler라는 모듈이 필요하다. 콘솔창에서 pip install line_profiler 로 설치하거나, 아니면 나처럼 아나콘다에서 설치하면 된다
line_profiler모듈을 설치하였으면 jupyter창에 %load_ext line_profiler 를 입력해주면 %lprun 를 쓸 준비가 끝난다
%lprun -f func1 -f func2 <statement1>;<statement2>%lprun 을 쓰기위해서는 무조건 -f 옵션으로 프로파일할 함수나 메소드를 지정해야 한다. -f옵션을 여러개 줘서 동시에 여러 함수를 비교해 볼 수 있다(당연히 하나씩만 할 수도 있음)
위에서 예로 들었던 sum_of_lists함수 외에 아래와 같은 fibo라는 함수도 있다고 생각해보자
def fibo(n):
a = 0
b = 1
allsum = 0
for i in range(n):
allsum += a
a, b = b, a+b
return allsum그리고 Jupyter에서 아래와 같이 실행해보자
%lprun -f sum_of_lists -f fibo fibo(100);sum_of_lists(100)결과는 아래와 같이 출력된다
Timer unit: 1e-07 s
Total time: 0.0001425 s
File: <ipython-input-2-f105717832a2>
Function: sum_of_lists at line 1
Line # Hits Time Per Hit % Time Line Contents
==============================================================
1 def sum_of_lists(N):
2 1 6.0 6.0 0.4 total = 0
3 6 39.0 6.5 2.7 for i in range(5):
4 5 1278.0 255.6 89.7 L = [j ^ (j >> i) for j in range(N)]
5 5 95.0 19.0 6.7 total += sum(L)
6 1 7.0 7.0 0.5 return total
Total time: 0.0001164 s
File: <ipython-input-43-17e1f071b390>
Function: fibo at line 1
Line # Hits Time Per Hit % Time Line Contents
==============================================================
1 def fibo(n):
2 1 15.0 15.0 1.3 a = 0
3 1 6.0 6.0 0.5 b = 1
4 1 4.0 4.0 0.3 allsum = 0
5 101 336.0 3.3 28.9 for i in range(n):
6 100 380.0 3.8 32.6 allsum += a
7 100 418.0 4.2 35.9 a, b = b, a+b
8
9 1 5.0 5.0 0.4 return allsum어느 부분에서 얼마나 시간이 걸리고 얼마나 수행되는지 보기 좋게 출력된다
결과 표에서 % time 열은 총 수행시간 대비 해당 라인이 수행된 시간의 퍼센테이지를 의미한다. sum_of_lists함수의 경우 대부분의 시간(89.7%)이 list comprehension에서 소모됨을 알 수 있다
%lprun 역시 여러 옵션이 있다
- -m : 해당 모듈내에 있는 모든 함수와 메소드들을 전부 보여준다. 파이썬 공식문서처럼 함수 이름과 사용법이 나오는게 아니라 그냥 함수 코드 그 자체를 다 보여준다. 내부함수도 포함된다. 평소 사용하던 모듈의 소스코드가 궁금하다면 사용해 보자
- -D 또는 -T : 위
%prun항목을 참고하자. 동일하게 수행되지만 유일한 차이점은lprun에서는 결과값을 Jupyter상에 표시해주지 않는다 - -r : 결과값을 line_profiler.LineProfiler 오브젝트 형태로 저장할 수 있다
lprun_test_result = %lprun -f sum_of_lists -f fibo -r fibo(100);sum_of_lists(100)- -u : 시간 단위를 설정할 수 있다. 예를들어 위 결과는 기본값으로 1e-07초(100나노초)단위로 표시되었지만, 만약 1나노초 단위로 보고싶다면
-u 0.000000001와 같은 옵션을 주면 된다
%%heat
프로파일 결과를 텍스트가 아닌 좀 더 직관적인 형태로 보여주는 Magic Command이다
사용하기 위해선 먼저 pip install py-heat-magic 로 설치를 한다. 그 후 Jupyter상에서 %load_ext heat 를 입력하여 사용할 준비를 마친다
좀 귀찮지만 %%heat 를 사용하기 위해서는 함수 실행부분 뿐만이 아니라 함수 정의까지 입력해 주어야 한다
위에서 보았던 fibo함수를 예를들어 설명해보자
%%heat
def fibo(n):
a = 0
b = 1
allsum = 0
for i in range(n):
allsum += a
a, b = b, a+b
if (b < a):
b += 1
return allsum
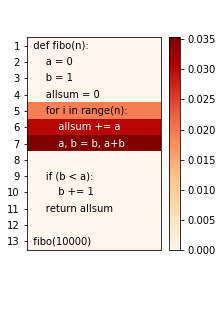
fibo(10000)위와 같이 입력하면 아래와 같은 결과물이 나온다
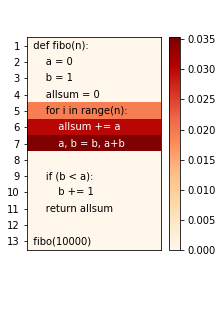
fibo 함수에서 for문 속 구문을 수행하는데 가장 오랜 시간이 걸리는 것을 heat map형태로 보여준다
%lprun에서 보았던것과 결과가 거의 비슷하게 나옴을 알 수 있다
- -o :
%%heat의 유일한 옵션이다. 결과물을 이미지 파일로 저장할 수 있다.%%heat -o file.png와 같이 사용하면 된다. 물론 이 옵션을 사용하지 않고 브라우저 상에서 '이미지를 다른 이름으로 저장' 을 사용할수도 있다
일단 너무 길어져서 메모리편은 나눠서 써야겠다
출처
https://jakevdp.github.io/PythonDataScienceHandbook
https://ipython-books.github.io/
https://ipython.readthedocs.io/en/stable/interactive/magics.html
https://nyu-cds.github.io/python-performance-tuning/02-cprofile/
https://towardsdatascience.com/speed-up-jupyter-notebooks-20716cbe2025
