📌 scikit-learn 라이브러리를 활용한 지도 학습
✏️ 분류는 종속변수(y, 정답, target)가 이진과 다중으로 나누어지는데 이는 연속적인 값이 아닌 범주형(type, class...)으로 오지선다 같은거. 회귀는 연속적인 값.
✏️ 이진분류는 예/아니오 식으로 나올 수 있도록 하는 것. 셋 이상의 클래스는 다중분류 예/아니오 식의 정답이 아니라 특정 정답이 나올 수 있는 것.
✏️ 독립변수는 x, Feature, Data라고 많이 함. 회귀는 부동소수점수(실수)를 예측하는 것.
✏️ 일반화 성능이 최대가 되는 모델이 최적. 모델이 복잡할수록(학습을 많이 시키면) 과대적합상태가 되어 새로운 데이터를 만났을 때 일반화되지 못한다.
✏️ k-최근접 이웃 알고리즘 - 가장 가까운 훈련 데이터 포인트를 최근접 이웃으로 찾아 예측에 사용.
✅ 예제
✔️ iris(붓꽃) 품종 분류
▶️ 독립변수(x, feature, data) : 꽃잎, 꽃받침의 길이(cm) 4가지(length, width)
▶️ 종속변수(y, class, target) : 꽃의 품종(setosa, virginica, versicolor)
(+ 꽃잎, 꽃받침 길이에 따른 아이리스 품종 분류이기 때문에 꽃잎, 꽃받침 길이가 독립변수가 되고 품종이 종속변수가 되는 것)
✏️ 데이터 준비하기
# 데이터 준비하기
from sklearn.datasets import load_iris
iris_dataset = load_iris()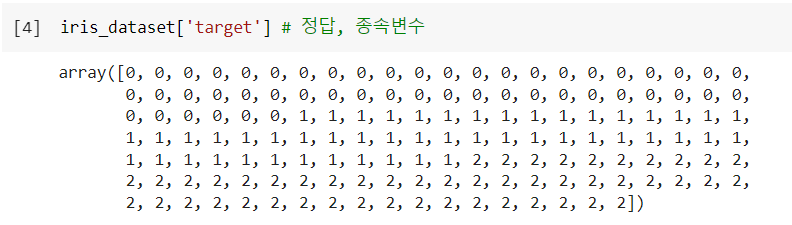

데이터 확인하면 numpy 배열형식으로 값이 입력되어있는 것을 알 수 있다.

shape을 이용해서 확인하면 행렬 형식(데이터프레임으로 생각하면 row가 150개인거고 column이 4개인것.)
✏️ 산점도 그래프 그리기
import matplotlib.pyplot as plt
import pandas as pd
# 데이터프레임을 사용하여 데이터 분석 -> 독립변수(feature)와 종속변수(label)의 연관성을 확인
iris_df = pd.DataFrame(iris_dataset['data'], columns=iris_dataset.feature_names)
# 각 독립변수(feature)들의 산점도 행렬 4x4
pd.plotting.scatter_matrix(iris_df, c=iris_dataset['target'], figsize=(15,15),
marker='o', hist_kwds={'bins':20}, s=60, alpha=.8)
plt.show()pandas 데이터프레임 형식으로 불러와서 산점도 차트를 그려보면
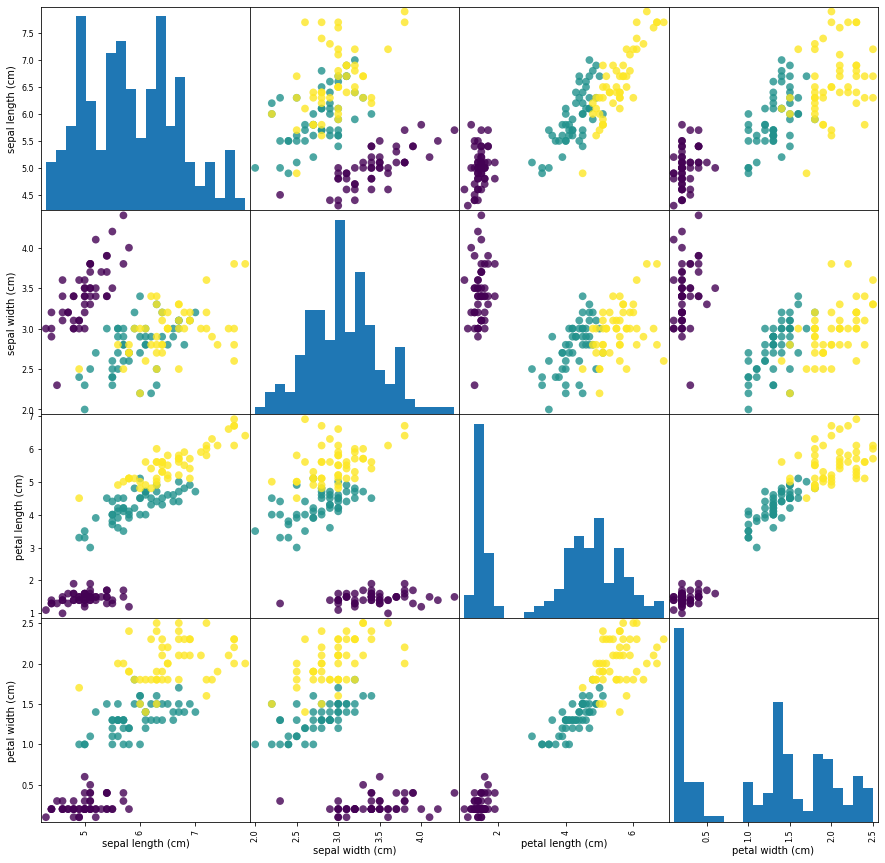
각 변수들 간의 분포형태를 확인할 수 있다.
x축이 petal length이고 y축이 petal width인 산점도가 가장 아이리스 품종이 눈에 띄게 분류되어있다.
import numpy as np
plt.imshow([np.unique(iris_dataset['target'])])
_ = plt.xticks(ticks=np.unique(iris_dataset['target']), labels=iris_dataset['target_names']) # underscore -> _(값을 출력하고 싶지 않을 때 사용, 변수에 담으면 출력이 안되니까)
종속변수(target)의 변수값이 어떻게 입력되어있는지 확인.
setosa가 0, versicolor가 1, virginica는 2.
iris_df2 = iris_df[['petal length (cm)', 'petal width (cm)']]위에서 가장 적합했던 산점도의 변수만 뽑아서 데이터프레임 새로 형성.
pd.plotting.scatter_matrix(iris_df2, c=iris_dataset['target'], figsize=(15,15),
marker='o', hist_kwds={'bins':20}, s=60, alpha=.8)
plt.show()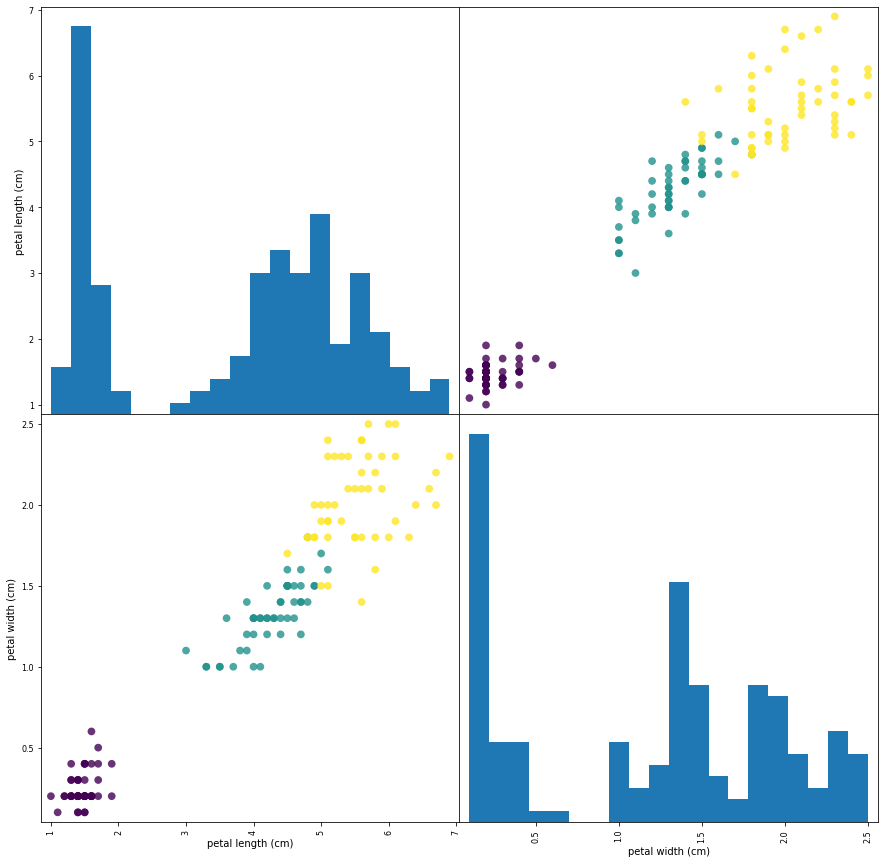
산점도를 그리면 2X2 형태로 그려짐
✏️ 훈련데이터와 테스트데이터 분리
# 훈련데이터 : 테스트 데이터 -> 7:3 or 75:25 or 80:20 or 90:10 비율.
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(iris_dataset['data'], iris_dataset['target'], # 대문자 -> 2개 이상일 때, 소문자 -> 1개
test_size=0.25, random_state=777) # random_state 고정된 시드부여
# 훈련데이터 확인하기 150 => 75% -> 112개
X_train.shape
# 테스트데이터 확인하기 150 => 25% -> 38개
X_test.shape✏️ 머신러닝 모델 설정 -> k-최근접 이웃 알고리즘
from sklearn.neighbors import KNeighborsClassifier
knn = KNeighborsClassifier(n_neighbors=1) # 이웃의 개수 1개로 지정
# 학습하기
knn.fit(X_train, y_train)
# 예측하기
y_pred = knn.predict(X_test)✏️ 모델 평가하기
# 정확도 확인하기
# 1) mean() 함수 사용해서 정확도 확인
np.mean(y_pred == y_test)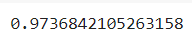
🔼 결과
# 2) score() 함수를 사용해서 정확도 확인 -> 테스트 셋으로 예측한 후 정확도 출력
knn.score(X_test, y_test)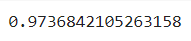
🔼 결과
# 3) 평가 지표 계산
from sklearn import metrics
knn_report = metrics.classification_report(y_test, y_pred)
print(knn_report)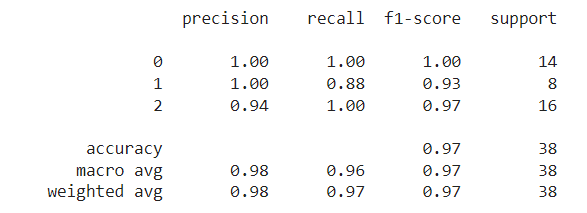
🔼 결과
✔️ forge
▶️ 인위적으로 만들어진 이진분류 데이터셋
# 설치
pip install mglearn
# 이진 분류 데이터셋 확인하기
import mglearn
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings('ignore')
# 데이터셋 다운로드
X , y = mglearn.datasets.make_forge()
# 데이터 확인하기
print('X.shape : ', X.shape)
print('y.shape : ', y.shape)# 산점도 그리기
plt.figure(dpi=100)
plt.rc('font', family='NanumBarunGothic')
mglearn.discrete_scatter(X[:,0], X[:,1], y)
plt.legend(['클래스 0', '클래스 1'], loc=4)
plt.xlabel('첫 번째 특성')
plt.ylabel('두 번째 특성')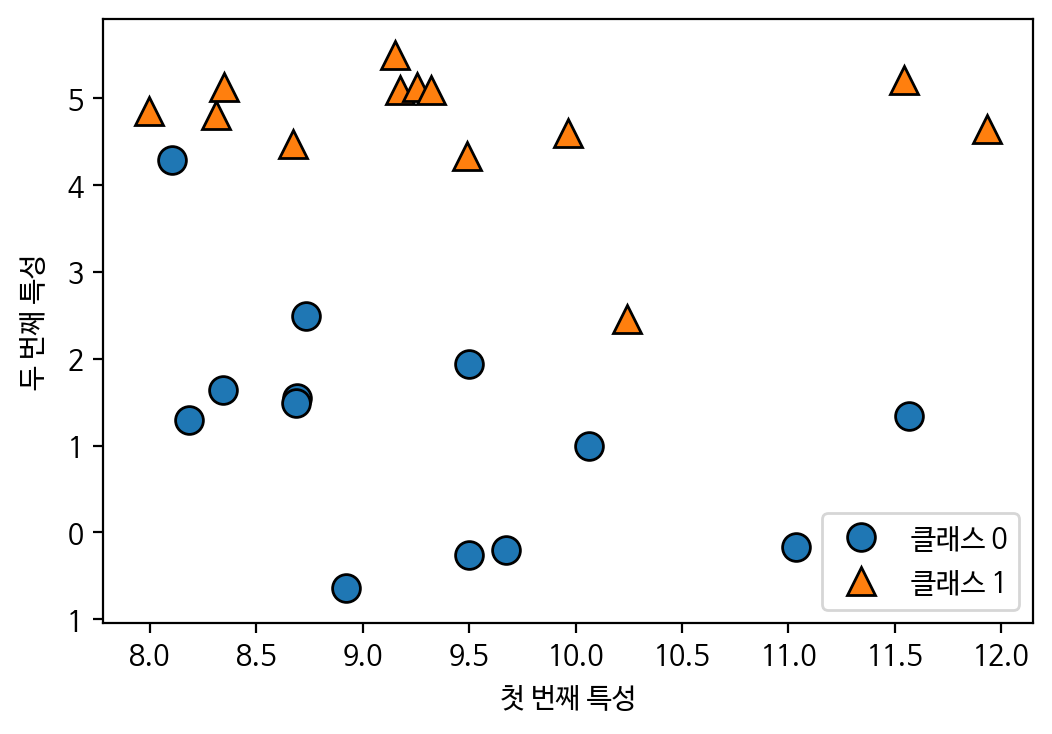
✏️ k-최근접 이웃 알고리즘
# 1-최근접
import mglearn
import matplotlib.pyplot as plt
import warnings
warnings.filterwarnings('ignore')
plt.figure(dpi=100)
mglearn.plots.plot_knn_classification(n_neighbors=1)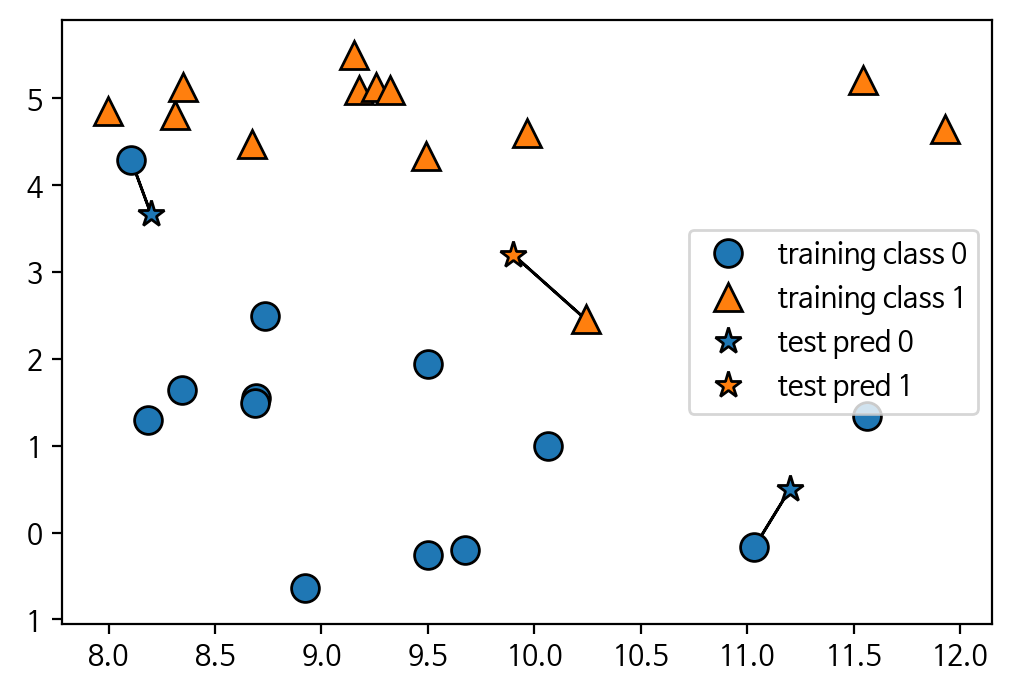
🔼 결과
# 3-최근접
plt.figure(dpi=100)
mglearn.plots.plot_knn_classification(n_neighbors=3)
🔼 결과
: 1일 때와 3일 때의 결과가 다르다. = > 최적점!
✏️ 이진 분류 문제 정의
# 데이터 준비하기
X, y = mglearn.datasets.make_forge() # X : 데이터(feature, 독립변수), y : 레이블(label, 종속변수)
from sklearn.model_selection import train_test_split
# 일반화 성능을 평가할 수 있도록 데이터 분리 -> 훈련셋, 테스트셋
X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=7) # 75:25
X_train.shape # 26 -> 19
X_test.shape # 26 -> 7
# k-최근접 이웃 분류 모델 설정 - 위의 결과 토대로
from sklearn.neighbors import KNeighborsClassifier
clf = KNeighborsClassifier(n_neighbors=3)
# 모델 학습하기
clf.fit(X_train, y_train)
# score 함수를 사용하여 예측 정확도 확인
clf.score(X_test, y_test)
clf.score(X_train, y_train) # -> 과대적합 상황✏️ KNeighborsClassifier 이웃의 수에 따른 성능평가
# 이웃의 수에 따른 정확도를 저장할 리스트 변수
train_scores = []
test_scores = []
n_neighbors_settings = range(1,15)
# 1 ~ 10까지 n_neighbors의 수를 증가시켜서 학습 후 정확도 저장
for n_neighbor in n_neighbors_settings:
# 모델 생성
clf = KNeighborsClassifier(n_neighbors=n_neighbor)
clf.fit(X_train, y_train)
# 훈련 세트 정확도 저장
train_scores.append(clf.score(X_train, y_train))
# 테스트 세트 정확도 저장
test_scores.append(clf.score(X_test, y_test))
# 예측 정확도 비교 그래프 그리기
plt.figure(dpi=100)
plt.plot(n_neighbors_settings, train_scores, label='훈련 정확도')
plt.plot(n_neighbors_settings, test_scores, label='테스트 정확도')
plt.ylabel('정확도')
plt.xlabel('이웃의 수')
plt.legend()
plt.show()
# 최적점은 3!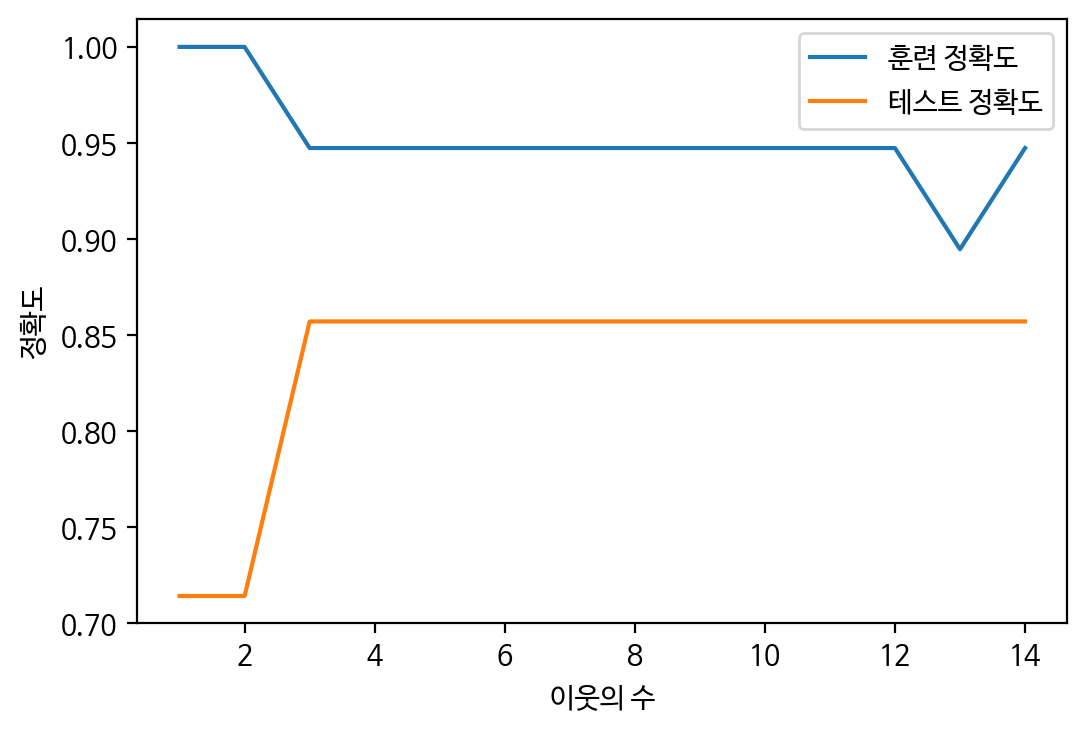
✔️ 위스콘신 유방암 데이터셋을 사용한 악성 종양(label 1) 예측하기
▶️ 독립변수(x, feature, data) : cancer.feature_names 치면 나옴
▶️ 종속변수(y, class, target) : 악성, 양성
# 데이터 불러오기
from sklearn.datasets import load_breast_cancer
cancer = load_breast_cancer()
# 산점도 그리기
import pandas as pd
df = pd.DataFrame(cancer['data'], columns=cancer.feature_names)
pd.plotting.scatter_matrix(df, c=cancer['target'], figsize=(15,15),
marker='o', hist_kwds={'bins':20}, s=10, alpha=.8)
plt.show()# 종속변수의 값 확인
import numpy as np
plt.imshow([np.unique(cancer['target'])])
_ = plt.xticks(ticks=np.unique(cancer['target']), labels=cancer['target_names'])
🔼 결과(0이 악성, 1이 양성)
# 데이터 분리하기(훈련데이터, 테스트데이터)
from sklearn.model_selection import train_test_split
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, random_state=7)
# 최적점 알아보
# 이웃의 수에 따른 정확도를 저장할 리스트 변수
train_scores = []
test_scores = []
n_neighbors_settings = range(1,21)
# 1 ~ 10까지 n_neighbors의 수를 증가시켜서 학습 후 정확도 저장
for n_neighbor in n_neighbors_settings:
# 모델 생성
clf = KNeighborsClassifier(n_neighbors=n_neighbor)
clf.fit(X_train, y_train)
# 훈련 세트 정확도 저장
train_scores.append(clf.score(X_train, y_train))
# 테스트 세트 정확도 저장
test_scores.append(clf.score(X_test, y_test))
# 예측 정확도 비교 그래프 그리기
plt.figure(dpi=100)
plt.plot(n_neighbors_settings, train_scores, label='훈련 정확도')
plt.plot(n_neighbors_settings, test_scores, label='테스트 정확도')
plt.ylabel('정확도')
plt.xlabel('이웃의 수')
plt.legend()
plt.show()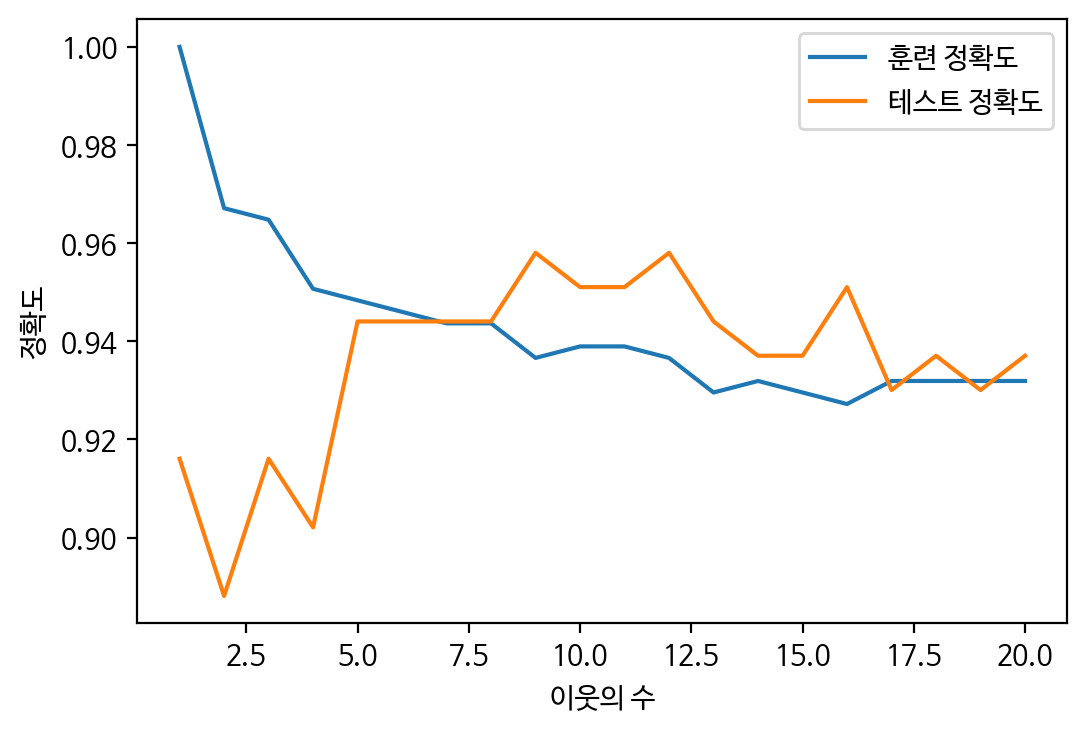
🔼 결과(최적점은 7-8!)