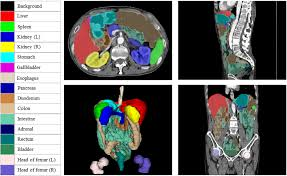
TotalSegmentator
(https://github.com/wasserth/TotalSegmentator)
-
CT image 내에서 각 장기 bounding box_min,max 좌표 (value)와 장기 코드(key) 포함된 nii 파일 반환
-
command에서
roi_subset으로 segmentation 할 장기 설정 -
기타 command setting

from concurrent.futures import ThreadPoolExecutor
import os
import csv
import pandas as pd
from tqdm import tqdm
import concurrent.futures
def process_series(args):
# args에서 각 요소를 분리하여 할당
base_input_folder, base_output_folder, series_tuple, gpu_id = args
# CUDA 환경 설정으로, 특정 GPU를 사용하도록 설정
os.environ['CUDA_VISIBLE_DEVICES'] = str(gpu_id)
# patient_id와 series_id를 series_tuple에서 추출
patient_id, series_id = series_tuple
# 입력 경로와 출력 경로 설정
input_path = os.path.join(base_input_folder, str(patient_id), str(series_id))
output_path = os.path.join(base_output_folder, f"{patient_id}_{series_id}.nii")
# status 초기화
status = None
# 이미 출력 경로에 파일이 존재한다면 'P' (Processed) 상태 할당
if os.path.exists(output_path):
status = 'P'
# 입력 경로에 디렉터리 또는 파일이 존재하지 않으면 'F' (Failed) 상태 할당
elif not os.path.exists(input_path):
status = 'F'
else:
# TotalSegmentator를 이용한 세그먼테이션 명령어 구성
command = f"""TotalSegmentator \
-i {input_path} \
-o {output_path} \
--ml \
-ot 'nifti' \
--roi_subset spleen kidney_left kidney_right liver esophagus colon duodenum small_bowel stomach"""
try:
# 시스템 명령어 실행으로 세그먼테이션 처리
os.system(command + " > /dev/null 2>&1")
# 성공적으로 처리되면 'S' (Success) 상태 할당
status = 'S'
except:
# 예외 발생 시 'E' (Error) 상태 할당
status = 'E'
# segmentation_status.csv 파일을 열어서 처리 상태를 기록
with open("segmentation_status.csv", "a", newline='') as csvfile:
fieldnames = ['patient_id', 'series_id', 'status']
writer = csv.DictWriter(csvfile, fieldnames=fieldnames)
writer.writerow({'patient_id': patient_id, 'series_id': series_id, 'status': status})
def segment_images(base_input_folder, base_output_folder, balanced_csv_path):
# pandas를 이용하여 CSV 파일 로드
balanced_data = pd.read_csv(balanced_csv_path)
# unique한 patient_id를 정렬하여 추출
unique_patient_ids = sorted(balanced_data['patient_id'].unique())
# segmentation_status.csv 파일을 새로 작성하고, 헤더를 설정
with open("segmentation_status.csv", "w", newline='') as csvfile:
fieldnames = ['patient_id', 'series_id', 'status']
writer = csv.DictWriter(csvfile, fieldnames=fieldnames)
writer.writeheader()
# 사용할 GPU의 수
num_gpus = 4
# GPU ID 리스트 생성
gpu_ids = list(range(num_gpus))
# GPU 순환을 위한 카운터
gpu_counter = 0
futures = []
# ThreadPoolExecutor를 사용하여 병렬 처리
with ThreadPoolExecutor(max_workers=num_gpus) as executor:
# 진행 상황을 표시하기 위해 tqdm을 사용
for patient_id in tqdm(unique_patient_ids):
# 해당 patient_id의 모든 series 정보를 가져옴
patient_series_info = balanced_data[balanced_data['patient_id'] == patient_id][['patient_id', 'series_id']].values
for series_tuple in patient_series_info:
# 각 작업에 대한 인수 구성
args = (base_input_folder, base_output_folder, series_tuple, gpu_ids[gpu_counter % num_gpus])
# 작업을 futures에 추가
futures.append(executor.submit(process_series, args))
# 다음 GPU ID 할당
gpu_counter += 1
# 모든 futures 작업이 완료될 때까지 대기하며 진행 상태 표시
for future in tqdm(concurrent.futures.as_completed(futures), total=len(futures)):
pass
# 주석 해제 후 올바른 경로를 설정하여 사용
# segment_images('base_input_folder', 'base_output_folder', 'balanced_csv_path')
-
os.system으로 파이썬 외부 명령어(total segmentator) 실행 -
ThreadPoolExecutor: 멀티 GPU 환경에서 병렬 처리 -> GPU 순환 할당해 균등하게 사용되도록 함 -> 효율적 리소스 활용 가능