
Concept
두 Cromosome에서 유전자들을 비교하는 가장 간단한 방법은 두 Chromosome에서 동일한 순에서 발견되는 가장큰 유전자집합을 찾아내는 것입니다. 그렇게 하기 위해 순열을 이용하는 것인데, 예시로 두 Chromosome이 n개의 유전자를 공유한다고 했을 때. 만약 하나의 크로모좀에 있는 유전자들을 순서대로 1번부터 n번까지 번호를 매긴다고 하면, 다른 크로모좀에는 1 ~ n 개의 공유된 유전자가 가능한 순열 중 하나로 나열되어 있을 것입니다. 가장 큰 개수의 숫자의 동일한 순서로 나열된 유전자를 찾기위해서는 순열에서 번호 순서대로 나열되어 있는 가장 큰 집합을 찾기만 하면 된다는 개념입니다. 간단한 개념 같아 보이지만 Longest Increasing Subsequence 는 전혀 쉽지 않은 개념이었습니다. 제가 이전에 올렸던 5 Principle of Dynamic Programming을 참조하여 풀었음을 알려드립니다.
Problem
여기 무차별로 섞인 수열이 있습니다. (5, 1, 4, 2, 3) 이 수열에서 일부분의 수열 집합을 뽑아내면 (5, 1, 4), (1, 2, 3), ... 등 개로 5에서 n개를 나열할 수 있습니다. 이 부분 집합을 subsequence 부분수열이라 합니다.
만일 이 부분수열의 요소들이 증가한다면 부분수열이 증가하고 있다하고, 반대로 줄어들고 있다면 부분수열이 줄어들고 있다고 말합니다. 예를들자면 위의 (5, 1, 4, 2, 3)에서 (1, 2, 3)은 요소가 증가하고 있으니 증가한다 말할 수 있을 것이고, (5, 4, 2) 또는 (5, 4, 3)은 줄어들고 있다고 말할 수 있을 것입니다. 그리고 수열이 길어져서, 문제에 나와 있는 (8, 2, 1, 6, 5, 7, 4, 3, 9) 수열의 부분수열 중 증가 수열은 (1, 2, 7, 9)가, 감소 부분수열은 (8, 6, 5, 4, 3) 등이 있을 것입니다. 우리 가 구할 것은 전체 수열중 가장 긴 증가부분수열과, 감소부분수열을 모두 구하는 것입니다.
Given : 10000개 이하의 수열이 주어집니다.
Return : 가장 긴 증가 부분수열과, 가장 긴 감소 부분수열을 구하는 것입니다.
예를 들어 (5, 1, 4, 2, 3) 이 수열로 주어진다면 반환값으로
1 2 3
5 4 2
가 출력이 되어야 한다는 것이죠.
Solution
이 문제는 dynamic programming 이라는 방식으로 풀수 있다고 합니다.(허접이라 dynamic programming이 뭔지 아직 감을 못잡았습니다.) 이 문제의 경우 Longest increasing problem에서 단순히 가장 긴 길이만 얻는 문제로 5 Principle of Dynamic Programming에서 했던 사고 방식을 적용하여 풀어보자면
1. Visualize Example
먼저 개념을 파악하기 위해 이미지를 그려봅니다. 그림을 통해 알 수 있는 것은 모든 수열은 노드로 표현이 될 수있고, 노드끼리의 Edge로 표현 될 수 있는 그래프라는 것입니다.
2. Find a Appropriate Subproblem, Find Relationship among Subproblems
일단 가장 작은 문제를 파악해야 합니다. 그러기 위해서는 가장 작은 규칙을 찾아야 하는데 이게 두번째로 어려운 것 같습니다. Longest Increasing Subsequence를 LIS라 하고, 어떤 index i 까지의 부분수열에서 가장 긴 수열의 길이를 LIS(i)라 했을때, 아래와 같은 규칙을 찾을 수 있습니다.
(8,2,1,6,5,7,4,3,9)
LIS(0) = 1 -> 8하나 밖에 없음으로 1개
LIS(1) = 1 -> 8 다음 2임으로 늘어나지 않아 1개
LIS(2) = 1 -> 위와동일한 이유로 1개
LIS(3) = 2 -> (2, 6), (1, 6) 이 존재함으로 최대 길이는 2
LIS(4) = 2 -> (2, 5), (1, 5) 이 존재함으로 최대 길이는 2
LIS(5) = 3 -> (1, 6, 7), (2, 6, 7)이 존재함으로 최대 길이는 3
LIS(6) = 2 -> (1, 4), (2, 4) 가 존재함으로 최대 길이는 2
LIS(7) = 2 -> (1, 3), (2, 3) 가 존재함으로 최대 길이는 2
LIS(8) = 4 -> (1, 6, 7, 9), (2, 6, 7, 9)가 존재함으로 최대 길이는 4가 됩니다.
위의 리스트를 자세히 살펴보면
LIS(0)는 수열 8이 가질 수 있는 가장 긴 수열인데, 8 하나밖에 없습니다. 그러므로 길이는 1이 됩니다. 그 다음 LIS(1)에서는 2값인데 아래에 자기보다 작은 수가 하나도 없습니다. 그러므로 자기 자신을 길이에 포함한 1이되는 것입니다. LIS(4)에서는 자기 자신이 6일때, 아래에 있는 수 중 6보자 작은 값은 2, 1이고, LIS(1), LIS(2)은 이미 알고 있습니다. 그러므로 둘중에 최대값인 1인 값에, 6 자기자신을 포함시키면 2가 되는 것을 알 수 있습니다.
3. Generalize the Relationship
위의 규칙을 일반화 하면 이라는 공식으로 정리됩니다. 이제 여기서 중요한 것은 우리가 원하는 것은 증가 부분수열과 감소 부분수열을 모두 구하는 것입니다. 그렇기에 약간의 트릭이 필요한데요, 바로 에서 max를 구할 때 사용된 k값을 적절하게 구하는 것입니다.
4. Implement by Solving Subproblems in Order
Reducible에서 제시한 구현법은 아래와 같습니다.
def LIS(A):
L = [1] * len(A)
for i in range(1, len(L)):
subproblems = [L[k] for k in range(i) if A[k] < A[i]]
L[i] = 1 + max(subproblems, default=0)
return max(L, default=0)하지만 우리가 구하려는 것은 증가 및 감소한 가장 긴 수열을 직접 구하는 것임으로 여러가지 장치가 더 필요합니다.
4-1 LS
우리에게 필요한 것은 증가, 감소에 모두 적용 가능한 함수인 Longest Sequence의 약자인 LS 함수를 만들어야 합니다. 이 함수는 증가하는 값의 인덱스 추적값과 원본 수열을 반환합니다. 증가는 어떻게든 만들 수 있을텐데 어떻하면 감소까지 만들 수 있을까요? 그건 간단합니다. 수열을 뒤집으면 감소가 증가가 됩니다.
(8, 2, 1, 6, 5, 7, 4, 3, 9)에서 감소값은 (8, 6, 5, 4, 3)입니다. 이를 수열을 뒤집어 증가값으로 구하면 (9, 3, 4, 7, 5, 6, 1, 2, 8) 의 가장 긴 증가값은 (3, 4, 5, 6, 8)이 되는 것을 알 수 있습니다.
그러므로
def LS(long_type, seq):
if long_type != "inc": seq = seq[::-1] # 순열을 뒤집으면 감소문제가 증가 문제로 바뀝니다.그 다음으로 필요한 것은 max 값에서 사용된 k값을 추적하는 것이죠. 여기서 문제가 하나가 있는데, 하나의 값에 연결된 노드들이 많을 수 있다는 것입니다. 예를 들자면 (8, 2, 1, 6, 5, 7, 4, 3, 9) 수열에서 6의 경우에는 8를 제외한 이전 값들과 연결이 되어 있습니다. 하지만 실제로 연산시에는 구별이 가지 않습니다. LIS(0), LIS(1), LIS(2)가 모두 값이 1입니다. 그러다면 LIS(3)을 구할 때 1 + max(LIS(0), LIS(1), LIS(2)) 일텐데 이 중에 어떤 값을 썼는지 어떻게 알 수 있을까요? LIS 값이 모두 1이라면, LIS(0~2)는 모두 감소하는 값이 됩니다. 만일 중간에 증가하는 수열이 있었다면 LIS 값은 1이 나오지 않았겠죠. 그러므로 가장 마지막 index인 2를 추적값으로 사용 할 수 있게 됩니다.
@tz.curry
def LS(long_type, seq):
if long_type != "inc":
seq = list(reversed(seq))
L = [1] * len(seq)
prev_idxes = []
for i in range(0, len(L)):
subproblems = [L[k] for k in range(i) if seq[k] < seq[i]]
L[i] = 1 + max(subproblems, default = 0)
if len(subproblems) == 0: prev_idxes.append(-1)
else:
last_idx = len(L[:i]) - L[:i][::-1].index(max(subproblems)) -1 # max에서 사용된 subproblem 값중 가장 큰값의 마지막 인덱스를 구하는 로직
prev_idxes.append(last_idx)
return prev_idxes, seqpython을 아직 잘 모르시는 분은 len(L[:i]) - L[:i][::-1].index(max(subproblems)) -1 파트가 잘 이해가 안가실지도 모르겠습니다. 그냥 L.index(max(subproblems)) 를 하게 되면 가장 앞에서 찾는 값의 index를 찾아 냅니다. 하지만 이 문제에서 요구하는 것은 가장 뒤의 값을 찾는 것이죠. 예를 들어 [1, 2, 2, 3, 3] 이 있다고 했을때, 가장 큰 값의 index를 찾고싶다고 하죠. 그러면 list.index(3)을 하게 되면 3이 나오지만, 우리가 원하는건 맨 뒤에서 큰 값이기 때문에 배열을 뒤집습니다. [3, 3, 2, 2, 1]이 되고, 뒤집힌 배열의 가장 큰 값의 index는 0이 되고 이를 배열 길이 5 에서 빼고, -1을 해주면 4가되어 우리가 원하는 값이 나오게 됩니다.
이렇게 메인 로직이 끝나면 추적한 인덱스들을 값으로 변환해주어야 합니다.
def decode_prev_idx(data_package):
prev_idxes, seq = data_package
vals = []
for cur_idx in range(len(prev_idxes) -1, -1, -1):
val = []
while cur_idx != -1:
cur_val = seq[cur_idx]
val.append(cur_val)
prev_idx = prev_idxes[cur_idx]
cur_idx = prev_idx
vals.append(val[::-1])
return vals위의 로직은 아래와 같은 flow chart로 표현 될 수 있습니다.
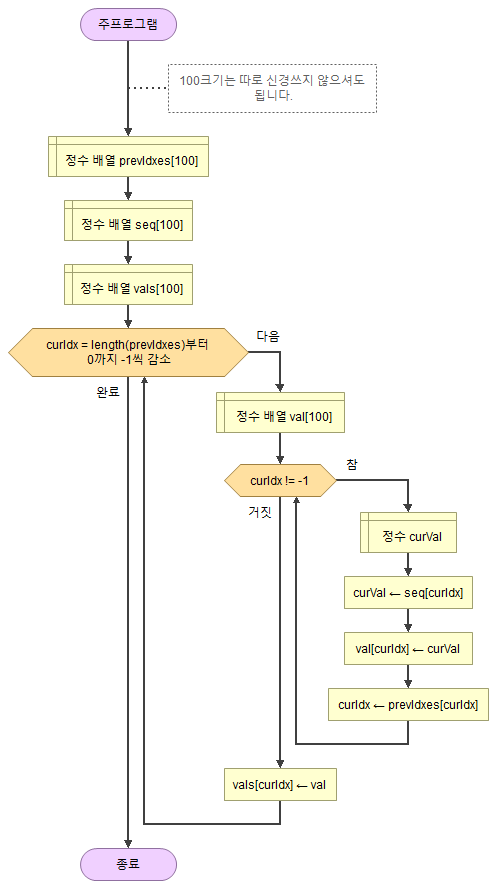
이 방식으로 데이터는 디코딩 되고, 이중에서 가장 긴 값을 뽑아 반환하면 되며, 출력시에 감소일 경우 반대로 뒤집어 주기만 하면 됩니다.
전체 코드
여기 저기 부딪치며 간신히 풀었네요. Rosalind를 풀 누군가에게 도움이 되었으면 좋겠습니다.
import toolz as tz
from toolz.curried import *
import numpy as np
test_seq = "8 2 1 6 5 7 4 3 9"
input_processor = tz.compose(list, map(int), tz.flip(str.split, " "))
@tz.curry
def LS(long_type, seq):
if long_type != "inc":
seq = list(reversed(seq))
L = [1] * len(seq)
prev_idxes = []
for i in range(0, len(L)):
subproblems = [L[k] for k in range(i) if seq[k] < seq[i]]
L[i] = 1 + max(subproblems, default = 0)
if len(subproblems) == 0: prev_idxes.append(-1)
else:
last_idx = len(L[:i]) - L[:i][::-1].index(max(subproblems)) -1
prev_idxes.append(last_idx)
return prev_idxes, seq
def decode_prev_idx(data_package):
prev_idxes, seq = data_package
vals = []
for cur_idx in range(len(prev_idxes) -1, -1, -1):
val = []
while cur_idx != -1:
cur_val = seq[cur_idx]
val.append(cur_val)
prev_idx = prev_idxes[cur_idx]
# print(f"prev_idx : {prev_idx} | cur_idx : {cur_idx} | cur_val : {seq[cur_idx]}")
cur_idx = prev_idx
vals.append(val)
return vals
@tz.curry
def print_result(long_type, seq):
if long_type == "inc": print(*seq[::-1])
else: print(*seq)
for long_type in ["inc", "dec"]:
tz.pipe(test_seq, LS(long_type), decode_prev_idx, topk(1, key=len), get(0), print_result(long_type))
굳이 toolz를 쓰는 이유가 있나요?