[Computer Vision] MRI Image data Segmentation
Computer Vision
- Image Segmentation Project
#############################################################
Image Segmentation Models
[ 0. Briefing ]
a) Project :: Liver Segmentation on MRI image
프로젝트의 1차적 목표는 MRI image를 바탕으로 Liver Region을 Segmentation 하는 딥러닝 모델을 만들어 하드웨어 팀의 데이터를 확보해주는 것이다. 그 동안은 오직 의료진의 레이블링된 데이터에만 의존했지만, 의료진에 따라 레이블링의 넘버와 범위가 모두 달라 전처리에 어려움을 겪고 있었다. 이를 위해 AI Segmentation 기술을 교수님께 건의드렸고, 해당 프로젝트를 부여받았다. 우선적으로 Liver Region 에 대한 Segmentation 을 진행하고 나아가 다른 Abdominal 장기들에 대한 Segmentation 이미지 데이터를 제공할 계획이다.
b) Data set :: Abdominal Region MRI raw image data (.dicom, .png)
{Fig.01 raw image data}
위와 같은 이미지들에서 Liver reigon을 Segmentation 하는 것이 목적이다.
[ 1. Referenced model : Unet(2015) ]
Olaf Ronneberger, Philipp Fischer, and Thomas Brox(2015), 'U-net: Convolution Networks from Biomedical Image Segmentation'
a) abstract
논문을 참고하여 Unet model 을 프로젝트의 다음 단계로 설정하였다. 논문의 Abstract에 나와있듯이, 의료 데이터를 학습시킬때는 쓸 수 있는 이미지의 수가 한정되어 있기 때문에 적은 데이터로도 좋은 결과를 내는 해당 모델이 유리했다. 또한, Unet 이전에 사용되었던 Sliding-window convolutional network 에 비해 수행 속도가 더 빠르다. 위와 같은 이유로 Unet model 을 이번 Segmentation 리서치에 목표 모델로 설정하게 되었다.
b) Introduction
Sliding-window conv. net은 입력 데이터에서 제공한 pixel 별로 구분된 국소 지역에 대한 정보(이하 patches)을 활용하여 pixel의 Class를 설정한다. 하지만 이러한 방법은 학습 데이터보다 더 많은 patches를 필요로 하며, patches 별로 중복이 다수 존재한다. 또한, 국소 지역 Accuracy와 이미지 Context 사이에 trade-off 가 존재하여 이를 개선하기 위한 추가적 Layer 들이 필요하다. 이로 인해 데이터 학습에 더 많은 시간들이 걸리게 되는 것이다.
이에 비해, 해당 논문에서 제안한 모델은 훨씬 적은 학습 이미지로 보다 정확한 Segmentation이 가능한 것으로 설명되었다. 이러한 이점은 직접 Labeling 을 진행해야하는 입장에서 Unet 모델을 선택하는게 합리적이라 생각이 들었다. Unet 모델이 더 적은 이미지로 높은 precision을 얻을 수 있는 이유는 DownSampling 과정에서 이전 층의 고해상도 이미지를 학습 이후 발생한 Feature map에 보완을 해주기 때문이다. 일종의 Fully Connected Network 의 개념을 활용한 것이라 설명되어 있다. 이와 반대로 UpSampling 과정은 DownSampling의 반대로 설정하여 전체적은 모델의 구조를 U 자 형으로 설정하게 되었다. 이 역시 맥락 정보를 각 층마다 주기 위한 구조이다. Unet 모델에서 항상 매끄러운 경계면을 갖게 만들어주기 위해 타일링 전략을 사용하였다. 경계면을 예측함에 있어 이전의 입력 이미지를 활용하여 추론하는 방식으로 이를 사용하지 않을 경우 GPU 메모리 한계를 넘을 수가 있다. 해당 논문에서는 Data Augmentation 을 강조하고 있다. 하지만 이번 프로젝트에서 수행할 작업은 이미지의 모습이 일정하게 입력될, 즉 Axis 방향의 연속적인 이미지를 앞뒤 양옆의 방향을 맞추어 제공할 것이기 때문에 따로 수행하지는 않을 것이다.
c) Network Architecture & Training
DownSampling 에서는 3x3 Conv.를 2번 진행하고 이때 Relu 함수를 사용하였다. 2x2 Maxpooling은 strdie를 2로 설정하였으며 padding은 주지 않았다. 하지만 프로젝트에서 만들 모델에 대해서는 패딩을 사용하여 후에 만들어질 이미지의 크기를 유지할 생각이다. UpSampling은 이와 반대로 진행된다. 역시나 3x3 Conv.를 2번 진행하고 Feature map 의 수를 반으로 줄여가며 이미지를 키운다. 마지막에 나오는 이미지에서는 1x1 conv.가 진행되며 64개의 분류해야할 Feature vector 들을 출력한다. 프로젝트의 경우, 512x512x2 의 차원으로 하나는 Liver region인지 다른 하나는 그 외의 region 인지를 나타내는 모습으로 mask가 생성될 것이다. 마지막 Feature map 에서는 pixel-wise soft-max 함수를 사용하여 확률의 모습으로 이미지를 출력시킬 예정이다.
[ 2. Prototype Unet ]
0. Environment :
Google Colab Pro | TensorFlow | Keras
a) dicom to Png
전체적인 과정을 진행하기 전에, 아래와 같은 방법으로 MRI 이미지의 ".dicom" 형식을 "png" 형식으로 전환할 수 있다.
import numpy as np
import pydicom
import cv2
import os
img = np.array(img, dtype = float)
img = (img - img.min()) / (img.max() - img.min()) * 255.0
img = img.astype(np.uint8)
cv2.imwrite(outdir + f.replace('.dcm','.png'),img)b) Labeling the answer
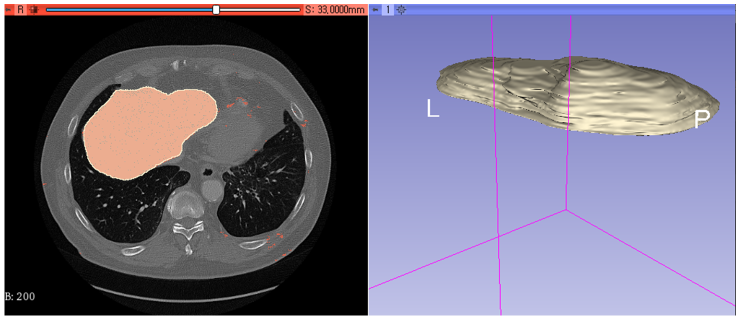
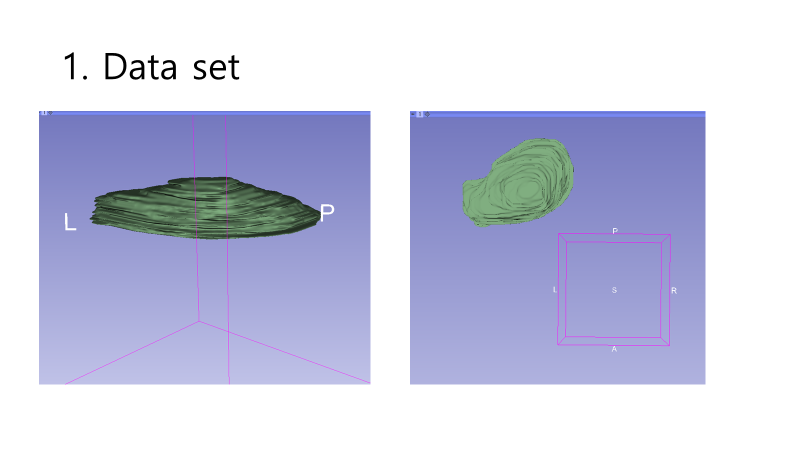
Unet model 은 Ground Truth를 바탕으로 하는 Supervised model 이다. 해당 프로젝트에서 받은 자료는 환자의 Abdominal MRI raw image 이기 때문에 Ground Truth를 직접 만들어야했다. 해당 작업은 3D slicer에서 진행하였으며, Segmentation tool 을 통해 마우스를 사용하여 그렸다. 총 5명에 환자들에 대해, 한 환자당 대락 50~60장씩 Axial 방향으로 Liver Region을 특정하였다. 이와같이 만들어진 Ground Truth 파일들은 .nii 형식으로 저장이 된다. 이하는 직접 그린 GT image의 예시이다.
 {Fig01. Labeled mask with raw image and without raw image}
{Fig01. Labeled mask with raw image and without raw image}
 {Fig.02 Labeled mask with raw images}
{Fig.02 Labeled mask with raw images}
-
1. Data Set :
a) X_train data : Png to Array_set
## FUNCTION :: Convert png 2 ndarray
def png2set(path):
file_list = os.listdir(path)
X_img = []
X_name = []
# >> "Extract name from file"
for name in file_list:
if name.find('.png') is not -1:
X_name.append(name)
X_name = sorted(X_name)
# >> "Build Train Set"
for i in range(len(X_name)):
img_tempo = cv2.imread(path + X_name[i], cv2.IMREAD_GRAYSCALE)
img = cv2.equalizeHist(img_tempo)
nimg = img / 255
X_img.append(nimg)
# >> "Reshape Train Set Dimension"
X_train = np.concatenate(X_img, axis=0)
#X_train = X_train.reshape(-1, 512, 512)
X_train = X_train.reshape(-1, 512, 512, 1)
return X_train구글 드라이브에 저장되어 있는 MRI png 파일을 계산가능한 numpy.ndarray 로 바꾸는 과정이다. 이미지의 색 분포가 고르지 않을 수 있기 때문에 모델 성능 향상을 위해 히스토그램 평활화( cv2.equalizeHist )를 진행하였다. 연속적인 이미지이기 때문에 Concatenate 를 사용하여 이미지들을 Axial 방향으로 연결시켰다. 마지막으로 이미지 사이즈를 512 x 512 로 맞추고 Dimension 을 통일해주었다.
b) Y_train data : nif to Array_set
## FUNCTION :: Convert nif 2 ndarray
def nif2set(path):
nif = nib.load(path)
nif_arr = nif.get_fdata()
y_train = np.transpose(nif_arr, (2,1,0))
y_train = y_train.reshape(-1, 512, 512, 1)
y_train = y_train.astype('bool')
return y_train3D Slicer에서 라벨링을 마친 데이터들은 nif 형식으로 저장되어있다. 이 역시 계산 가능한 numpy.ndarray 로 데이터를 변환시켜주었다. nif에서 import 할 때, 기본 방향이 Sagittal 방향이였기 때문에 이를 Axis 방향으로 설정해주었다. 오직 Liver에 대한 이미지 분석이기 때문에 y_train의 type을 bool로 변경하였다.
C) Checking the train_set
X_train1 = png2set('/content/drive/My Drive/1/')
y_train1 = nif2set('/content/drive/My Drive/1.nii')
X_train2 = png2set('/content/drive/My Drive/2/')
y_train2 = nif2set('/content/drive/My Drive/2.nii')
X_train3 = png2set('/content/drive/My Drive/3/')
y_train3 = nif2set('/content/drive/My Drive/3.nii')
만들어진 함수들을 사용하여 train_set 을 준비 완료하였다. 이하는 만들어진 train_set을 확인한 결과이다.
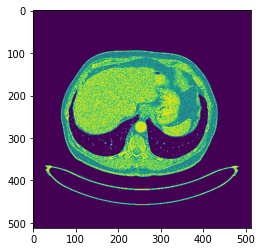
plt.imshow(X_train1[40].reshape(512, 512))
plt.imshow(y_train1[40].reshape(512, 512))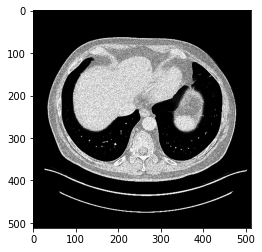
plt.imshow(X_train3[35].reshape(512, 512), cmap='gray')
plt.imshow(y_train3[35].reshape(512, 512), cmap='gray')-
2. Build the Unet model :
a) Unet model
## Model :: Unet
from keras.models import Model
import tensorflow as tf
#==========================================================================
Channel_in = Input(shape=(512, 512, 1))
conv1 = Conv2D(32, (3, 3), activation='relu', padding='same')(Channel_in)
conv1 = Dropout(0.2)(conv1)
conv1 = Conv2D(32, (3, 3), activation='relu', padding='same')(conv1)
pool1 = MaxPooling2D((2, 2))(conv1)
conv2 = Conv2D(64, (3, 3), activation='relu', padding='same')(pool1)
conv2 = Dropout(0.2)(conv2)
conv2 = Conv2D(64, (3, 3), activation='relu', padding='same')(conv2)
pool2 = MaxPooling2D((2, 2))(conv2)
#==========================================================================
conv3 = Conv2D(128, (3, 3), activation='relu', padding='same')(pool2)
conv3 = Dropout(0.2)(conv3)
conv3 = Conv2D(128, (3, 3), activation='relu', padding='same')(conv3)
up1 = concatenate([UpSampling2D((2, 2))(conv3), conv2], axis=-1)
conv4 = Conv2D(64, (3, 3), activation='relu', padding='same')(up1)
conv4 = Dropout(0.2)(conv4)
conv4 = Conv2D(64, (3, 3), activation='relu', padding='same')(conv4)
up2 = concatenate([UpSampling2D((2, 2))(conv4), conv1], axis=-1)
conv5 = Conv2D(32, (3, 3), activation='relu', padding='same')(up2)
conv5 = Dropout(0.2)(conv5)
conv5 = Conv2D(32, (3, 3), activation='sigmoid', padding='same')(conv5)
#==========================================================================
Channel_out = Conv2D(2, (1, 1) , padding='same')(conv5)
model = Model(Channel_in, Channel_out)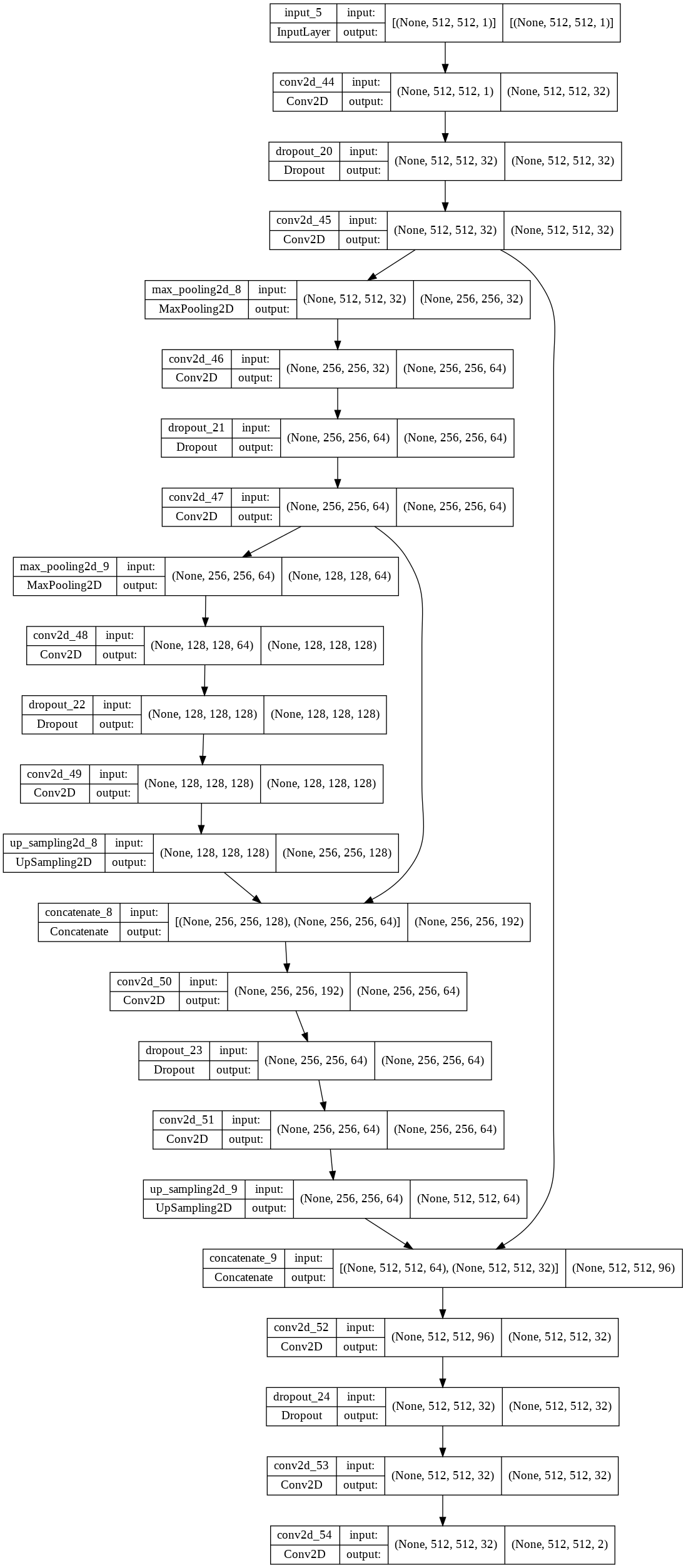
model.summary
tf.keras.utils.plot_model(model, show_shapes=True)프로토타입인 만큼, 데이터의 크기 (50 x 512 x 512)와 환경적인 요인 (Colab TPU)을 고려하여 짧은 시간 안에 간단한 결과물이 나올 수 있도록 설계하였다. MaxPooling 과 UpSampling 을 2번씩 진행하였으며, 각 차원에 맞는 Pooling 전 데이터를 UpSampling 이후에 Concatenate 하였다.
b) Compile
adam = keras.optimizers.Adam(lr=0.001)
model.compile(optimizer=adam,
loss='binary_crossentropy',
metrics=['accuracy'])Optimizers는 Adam으로 Learning rate=0.001로 설정하였다. 이미 One_Hot encoding이 완료되었기 때문에, loss="binary_crossentropy"를 사용하였다.
-
3. Learning & Result :
batch_size = 10
epochs = 40
history1 = model.fit(X_train1, y_train1,
batch_size=batch_size,
epochs=epochs,
validation_split=0.1)Colab GPU로 환자별 즉, data_set 별로 학습을 걸어두었다. 층이 매우 얕기 때문에 한 명의 MRI image data set (50x512x512x1) | epochs=40 에 대해 10분 안에 결과가 나온다.
from sklearn.preprocessing import MinMaxScaler
img_test = cv2.imread(tester_name, cv2.IMREAD_GRAYSCALE)
img = cv2.equalizeHist(img_test)
nimg = img /255
nimg_resized = nimg.reshape(-1, 512, 512, 1)
result = model.predict(nimg_resized)
result_r = result.reshape(512, 512, 2)
result_new = np.transpose(result_r, (2,0,1))
scaler = MinMaxScaler()
normalres1 = scaler.fit_transform(result_new[0])
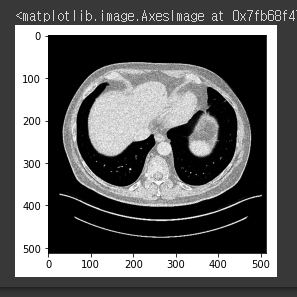
normalres2 = scaler.fit_transform(result_new[1])나온 결과물 마스크에 대해 정규화( MinMaxScaler )를 진행하였다. 임시 결과물을 보기 위해 하나의 이미지에 대해 model prediction 을 시도해보았다. test image는 1번 환자의 240.png 이다. 해당 이미지는 아래와 같다.
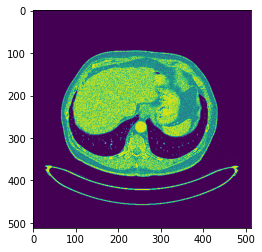
{Fig.03 Test image}
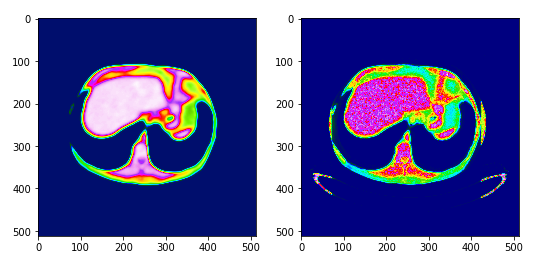 {Fig.04 Mask and Result image in epoch=40}
{Fig.04 Mask and Result image in epoch=40}
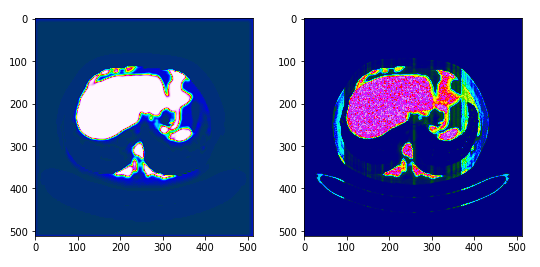 {Fig.04 Mask and Result image in epoch=80}
{Fig.04 Mask and Result image in epoch=80}
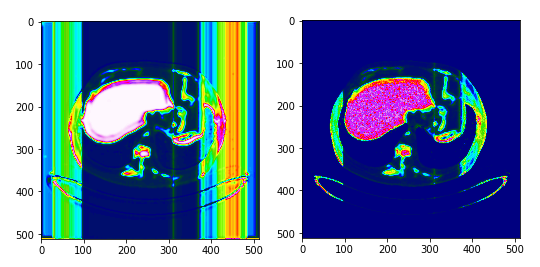 {Fig.04 Mask and Result image in epoch=120}
{Fig.04 Mask and Result image in epoch=120}
목표를 판단하는데 있어서 색깔도 중요한 판단요소이기 때문에 주변에 비슷한 색을 갖는 목표 이외의 요소들이 같이 출력되었다. 위와같은 문제점은 Unet의 Layer를 더 깊게 쌓아 충분히 해결할 수 있을것이라 판단된다.
[ 3. Image Segmentation model ]
0. Environment :
Google Colab Pro | TensorFlow | Keras
-
1. PreProcessing :
a) png2set :
## FUNCTION :: Convert png 2 array_set
def png2set(Input_dimension, path):
Img_width, Img_height = Input_dimension
file_list = os.listdir(path)
X_img = []
X_name = []
# Extract name from file
for name in file_list:
if name.find('.png') is not -1:
X_name.append(name)
X_name = sorted(X_name)
# Build Train Set
for i in range(len(X_name)):
img_tempo = cv2.imread(path + X_name[i], cv2.IMREAD_GRAYSCALE)
img = cv2.equalizeHist(img_tempo)
nimg = img / 255
X_img.append(nimg)
# Reshape Train Set Dimension
X_train = np.concatenate(X_img, axis=0)
X_train = X_train.reshape(-1, Img_width, Img_height, 1)
return X_trainpng2set 함수는 X_train 데이터를 처리하는 함수이다. .png 파일로 저장된 MRI raw image 를 계산가능한 ndarray 형식으로 변환한다. 이미지들의 픽셀값은 equalizeHist 를 통해 히스토그램을 평활화 시킨다. 개별적인 이미지가 아닌 하나의 연속성을 갖는 이미지이기 때문에, Axis 방향의 차원을 가장 앞에 명시하였다. 따라서 하나의 Image data set은 50x512x512x1 의 형태를 갖는다. 마지막의 x1 은 이미지가 하나 들어갔다는 의미로 모델 내에서 여러 layer들은 여러장을 하나의 차원에 갖기 때문에 raw image 데이터를 전처리할 때 미리 저장해주었다.
b) nif2set :
## FUNCTION :: Convert nif 2 array_set
def nif2set(Input_dimension, path):
Img_width, Img_height = Input_dimension
nif = nib.load(path)
nif_arr = nif.get_fdata()
y_train = np.transpose(nif_arr, (2,1,0))
y_train = y_train.reshape(-1, Img_width, Img_height, 1)
y_train = y_train.astype('bool')
return y_trainnig2set 함수는 y_train 데이터를 처리하는 함수이다. 3D slicer 에서 직접 labeling 한 데이터들은 .nif 의 형식으로 되어있다. 이 역시 TF 에서의 계산을 용이하게 하기 위해 ndarray 의 형태로 모양을 바꾸어주었다. 역시나 MRI 데이터인 만큼 각 픽셀의 데이터를 히스토그램 평활화 시켜주었다. 각 Ground Truth 데이터는 그에 해당하는 X_train 데이터에 1대1 대응이 가능해야하기 때문에 같은 방법으로 Axis 차원을 y_train 역시 50x512x512x1 로 만들어주었다.
-
2. UnetModel :
a) InputLayer :
def InputLayer(layerin, Filters=0):
layerout = Conv2D(Filters, (3, 3), strides=2, padding='same')(layerin)
layerout = BatchNormalization()(layerout)
layerout = Activation('relu')(layerout)
return layeroutInput 으로 받은 1x512x512x1 의 이미지에 대해 3x3 Conv2D 를 실행하였다. Conv2D를 512x512 의 크기에 대해 진행하기 때문에 strides=2로 주었다. 데이터 특성상 이미지의 가장자리에는 의미없는 데이터만 존재하기 때문에 Padding은 데이터의 차원이 줄어들지 않는 선에서만 실시하였다.
b) DownSampling :
def DownSampling(layerin, Filters=0):
# The number of Filters = Filters
layerout = Activation('relu')(layerin)
layerout = SeparableConv2D(Filters, (3, 3), padding='same')(layerout)
layerout = BatchNormalization()(layerout)
layerout = Activation('relu')(layerout)
layerout = SeparableConv2D(Filters, (3, 3), padding='same')(layerout)
layerout = BatchNormalization()(layerout)
layerout = MaxPooling2D((3, 3), strides=2, padding='same')(layerout)
residual = Conv2D(Filters, (1, 1), strides=2, padding='same')(layerin)
layerout = add([layerout, residual])
return layeroutUnet 에서 이미지의 크기를 줄여가며 특징들을 추출하는 블록이다. Activation='relu' 이며 각 부분에 대해 3x3 SeparableConv2D를 적용하였다. 각 단계마다 BatchNormalizeation()을 사용하여 가중치들의 분포들을 바로잡아 깊어지는 Layer 층을 미리 대비하였다. 해당 단계를 2번 거쳐 MaxPooling2D로 가장 가치있는 정보를 추려내었다. 이와 동시에 블록의 Input으로 들어온 Layer를 Conv2D를 통해 가공하고 이 결과를 MaxPooling을 통해 나온 가중치들을 보완하는데 사용하였다.
c) UpSampling :
def UpSampling(layerin, Filters=0):
# The number of Filters = Filters
layerout = Activation('relu')(layerin)
layerout = Conv2DTranspose(Filters, (3, 3), padding='same')(layerout)
layerout = BatchNormalization()(layerout)
layerout = Activation('relu')(layerout)
layerout = Conv2DTranspose(Filters, (3, 3), padding='same')(layerout)
layerout = BatchNormalization()(layerout)
layerout = UpSampling2D(2)(layerout)
residual = UpSampling2D(2)(layerin)
residual = Conv2D(Filters, (1, 1), padding='same')(residual)
layerout = add([layerout, residual])
return layeroutDownSampling에서 가공한 정보들을 다시 Input 이미지와 비슷한 형식으로 만들어가는 과정이다. SeparableConv2D 대신 Conv2DTranspose를 통해 차원의 크기를 다시 늘려나간다. 앞선 두 결과를 통해 나온 가중치들과 Input 으로 들어온 데이터를 Conv2D로 가공한 가중치들을 합산해주어 데이터를 가공시켜 return 시켜준다.
d) build :
## Building Unet model
def Build(Input_dimension):
# Input Layer
Img_width, Img_height = Input_dimension
Channel_in = Input(shape=(Img_width, Img_height, 1))
layer1 = InputLayer(Channel_in, Filters=32)
# DownSampling
layer2 = DownSampling(layer1, Filters=64)
layer3 = DownSampling(layer2, Filters=128)
layer4 = DownSampling(layer3, Filters=256)
# UpSampling
layer5 = UpSampling(layer4, Filters=256)
layer6 = UpSampling(layer5, Filters=128)
layer7 = UpSampling(layer6, Filters=64)
layer8 = UpSampling(layer7, Filters=32)
# Output Layer
Channel_out = Conv2D(2, (3, 3), activation='softmax', padding='same')(layer8)
return Model(Channel_in, Channel_out)앞서 만들어진 블록들을 활용하여 본격적으로 Unet을 설계하는 단계이다. Channel_in 을 사용하여 Input_image의 차원을 받아 InputLayer로 만들어주었다. 나아가 DownSampling을 3번, 각 필터의 크기를 64, 128, 256으로 늘려가며 정보를 추출하였다. 다시 사용자가 인식할 수 있는 이미지로 출력하기 위해 UpSampling을 4번 통과하였다. 각 필터의 크기는 DownSampling의 역순으로 256, 128, 64, 32 이며 32는 InputLayer를 거쳤을때 출력된 Layer의 차원과 같다. 결과적으로 나온 Channel_out layer는 Activation=Softmax 로 설정하여 이미지 내에 해당 pixel이 목표하는 부분인지 확률로 계산되게 만들었다. Channel_out 은 1x512x512x2로 마지막 x2는 각각 목표를 출력하는 마스크, 목표 만을 제외한 이미지를 출력하는 마스크를 뜻한다.
e) Compiler :
def Compiler(model):
return model.comiple(optimizer='rmsprop', loss=SparseCategoricalCrossentropy(from_logits=True), metrics=['accuracy'])Proto-Unet 과는 다르게 Loss 값을 SparseCategoricalCrossentropy 를 사용하였다. Proto-Unet은 loss='binary_crossentropy'를 사용하였지만, 이미지를 Categorical 하게 분류된 One-Hot encording이 아니기때문에 loss를 바꾸는게 맞겠다는 판단을 하게 되었다. 여러가지 loss 함수를 사용하여 Trial&Error를 반복한 끝에 실험적으로 나온 결과이다.
f) Fit & Saver
def Fit(model, X, Y, BatchSize=0, Epochs=0, ValidationSplit=0):
return model.fit(X, Y, batch_size=BatchSize, epochs=Epochs, validation_split=ValidationSplit)
def Saver(model, name):
return model.save(name)Fit은 컴파일이 완료된 모델을 Fit 함수를 통해 학습시키는 부분이다. Saver는 만들어진 모델을 미리 저장하는 부분이다. 아무래도 Colab 환경이기 때문에 가끔 런타임이 끊어지면 몇 시간을 돌린 결과가 사라지기 때문에 필수적으로 필요한 함수였다. 이들은 keras에 있는 기본함수를 단순히 return한 함수이다.
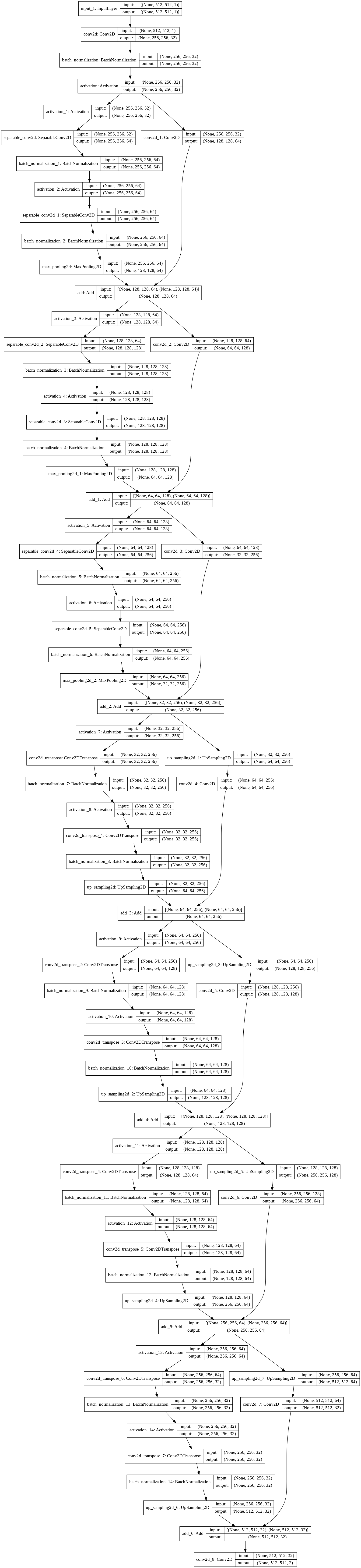
{Fig.01 Completed Unet model}
-
3. Prediction :
a) Predictor
def Predictor(model, Test_img):
# Test image Preprocessing
img_test = cv2.imread(Test_img, cv2.IMREAD_GRAYSCALE)
Img_width, Img_height = img_test.shape()
img = cv2.equalizeHist(img_test) / 255
img_resized = img.reshape(-1, Img_width, Img_height, 1)
# Segmentation result
result = model.predict(img_resized)
result_r = result.reshape(Img_width, Img_height, 2)
result_new = np.transpose(result_r, (2, 0, 1))
scaler = MinMaxScaler()
normalres1 = scaler.fit_transform(result_new[0])
After_seg1 = img * normalres1 # Target
#normalres2 = scaler.fit_transform(result_new[1])
#After_seg2 = img * normalres2 # rests
return After_seg1나온 결과물을 마지막으로 가공시켜 결과를 출력하는 모듈이다. 테스트 해야할 이미지에 대해 학습된 모델로 Predict를 진행한다. 마스크에 대해 표준화를 진행시켜 준 뒤, 이를 테스트 이미지에 씌워주면 결과적으로 목표했던 부분만 출력할 수 있다.
-
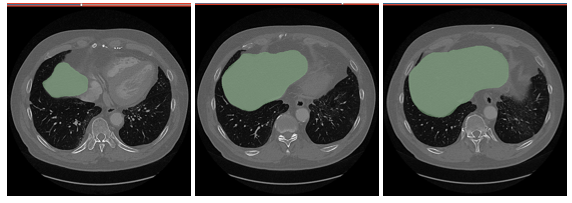
4. Result : 2D
a) Image with Target region
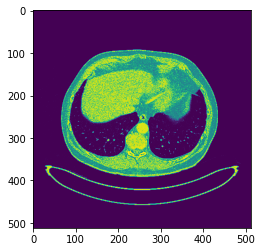
{Fig.02 MRI image with Liver region}
완성된 모델을 지속적으로 학습시킴에 따라 위와 같은 Test image 로 prediction 을 실시하였다. 위의 이미지는 Segmentation 을 목표가 존재하는 이미지이다. 결과는 이하와 같다.

{Fig.03 Prediction at Epoch 100}
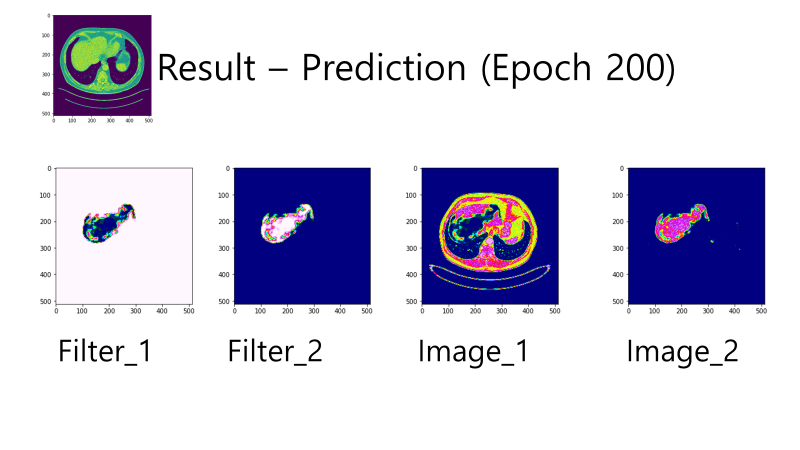
{Fig.04 Prediction at Epoch 200}
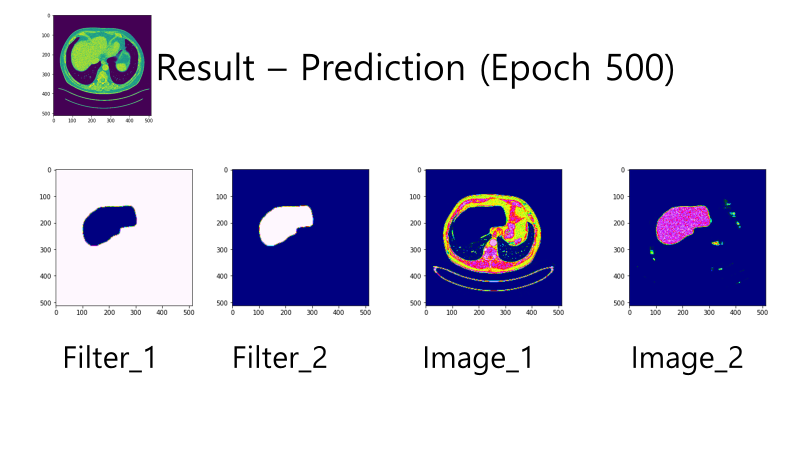
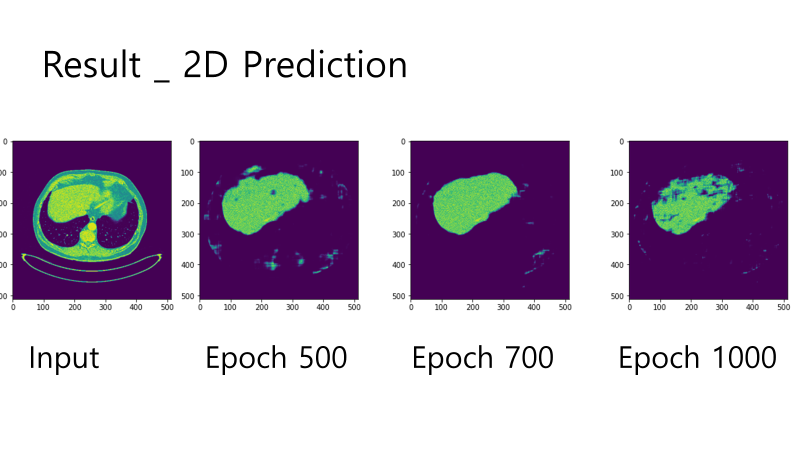
{Fig.05 Prediction at Epoch 500}
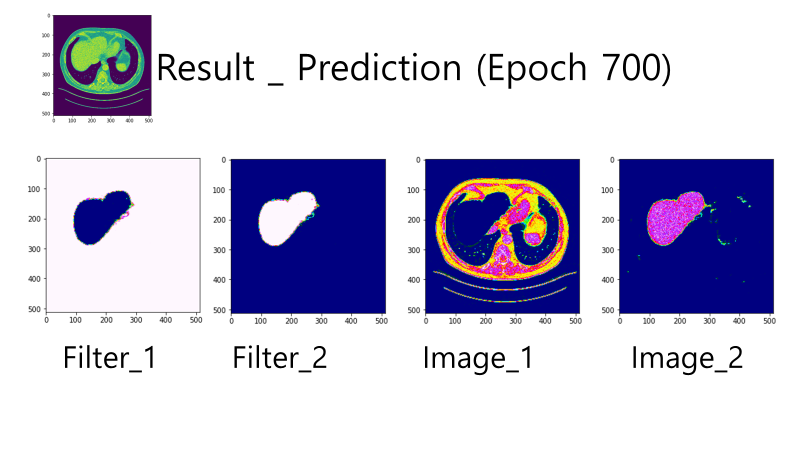
{Fig.06 Prediction at Epoch 700}
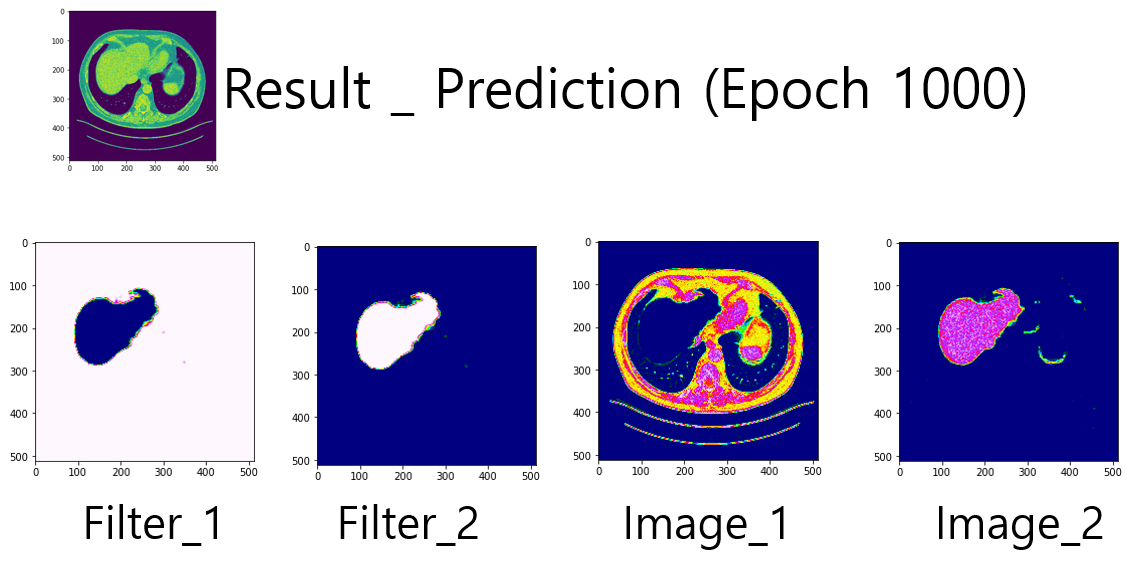
{Fig.07 Prediction at Epoch 1000}
{Fig.08 GT image and Predicted image by the model in Epoch= 500, 700, 100}
-
Epoch 에 따라 학습된 결과들을 확인할 수 있다. Epoch100 에서 Segmentation 의 목표인 Liver Region 의 윤곽이 들어났다. Epoch200 에 대해서 Liver Region 의 많은 반 이상의 부분을 Segmentation 한 것을 확인할 수 있다. 나아가 Epoch500 에 도착해서 목표와 상당히 유사한 윤곽을 잡아나가는 것을 확인하였으며, Epoch700 에서 Ground Truth 에 가장 근접하게 모델이 Prediction 을 수행하였다. 하지만 Epoch1000 에서는 오히려 Liver 의 이미지가 잘려나갔는데, 이를 Overfitting 으로 보기로 하였다.
b) Image without Target region
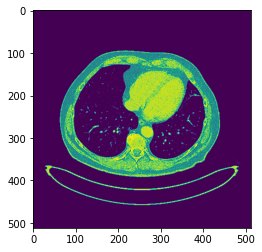
{Fig.09 MRI image without Liver region}
하지만 Liver region 이 없는 이미지에 대해서도 확인을 해야하기 때문에, 가장 Ground Truth 와 비슷했던 Epoch500, Epoch700 모델을 사용하여 위와 같은 이미지로 새롭게 Prediction 을 실시하였다. 결과는 이하와 같다.
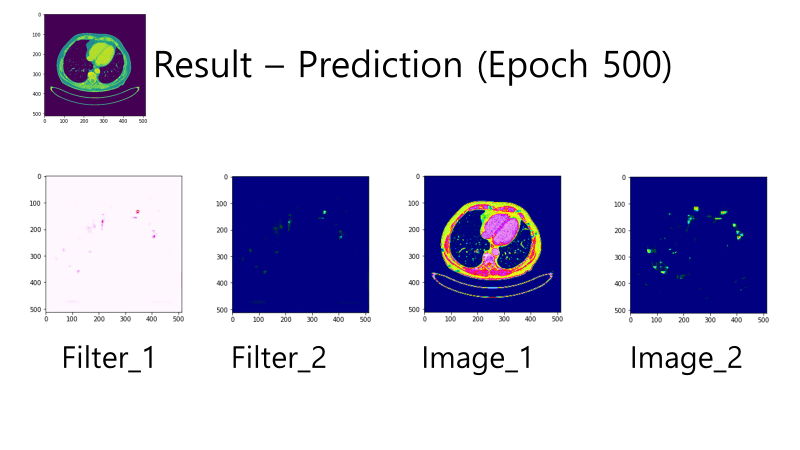
{Fig.10 Prediction by Epoch=500 model}
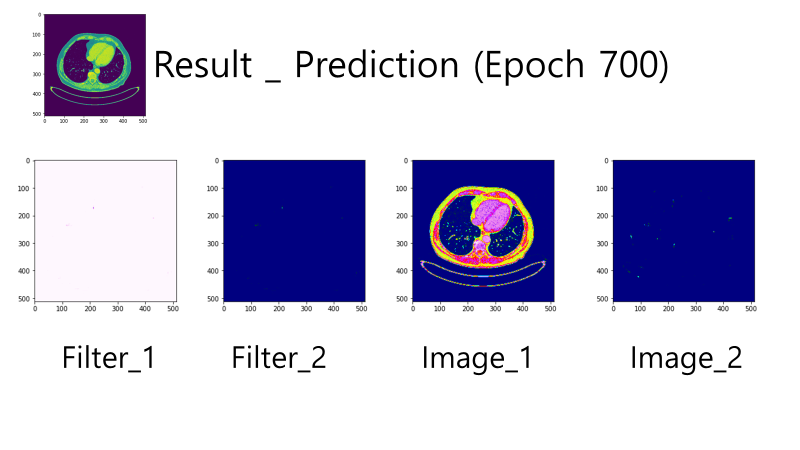
{Fig.11 Prediction by Epoch=700 model}
-
목표인 Liver region이 존재하지 않는 이미지이기 때문에, 이론적으로는 Image2 에서는 아무것도 나오면 안된다. 이에 가장 부합한 결과는 Epoch700 모델이다. 따라서 Epoch700에서 저장된 모델을 완성 모델로 설정하였다.
-
5. Result : 3D
a) Result in 3D slicer
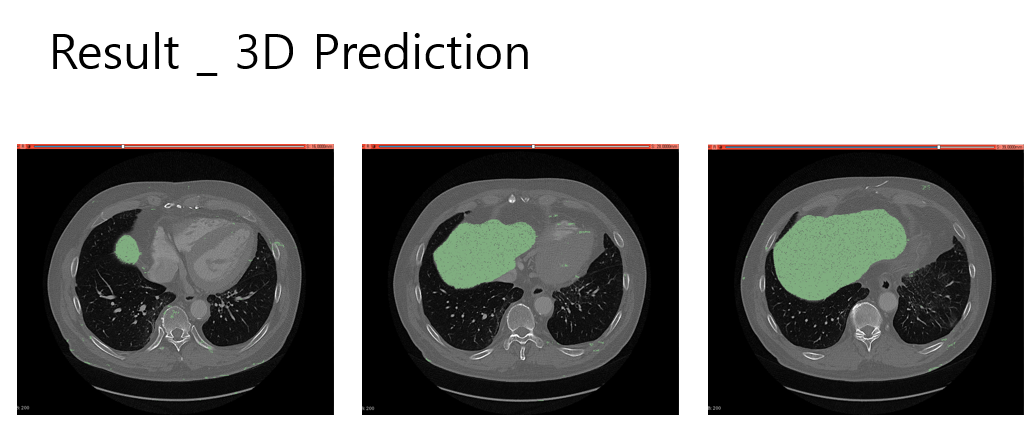
{Fig.12 Predictions by Completed model}
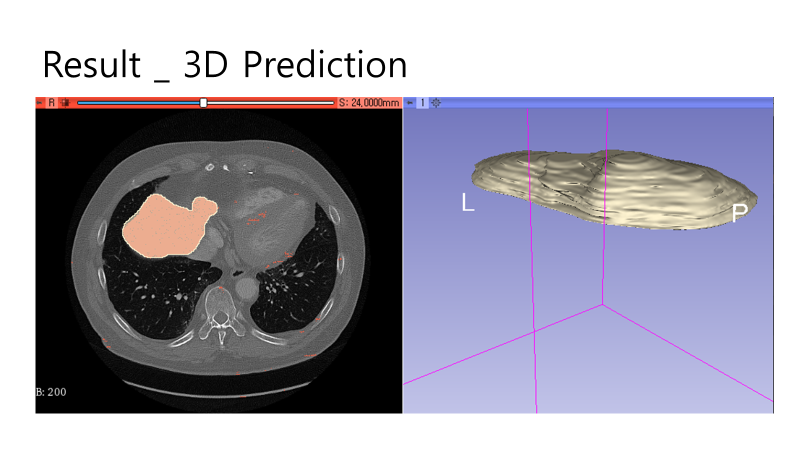
{Fig.13 Predictions colored in red and GT region colored in white}
{Fig.14 Predictions colored in red and GT region colored in white}
-
완성된 모델에 이미지를 Axis 방향으로 연속적인 Prediction 을 진행한 결과이다. Fig.12 는 Predicted region 이 Green 으로 마킹되었다. 시각적으로는 다양한 크기의 target region에 prediction 이 잘 마무리 된 것으로 확인된다.
Fig.13 ~ Fig.15 는 연속적인 이미지를 다시 3D slicer 에서 출력한 결과이다. Fig.13 과 Fig.14 에서는 Ground Truth image와 Prediction이 매우 잘 맞는 것을 볼 수 있다.
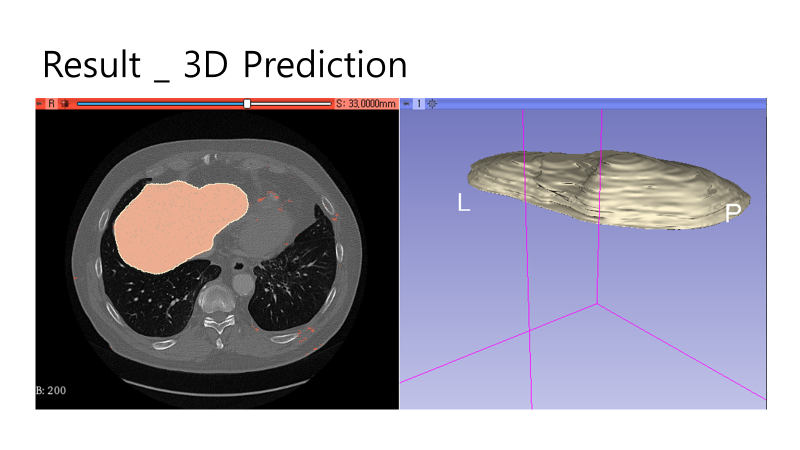
b) Problems & For Improvement
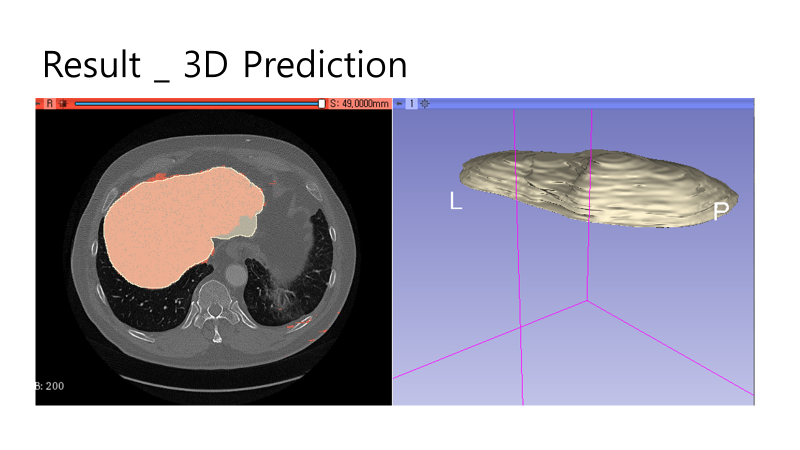
{Fig.15 Predictions colored in red and GT region colored in white}
가장 큰 문제점은 Fig.15 와 같이 Prediction region 과 GT region이 다른 이미지들이 존재한다는 것이다. 완성된 모델 (Epoch=700)에서 DICE Score 가 아닌 단순한 Accuracy는 99.04% 이다. 하지만 이는 target region이 아닌 부분에 대한 정확도 역시 포함되기 때문에 의미가 퇴색된다. 또한, 앞선 이미지들에도 target region 이 아닌 부위에 작게나마 Island 들이 생성된 것을 볼 수 있다. 이를 해결하기 위해 Argmax 또는 Image Filtering 방법을 통해 target region 내의 작은 hole 들과 target region 밖에 작은 island 들을 지워내는 작업을 수행해도 좋을 듯 하다.
