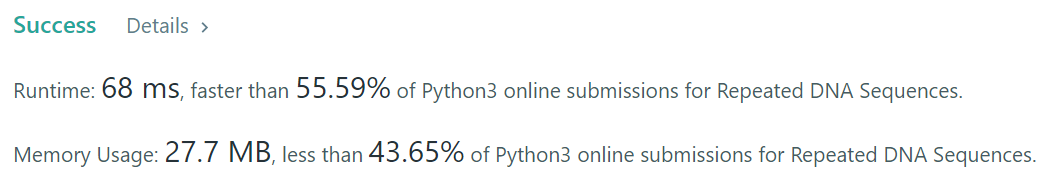
📃 문제 설명
(MEDIUM) https://leetcode.com/problems/repeated-dna-sequences/
- 주어진 DNA sequence에서 두 번 이상 반복되는 10자리 substring sequence를 전부 출력하는 문제이다. 해당 DNA sequence는 최대 100,000자리까지 길어질 수 있어 꽤나 길다고 할 수 있다.
🔎 문제 접근
- DNA sequence에서 발생할 수 있는
모든 10자리 substring에 대한 dictionary를 제작해주어 문제를 해결했다. - dictionary에 해당 key가 존재하는지 확인하기 위해 key 배열을 출력해 확인하곤 하는데, 나는
try - except문을 사용하기를 선호한다. 해당 key 값에 해당하는 value 값을 1씩 올려주는 operation을 진행해, 에러가 생기면 key가 없는 것이고 에러가 없다면 해당하는 key가 있는 것이기 때문에 계산 속도가 조금이나마 빨라지지 않을까 생각한 나름의 잔머리이다.
✨ 문제 풀이
class Solution:
def findRepeatedDnaSequences(self, s: str) -> List[str]:
seq_dict = dict()
ans = []
for i in range(len(s) - 9):
seq = s[i:i+10]
try:
seq_dict[seq] += 1
if seq_dict[seq] == 2:
ans.append(seq)
except:
seq_dict[seq] = 1
return ans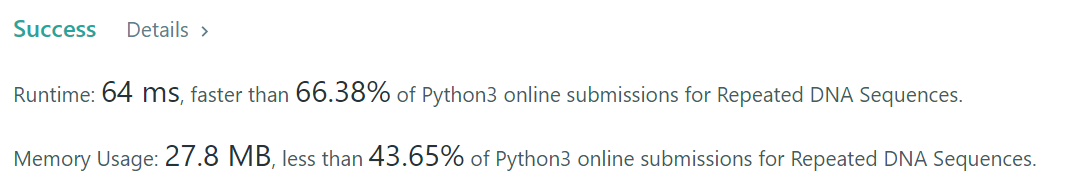
-
실제로 keys() 내에 존재 여부를 확인하는 코드와의 시간을 비교해본 결과, try - except 문을 사용하는 게 미미하게나마 빠름을 확인할 수 있었다.
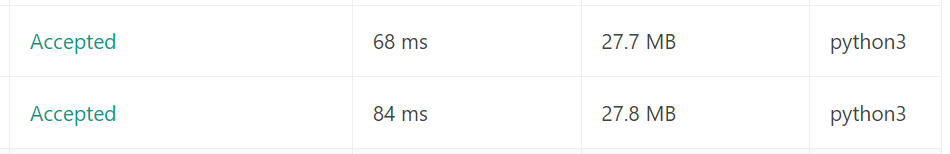
-
44ms 정도의 속도를 보이는 코드들은 모두
set을 사용했다. in 함수를 사용함에도 불구하고 더 빠르다니, 만약 TLE가 걸린다면 set으로 변환할 수 있는 부분이 있나 확인해보는 것도 좋을 것 같다.