Overview
Problem
- non-coding DNA 변이가 유전자 조절에 미치는 영향을 예측하기
Pain Point
- 입력 sequence의 길이가 길면 해상도가 떨어지고, 해상도를 높이면 입력 범위가 제한되었다.
- 특정 task(ex. 발현량, splicing)만 예측할 수 있는 모델이 대부분이었다 (generalization X)
- 단일 변이에 대한 다양한 분자적 영향 평가가 잘 되지 않았다.
Focus Point
- Long sequence-context at high resolution: 최대 1백만 bp 길이에, base 단위 예측 가능
- Comprehensive multimodal prediction: gene expression, transcription initiation, chromatin accessibility, histone modifications, transcription factor binding, chromatin contact maps, splice site usage, splice junction coordinates and strength 등을 동시에 예측
- Efficient variant scoring: 원본 sequence와 mutate된 sequence를 비교하여 초단위 연산으로 변수 영향 평가
- Novel splice-junction modeling: 변이가 splicing junction 위치 및 사용량에 미치는 영향을 직접 모델링
Limitation
- 매우 먼 (>100kb) 거리 조절 인자 예측 어렵다.
- 특정 세포 수준 패턴은 아직 완벽히 포착되지 않았다. → 세포/조직 특이성 강화 필요
- 분자 수준 예측에 초점을 맞추었으므로 개인 전체 유전체에서 조절 맥락을 통합적으로 예측하는 데에는 적합하지 않다. (특히, 여러 개의 변이가 상호작용하는 polygenic context에서는 제한적)
- 환경, 발달, 생리적 융합 효과를 반영하지 않으므로 복잡한 표현형 예측(ex. 자폐, 암과 같은 질병으로 이어질 것인가?)에 한계가 있다.
Details
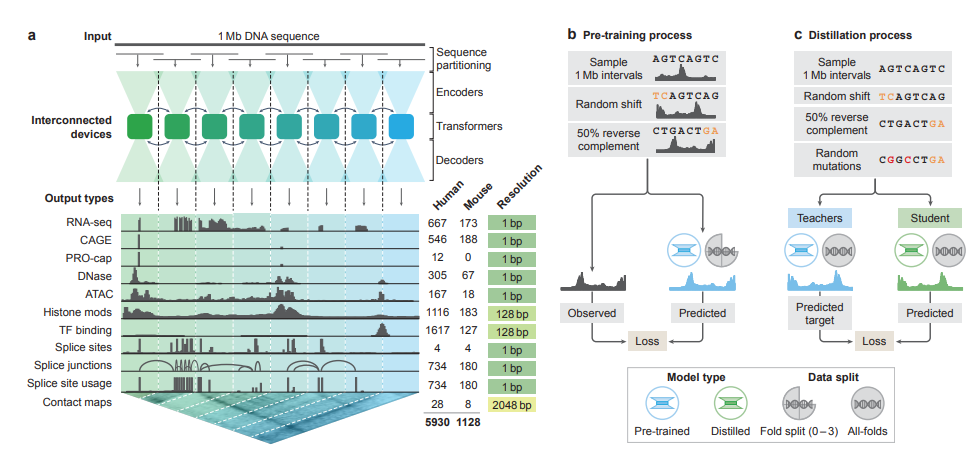
Model Architecture
- convolution layers: 짧은 motif 추출
- Transformer layers: 전체 sequence 간 장거리 정보 통합
- Task-specific heads: 각각의 분자 기능적 예측 출력
Evaluation
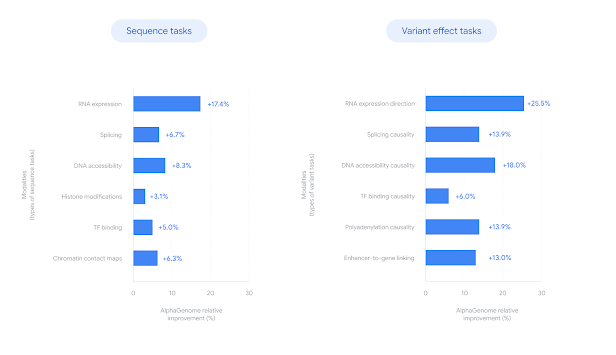
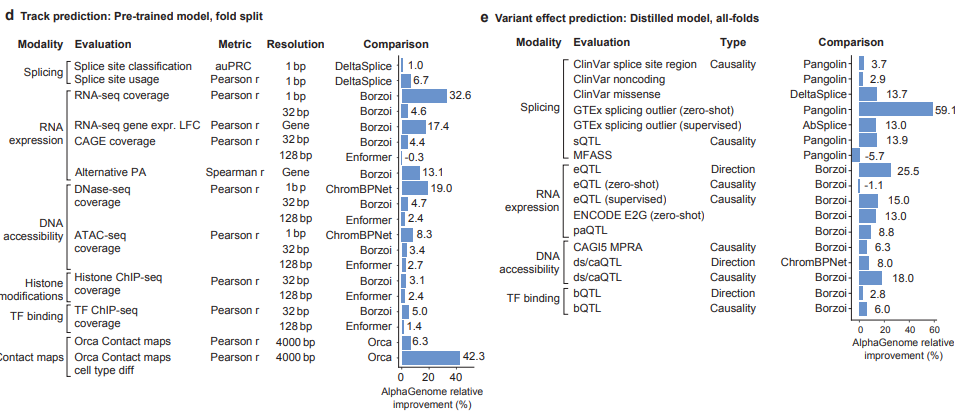
- Benchmark: 24개 single sequence 예측 task 중 22개, 변이 효과 예측 task 24/26개에서 SOTA
- case study: T-ALL 관련 TAL1 유전자 인접 변이에 대해 MYB motif 생성 예측으로 기전 확인
- dataset: ENCODE, GTEx, 4D Nucleome, FANTOM5 (인간/쥐) 등의 염기서열, 분자 실험 데이터를 다량 활용
Reference
- AlphaGenome paper: https://www.biorxiv.org/content/10.1101/2025.06.25.661532v1
- Deepmind webpage: https://deepmind.google/discover/blog/alphagenome-ai-for-better-understanding-the-genome/
- ChatGPT
