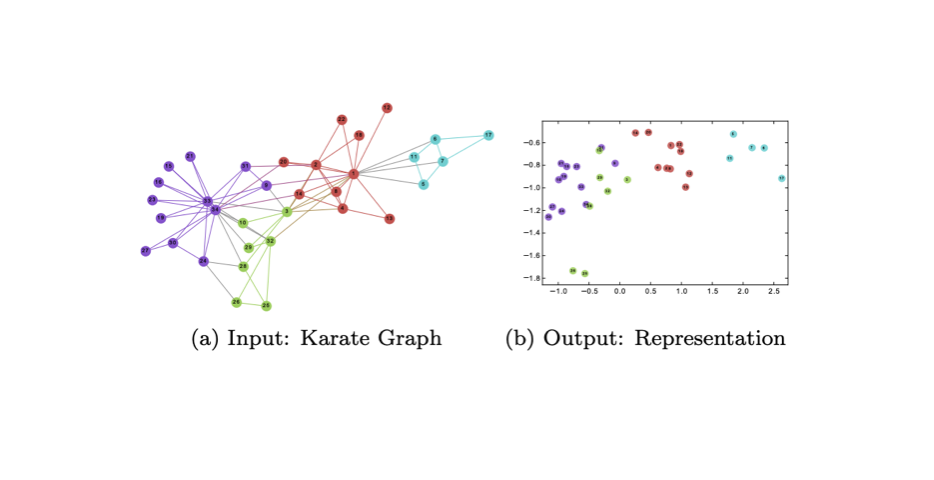
위의 그림은 karate club 의 34명의 회원들을 대상으로한 소셜 네트워크 그래프입니다. 위의 그림이 DeepWalk 입력과 출력을 잘 보여주고 있습니다.
오늘은 그래프 형태의 데이터를 효과적으로 임베딩할 수 있는 방법 중 하나인 DeepWalk 에 대해서 써보려고 합니다. 정확히 말하자면 그래프의 노드 를 임베딩할 수 있는 방법인데요. 핵심을 요약하면 다음과 같습니다.
- 데이터를
그래프형태로 나타낸다.random walk를 활용해 정해진 길이의walk만큼 sampling 한다.skip gram을 학습시킨다.
모든 과정을 이해하기 위해서는 먼저 skip gram 에 대해서 이해하면 좋습니다. 오늘 포스팅에서는 DeepWalk 를 이해하기 이해 하기 위해 필요한 사전 지식과 관련된 코드를 함께 포스팅 해보려고 합니다.
1. skip-gram
skip-gram 은 word2vec 의 방법 중 하나입니다. 대상 단어와 주변 단어를 관계를 이용해 함께 자주 등장한 단어일수록 유사한 백터공간에 표현하는 것이 목적입니다. 입력은 단어 뭉치이고 학습을 완료했을 때 최종 결과 값은 가중치 행렬입니다. 이 가중치 행렬은 각 단어의 벡터 값을 포함하고 있습니다.
skip gram 은 대상 단어가 입력으로 들어왔을 때 주변 단어를 예측하는 식으로 학습이 진행됩니다. window size 는 대상 단어가 주어졌을 때 주변 단어를 몇 개 까지 확인할 것인지를 나타내는 하이퍼파라미터 입니다. 만약 window size 가 2라면 대상 단어의 양 옆 2개의 단어까지를 학습합니다.
['나는', '그래프', '이론', '공부', '열심', '어제', '카페'] 라는 문장의 단어들을 skip-gram 을 통해서 학습한다고 해봅시다. (실제로는 훨씬 더 많은 단어로 구성된 말뭉치를 학습하게 됩니다.)
우리의 예시에서는 window size = 2 으로 가정하고 설명하겠습니다.
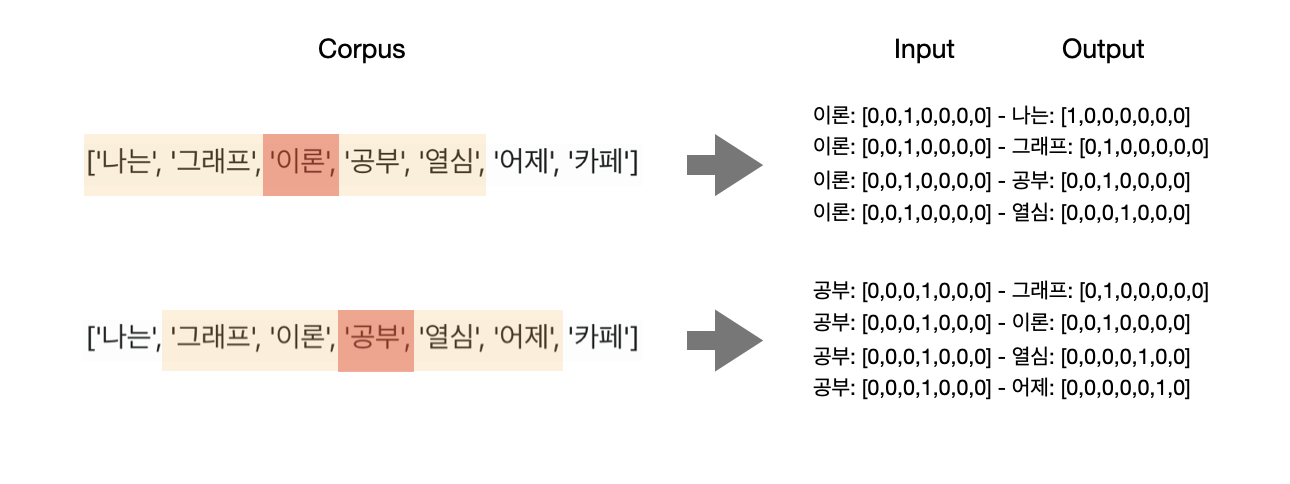
사실 학습의 첫번째 스텝은 '나는'이 대상 단어이고 '그래프', '이론'이 주변단어 입니다. 앞이나 뒤에 존재하는 대상단어가 존재하지 않을 때는 그 부분을 생략하고 학습을 진행합니다. 깔끔한 예시를 위해 주변 단어가 모두 존재하는 부분을 예로 가져왔습니다.
학습의 과정을 살펴보면 '이론' 이라는 단어가 들어왔을 때 주변 단어인 '나는', '그래프', '공부', '열심' 이라는 단어가 등장하는 방식으로 학습이 진행됩니다. 그 다음 스텝은 '공부' 라는 단어가 들어왔을 때 '그래프', '이론', '열심', '어제' 라는 단어가 등장하는 방식으로 학습이 진행되겠습니다.
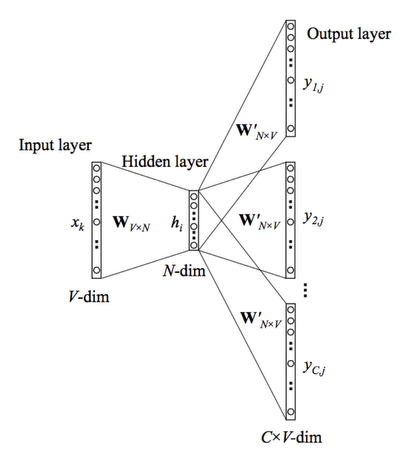
skip-gram 의 아키텍처는 생각보다 단순합니다. 은닉층의 레이어 개수가 1개인 신경망 모델을 학습하는 방식입니다. 입력(대상 단어)가 주어졌을 때 출력(주변 단어)를 예측하는 방식으로 가중치 행렬 (W, W') 를 학습하게 됩니다. 기존의 one-hot encoding 형태로 표현했던 단어들을 더 밀집된 저차원으로 임베딩을 시킬 수 있는데요. 이 차원에 해당하는 값이 은닉층의 노드의 개수(N-dim)입니다. N 값을 200으로 하게되면 은닉층의 노드가 200개인 신경망 모델을 학습하게되는 것이고 최종적으로 각 단어를 200차원의 임베딩 벡터로 표현할 수 있습니다.

앞서서 모델의 최종 결과값이 가중치 행렬이라고 했는데요. 위의 그림에서 W가 최종 결과 값이 됩니다. W의 형태를 보면 V(전체 단어의 개수) x N(임베딩 차원) matrix 입니다. 만약 '이론' 이라는 임베딩 벡터를 알고싶으면 가중치 행렬과 이론의 one-hot vector를 곱해주면됩니다.
그래프의 노드를 임베딩 시키는 방식 중 하나인 DeepWalk 는 텍스트를 임베딩하는데 활용되는 skip-gram 방식을 활용하는데요. 그래프 형태의 입력을 위의 예시의 말뭉치의 형태로 나타냅니다.
2. 그래프
2.1 용어
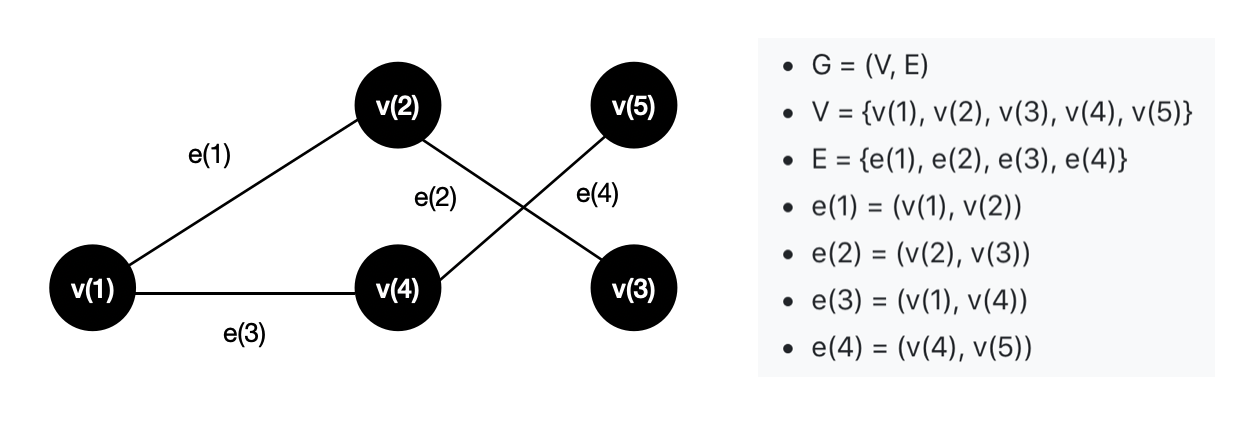
그래프(G)는 노드(V) 와 간선(E) 으로 표현할 수 있는 자료 구조의 한 종류입니다. 그래프의 종류는 크게 방향성 그래프(DiGraph) 와 비방향성 그래프(Graph)로 나눌 수 있습니다. 위의 그래프는 방향성이 없는 비방향성 그래프입니다.
다음으로 워크(walk) 는 어떤 노드를 출발해서 다른 노드로 도달하기 위한 인접한 노드의 순서열을 의미합니다. 위의 그래프에서는 [v(1) - v(2) - v(3)]와 [v(1) - v(4) - v(5)]를 예로 들 수 있습니다.
그래프를 NLP 관점에서 바라 보면, 그래프 = 문서, 워크 = 문장, 노드 = 단어 로 빗대어 표현할 수 있습니다. 즉, '그래프라는 문서는 워크라는 문장 속 노드라는 단어로 구성되어있다.' 라고 설명할 수도 있겠습니다. 단어들을 임베딩하는 방식이 skip-gram이니 그래프에 존재하는 노드를 임베딩 하는데 활용할 수도 있겠습니다. (참고로 그래프 자체를 임베딩하는 방식 중 하나인 Graph2vec 은 문서를 임베딩 하는 방법 중 하나인 Doc2vec를 활용합니다.)
2.2 random walk
DeepWalk는 위의 그림에서 봤을 때 v(1) ~ v(5)를 임베딩 시키는 과정입니다. DeepWalk는 random walk 를 활용해서 모델의 Input을 만듭니다. random walk는 말 그대로 노드 위를 걸어다니는 것을 의미하는데 한 노드에서 출발해서 간선으로 연결된 임의의 이웃으로 이동하고 이동된 노드의 임의의 이웃으로 이동하는 일련의 과정을 의미합니다.
위의 그래프를 예로 들면 [v(1), v(4), v(5), v(1)], [v(3), v(2), v(3), v(2)], [v(4), v(1), v(2), v(3)] ... 등의 random walk를 만들 수 있습니다. skip gram의 입력을 만들기 위해서는 random walk의 개수와 길이를 사전에 지정해줘야 합니다. 논문에서는 각 노드 별로 32 ~ 64개 정도의 random walk를 만들고 길이는 40 정도가 적당하다고 합니다.
위의 과정을 통해 노드로 구성된 워크를 만들 수 있습니다. 이제 skip-gram의 입력으로 넣어주기만 하면 각 노드들의 벡터 값을 추론할 수 있습니다.
3. Covid-19 citation graph - embedding using DeepWalk
Covid-19 와 관련된 논문 들의 인용(citation) 관계를 DeepWalk를 통해 임베딩을 해보는 예시를 들어보려고 합니다. 관련 데이터 셋은 이곳 에서 확인하실 수 있으며 실제 코드를 통한 이해를 돕기 위해 이곳 커널을 참고하였습니다.
3.1 Load data
먼저 관련 library 들을 불러옵니다.
networkx: 데이터를 그래프 형태로 표현하는데 좋은 패키지gensim: skip-gram 을 활용할 수 있는 패키지
import numpy as np
import pandas as pd
import os
import json
from multiprocessing import Pool
import random
import pickle
import re
from functools import reduce
import networkx as nx
from gensim.models import Word2Vec
import gc데이터를 확인해봅시다. 전체 논문의 개수는 1,625,243편 입니다.
filenames_list = []
for dirname, _, filenames in os.walk('/kaggle/input'):
for each_filename in filenames:
filenames_list.append(os.path.join(dirname, each_filename)) 3.2 Graph 형태로 데이터 표현
대부분의 논문들은 인용 논문들을 따로 적어놓는데요. 주어진 데이터셋의 어떤 논문을 인용했는지 아래와 같은 json 형태로 저장해놓고 있습니다.
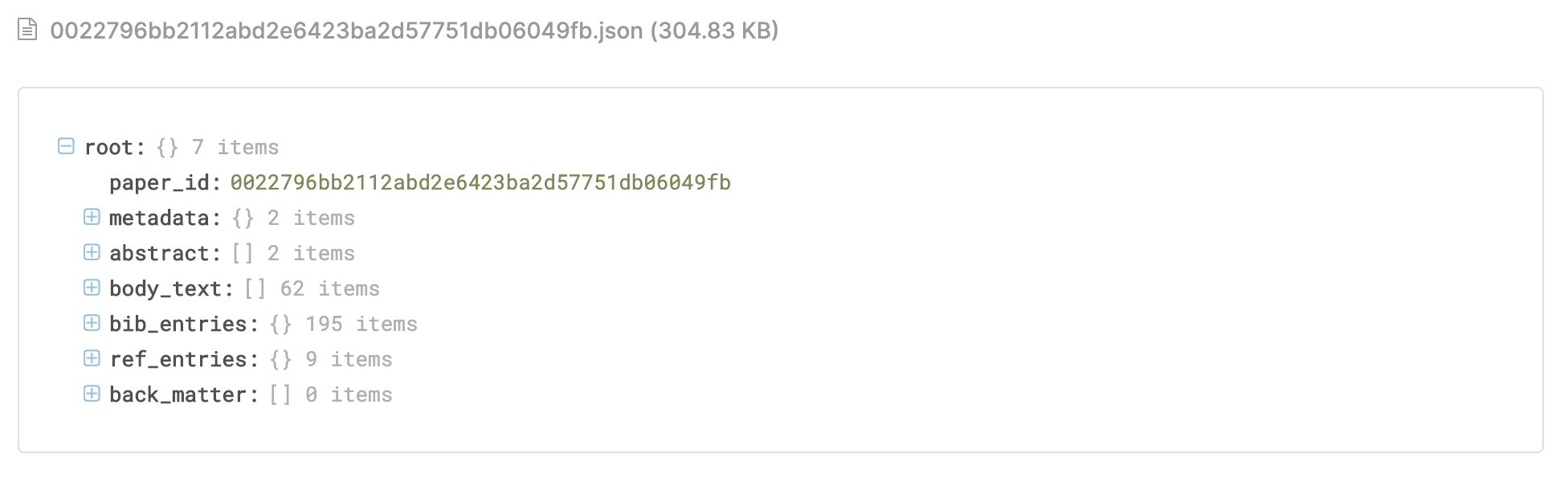
research_paper_title_list = []
for filename in filenames_list:
if filename.split(".")[-1] == "json":
ifp = open(os.path.join(dirname, filename))
research_paper = json.load(ifp)
research_paper_title_list.append(research_paper["metadata"]["title"])
for each_ref in research_paper["bib_entries"]:
research_paper_title_list.append(research_paper["bib_entries"][each_ref]["title"])
networkx 패키지를 활용해서 데이터 셋을 그래프 형태로 표현하기 위해서는 아래와 같이 딕셔너리(dictory) 형태로 데이터를 표현해놓는 것이 편합니다.
paper_id_dict= dict(zip(research_paper_title_list, list(map(lambda x: str(x), range(len(research_paper_title_list))))))
id_paper_dict = dict(zip(paper_id_dict.values(), paper_id_dict.keys()))
paper_undirected_degree_dict= dict(zip(research_paper_title_list, [0]*len(research_paper_title_list)))
adj_mat = {}
for filename in filenames_list[:100]:
if filename.split(".")[-1] == "json":
ifp = open(os.path.join(dirname, filename))
research_paper = json.load(ifp)
adj_mat[paper_id_dict[research_paper["metadata"]["title"]]] = [paper_id_dict[research_paper["bib_entries"][each_key]["title"]] for each_key in research_paper["bib_entries"]]
paper_undirected_degree_dict[research_paper["metadata"]["title"]] += len(adj_mat[paper_id_dict[research_paper["metadata"]["title"]]])
for each_key in research_paper["bib_entries"]:
paper_undirected_degree_dict[research_paper["bib_entries"][each_key]["title"]] += 1
아래의 코드를 활용하면 그래프 형태의 데이터를 시각화 해볼 수 있는데요. 몇 개의 논문만을 샘플링해서 그래프를 시각화 해보았습니다. (그래프가 좀 예쁘게 나오기를 기대해는데 좋은 예시를 샘플링하는게 까다롭군요 ^^..)
from IPython.core.display import Image
from networkx.drawing.nx_pydot import to_pydot
citation_graph = nx.from_dict_of_lists(adj_mat)
d2 = to_pydot(citation_graph)
d2.set_dpi(300)
d2.set_rankdir("LR")
d2.set_margin(1)
Image(d2.create_png(), width=100)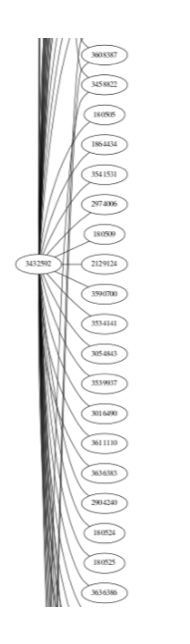
메모리 제한 때문에 한 논문에서 인용한 논문 중 25개만 샘플링한 pruned_adj_mat 를 새로 만들어줬습니다.
pruned_adj_mat = {}
for each_key in adj_mat:
freq_ref = [paper_undirected_degree_dict[id_paper_dict[each_id]] for each_id in adj_mat[each_key]]
ref_freq_dict = dict(zip(adj_mat[each_key], freq_ref))
pruned_adj_mat[each_key] = sorted(adj_mat[each_key], key=lambda x: ref_freq_dict[x], reverse=True)[:25]
3.3 random walk를 통한 sampling
이제 random walk를 통해 그래프로 부터 임의의 walk를 추출해줘야 합니다. 아래의 과정을 통해서 이를 수행할 수 있습니다. 위에서 설명했던 것 처럼 num_walks, walk_length를 통해 워크의 개수와 워크의 길이를 설정해줄 수 있습니다.
def random_walk(arg):
root_node, walk_length = arg
walk = [root_node]
for i in range(1, walk_length):
cur = walk[i-1]
neighbours = list(citation_graph.neighbors(cur))
if len(neighbours) > 0:
walk.append(random.choice(neighbours))
else:
walk = walk[:-1]
break
return walkdef deepwalk_random_walks(num_walks, walk_length):
nodes = list(citation_graph.nodes())
walks = []
for i in range(num_walks):
print("walk no. ", i)
random.shuffle(nodes)
with Pool(processes=32) as pool:
walks = walks + pool.map(random_walk, zip(nodes,[walk_length]*len(nodes)))
return walks여기서는 각 노드별로 20개의 워크를 랜덤 추출했으며 워크의 길이는 10으로 설정해줬습니다.
2번째 워크를 살펴보면 다음과 같습니다.
random_walks = deepwalk_random_walks(20, 10)
random_walks[2] > ['1058658',
'164703',
'1521202',
'21047',
'59640',
'39596',
'1622401',
'432526',
'432546',
'432526']3.4 skip-gram
random walk를 통해 skip-gram 의 input 을 만들어주었습니다. 이제 본격적으로 학습을 해봅시다.
gensim 패키지의 Word2Vec 을 활용하면 아주 간단하게 skip-gram 을 학습시킬 수 있습니다.
model = Word2Vec(random_walks,
size=32, # 임베딩 벡터의 차원 (32차원으로 임베딩)
window=4, # 고려할 앞 뒤 폭 (앞 뒤 4개의 노드)
min_count=1, # 사용할 노드의 최소 빈도 (1회 이하의 노드 무시)
sg=1) # 1: skip-gram / 0:cbow)결과의 몇몇 예시를 살펴봅시다. 코사인 유사도를 기준으로 'Discovery and Characterization of Novel Bat Coronavirus Lineages from Kazakhstan. Viruses' 노드와 가장 유사한 노드를 찾아봅시다.
def most_similar_papers(title, topn=20):
return [(id_paper_dict[each[0]], each[1]) for each in model.wv.most_similar(paper_id_dict[title], topn=topn)]
most_similar_papers('Discovery and Characterization of Novel Bat Coronavirus Lineages from Kazakhstan. Viruses')[:2]> [('Detection of rickettsial DNA from Ixodid ticks of the West Kazakhstan region',
0.9993804693222046),
('Tick-Borne Encephalitis Virus, Coxiella burnetii & Brucella spp. in Milk, Kazakhstan',
0.9993616342544556),
('Investigating the presence of Rickettsia spp. and Yersinia pestis in flea from the natural plague foci of Kazakhstan',0.9993240833282471)]4. 결론
4.1 요약
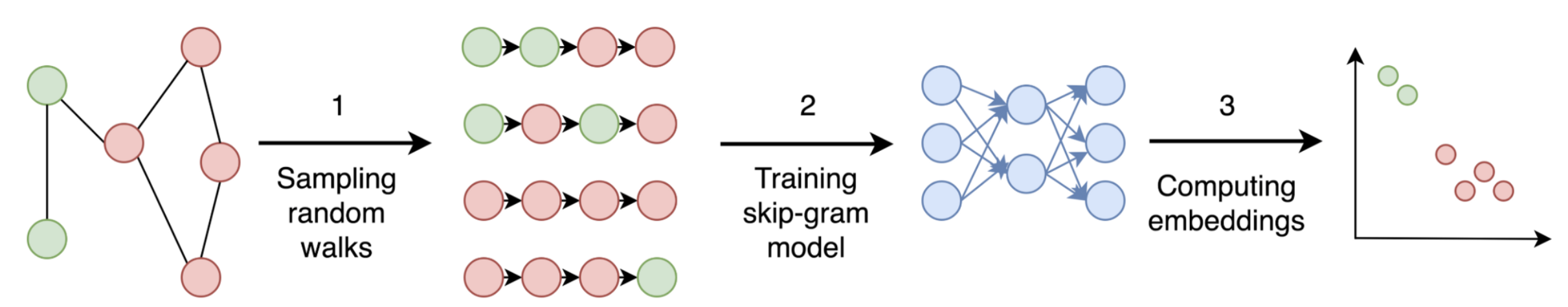
DeepWalk 는 그래프의 노드를 임베딩하는 방법입니다. 과정을 요약하면 아래와 같습니다.
(1) 데이터를 그래프 형태로 표현한다.
(2) random walk를 통해서 walk를 샘플링한다.
(3) 이 결과 값을 skip-gram을 통해 학습한다.
4.2 DeepWalk의 한계점
DeepWalk는 random walk를 임의로 만들기 때문에 각각의 노드의 이웃들 간의 더 강한 관계를 파악하지 못한다는 단점이 있습니다. 이를 보완하기 위해 node2vec, SDNE 와 같은 방법을 사용하기도 합니다. (추후에 관련된 내용도 포스팅 해보도록 하겠습니다.)
5. 느낀점
그래프를 NLP 관점에서 바라본 것이 저의 뇌피셜(?) 이지만 Deep Learning 과 관련된 많은 이론들은 서로 연결되어 있는 것 같습니다. 기초를 더욱 튼튼하게 공부해야 겠습니다. 최근부터 cs224w machine learning with graphs 를 듣기 시작했는데 그래프 이론 정말 매력적이고 활용도가 높은 것 같습니다.
모빌리티 에서도 그래프 이론을 접목시켜서 많은 것을 해볼 수 있을 것 같은데요. 한 예로 A에서 B까지 가는 이동수단을 생각해봤을 때. A, B를 하나의 노드로 간선을 걸리는 시간이나 거리로 생각한다면 (출발지, 도착지, 소요시간, 거리) 로 표현할 수 있는 거대한 그래프를 만들 수 있을 것 같습니다.
긴 글 읽어주셔서 감사합니다. 제 글이 도움됐다면 좋아요 하나씩 부탁드립니다.
Reference
- DeepWalk: Online Learning of Social Representations
- Word2Vec에 대한 자세한 설명이 포함된 정말 유명한 블로그 입니다.
- networkx 에 대한 좋은 설명이 포함된 사이트 입니다.
- Graph Embedding 에 관련된 전반적인 내용을 요약해놓은 자료입니다.
- 코드를 참고한 Kaggle kernel 입니다.
- Karate 데이터셋은 이곳에서 확인할 수 있습니다.

https://protospielsouth.com/user/77682
https://whyp.it/users/100591/chinabamboo
https://bulkwp.com/support-forums/users/chinabamboo/
https://spinninrecords.com/profile/chinabamboo
https://fyers.in/community/member/MAGfDcP7tF
https://velog.io/@chinabamboo/posts
https://robertsspaceindustries.com/en/citizens/chinabamboo
https://unityroom.com/users/chinabamboo
https://www.aseeralkotb.com/ar/profiles/china-bamboo-bfc-116365576188419692513-1755394900
https://mathlog.info/users/Y7CG8ma0cAeikQnPJW0IDMbf8EW2
https://mathlog.info/articles/RW0eKy1Cls8auLMkkODN
https://3dwarehouse.sketchup.com/by/chinabamboo
https://blog.ulifestyle.com.hk/chinabamboo
https://routinehub.co/user/chinabamboo
https://hub.docker.com/u/chinabamboo
https://pantip.com/profile/9010914#topics
https://tamilculture.com/user/china-bamboo
https://www.passes.com/chinabamboo
https://aiplanet.com/profile/chinabamboo
https://bookmeter.com/users/1612789
https://linkfly.to/70816G7up4g
https://www.papercall.io/speakers/chinabamboo
https://sketchersunited.org/users/273241
https://biomolecula.ru/authors/82748
https://linkr.bio/chinabamboo/store
https://smartprogress.do/user/752262/
https://swaay.com/u/bothbest/
https://2024.hackerspace.govhack.org/profiles/china_bamboo
http://www.ssnote.net/users/chinabamboo
https://www.notebook.ai/@chinabamboo
https://cofacts.tw/user/chinabamboo
https://bio.site/bambooflooring
https://www.myminifactory.com/users/chinabamboo
https://www.upcarta.com/profile/chinabamboo
https://chinabamboo.stck.me/
https://coub.com/chinabamboo
https://portfolium.com/chinabamboo
https://motion-gallery.net/users/817902