논문 리뷰에 앞서
논문 리뷰는 다회독을 하며 내용을 수정해 나갈 게시글입니다.
현재 1회독 중이며, Abstract, Introduction을 정리합니다.(22.10.26)
2회독 예정입니다.
논문 간단 요약
해당 논문에선 CNN로 코로나 바이러스 DNA Seq를 분석하여 SARS-CoV-2를 식별합니다. 또, 분류에 사용되는 Seq feature를 시각화 하고, primer를 디자인 합니다.
1회차 리뷰
Abstract
해당 논문에선 우선 CNN을 이용해 553개의 바이러스 DAN Seq 중에서 코로나 바이러스의 Genorm을 98.73%의 정확도로 분리합니다. 이후 네트워크의 동작을 분석하여 모델이 SARS-CoV-2를 식별하는 데 사용한 Seq를 밝혀냅니다. Seq 중 하나를 선택하여 프라이머 세트를 생성하고, 다른 프라이머 세트와 비교하여 테스트 합니다.
Introduction
인간의 SARS-CoV-2 감염을 확인하기 위해 RT-qPCR 검사가 사용되었습니다. 하지만, RT-qPCR검사는 거짓 음성 테스트 문제, 거짓 양성 반응 등의 문제점이 제기되었습니다. SARS-CoV-2 확산 억제를 위해 CT 스캔과 딥러닝을 결합한 진단도구가 제안되었으며, 향상된 탐지 정확도를 달성하였고, DNA 염기서열 분석 역시 해결책으로 제시되었습니다.
바이러스 염기 서열 분석을 위해 주로 FASTA, BLAST와 같은 정렬에 기반한 방법들이 사용 되었습니다. 하지만 이러한 방법은 cDNA가 공통 특징을 공유한다는 가정에 의존하며, 때문에 탐지를 위해 참조할 수 있는 기본 Sequence가 필수적으로 필요하기 때문에 종종 어려움을 겪습니다.
이러한 방법의 대안으로 딥러닝을 이용한 DNA Seq의 분류 방법이 제안되었습니다. 딥러닝을 이용한 기법에선 DNA Seq의 식별 및 분류에 미리 선택된 특징이 필요하지 않습니다. 제안된 방법에서는 One-hot Encoding과 CNN을 사용하여 DNA를 식별 및 분류하였습니다.
딥러닝을 기반으로 한 방법에선 Seq을 고정된 길이로 나누어 바이러스를 분류하였습니다. 하지만, 이러한 방법은 입력 Seq에 포함된 정보중 일부를 무시하는 부작용이 있으며, 고정된 길이를 다 채우지 못한 짧은 조각들 역시 무시됩니다. 이에 정렬에 기반하지 않는 새로운 방법이 연구되기 시작했습니다. 이후 제안된 모델들은 높은 정확도로 DNA를 분류하였지만, 분류 과정이 인간이 이해할 수 있는 정보가 없는, 블랙박스 절차를 거치기 때문에 해당 분류를 통해 얻을 수 있는 생물학적 통찰은 제한적이었습니다. 해당 논문에선 전문가에게 더 많은 이해를 제공할 수 있는 XAI 모델의 개발에 주목합니다.
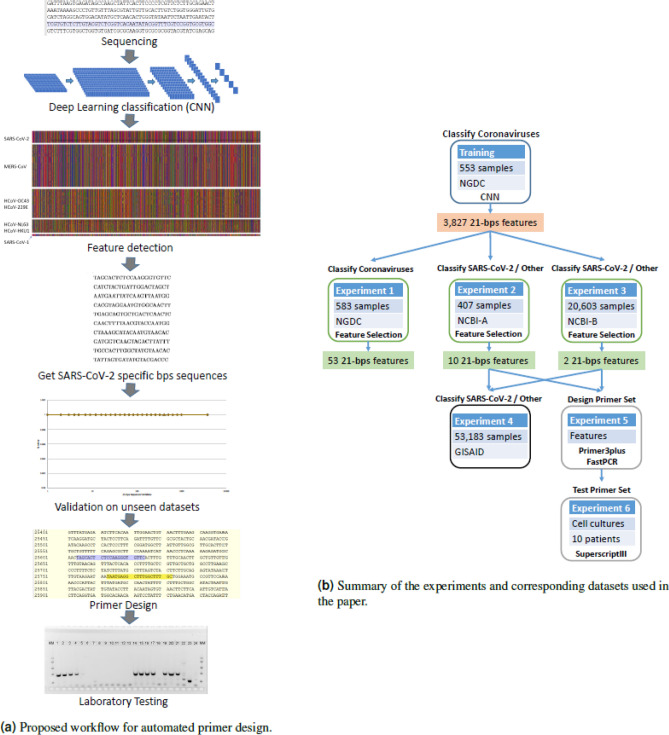
해당 논문에선 CNN을 사용하여 SARS-CoV-2를 포함한 다양한 코로나 바이러스를 분류하며, 모델이 바이러스를 분류하는 데 사용하는 대표적인 cDNA Seq를 발견합니다. 이후 훈련에 사용되지 않은 데이터세트를 통해 해당 cDNA Seq들을 검증하고 분류에 도움이 되는 Seq feature set를 만듭니다. 이 Seq들은 추후 전문가들에 의해 검사되고 분석될 수 있습니다. 이렇게 발견된 Seq중 일부는 해당 Seq의 확인만으로도 SARS-CoV-2를 식별하는 데 큰 도움을 줄 수 있기에 Primer가 될 수도 있습니다.
아래 그림은 프라이머 디자인을 위한 워크플로우(a)와 각 연구에 사용된 데이터세트와 실험 요약입니다.