참조: https://github.com/usuyama/pydata-medical-image
Retina image와 Lung CT에 대해서 딥러닝을 적용해 보고자 한다!
Diabetic Retinopathy Detection(당뇨망막병증)
우선 retina dataset을 다운받자!
용량이 82기가라 포기했다..!^^->정리만...!
https://www.kaggle.com/c/diabetic-retinopathy-detection/data?select=sampleSubmission.csv.zip
Diabetic Retinopathy
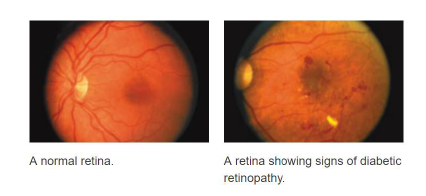
- 당뇨병 환자들에게 흔한 눈병
- 고혈당 수준은 망막 혈관에 피해를 준다!
- 흐린 시야와 시각 손해를 유발
DR: Severity Scale

input,output
Input: Retinal image, JPEG
Output: severity
Method overview
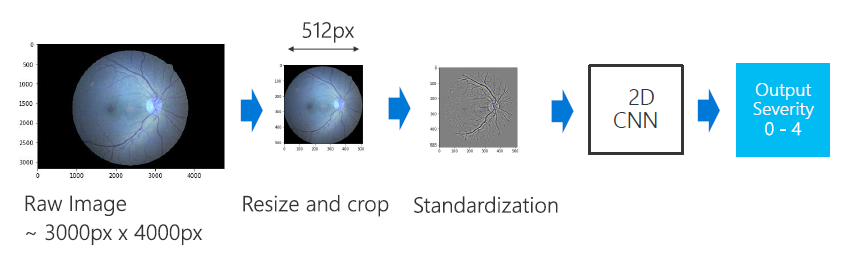
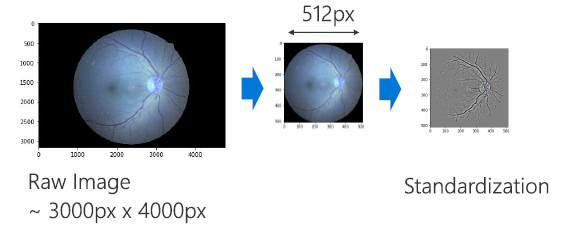
DR: Preprocess
- Crop and resize to 512x512(cv2.resize)
- Subtract local average(cv2.GaussianBlur, cv2.addWeighted)
DR: Data Augmentation 데이터 증강
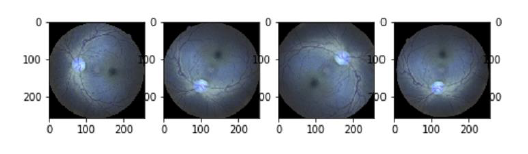
- Deep learning은 genaralization ablity를 위한 큰 데이터셋을 요구한다
- "Augment" imagesdom transformation
-EX: Rotate, Flip, Scale(Zoom), etc
-transformation을 거치면, 이 변환들에 대해서 불변해진다! 즉 영향을 안받는다는 의미
데이터 증강(Data Augmentation)은 적은 양의 데이터를 바탕으로 다양한 알고리즘을 통해 데이터의 양을 늘리는 기술
딥러닝 모델:2D CNN

Training
import cv2, glob, os
import numpy as np
import pandas as pd
# See Dr. Graham's preprocessing's method
# https://www.kaggle.com/c/diabetic-retinopathy-detection/discussion/15801
def estimate_radius(img):
mx = img[img.shape[0] // 2,:,:].sum(1)
rx = (mx > mx.mean() / 10).sum() / 2
my = img[:,img.shape[1] // 2,:].sum(1)
ry = (my > my.mean() / 10).sum() / 2
return (ry, rx)
def subtract_gaussian_blur(img):
# http://docs.opencv.org/trunk/d0/d86/tutorial_py_image_arithmetics.html
# http://docs.opencv.org/3.1.0/d4/d13/tutorial_py_filtering.html
gb_img = cv2.GaussianBlur(img, (0, 0), 5)
return cv2.addWeighted(img, 4, gb_img, -4, 128)
def remove_outer_circle(a, p, r):
b = np.zeros(a.shape, dtype=np.uint8)
cv2.circle(b, (a.shape[1] // 2, a.shape[0] // 2), int(r * p), (1, 1, 1), -1, 8, 0)
return a * b + 128 * (1 - b)
def crop_img(img, h, w):
h_margin = (img.shape[0] - h) // 2 if img.shape[0] > h else 0
w_margin = (img.shape[1] - w) // 2 if img.shape[1] > w else 0
crop_img = img[h_margin:h + h_margin,w_margin:w + w_margin,:]
return crop_img
def place_in_square(img, r, h, w):
new_img = np.zeros((2 * r, 2 * r, 3), dtype=np.uint8)
new_img += 128
new_img[r - h // 2:r - h // 2 + img.shape[0], r - w // 2:r - w // 2 + img.shape[1]] = img
return new_img
def preprocess(f, r, debug_plot=False):
try:
img = cv2.imread(f)
ry, rx = estimate_radius(img)
if debug_plot:
plt.figure()
plt.imshow(img)
resize_scale = r / max(rx, ry)
w = min(int(rx * resize_scale * 2), r * 2)
h = min(int(ry * resize_scale * 2), r * 2)
img = cv2.resize(img, (0,0), fx=resize_scale, fy=resize_scale)
img = crop_img(img, h, w)
print("crop_img", np.mean(img), np.std(img))
if debug_plot:
plt.figure()
plt.imshow(img)
img = subtract_gaussian_blur(img)
img = remove_outer_circle(img, 0.9, r)
img = place_in_square(img, r, h, w)
if debug_plot:
plt.figure()
plt.imshow(img)
return img
except Exception as e:
print("file {} exception {}".format(f, e))
return None
input_path = "../input"
df = pd.read_csv(os.path.join(input_path, "trainLabels.csv"))
train_files = glob.glob(os.path.join(input_path, "train", "*.jpeg"))
out_directory = "../preprocess/512/train"
if not os.path.exists(out_directory):
os.makedirs(out_directory)
def process_and_save(f):
basename = os.path.basename(f)
image_id = basename.split(".")[0]
if len(df[df['image'] == image_id]) < 1:
print("missing annotation: " + image_id)
return
target_path = os.path.join(out_directory, basename)
print("processing:", f, target_path)
if os.path.exists(target_path):
print("skip: " + target_path)
return
result = preprocess(f, 256)
if result is None:
return
# NOTE: Filter low contrast images for tutorial
std = np.std(result)
if std < 12:
print("skip low std", std, f)
return
if result is not None:
print(cv2.imwrite(target_path, result))
from joblib import Parallel, delayed
# Specify the number of cpu cores
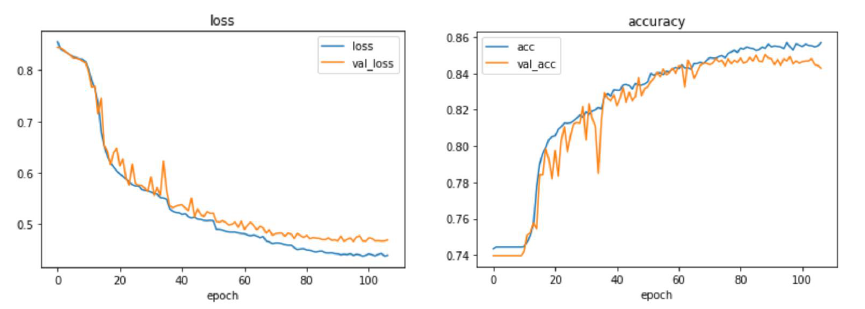
Parallel(n_jobs=16)(delayed(process_and_save)(f) for f in train_files)Training: Learning Curve
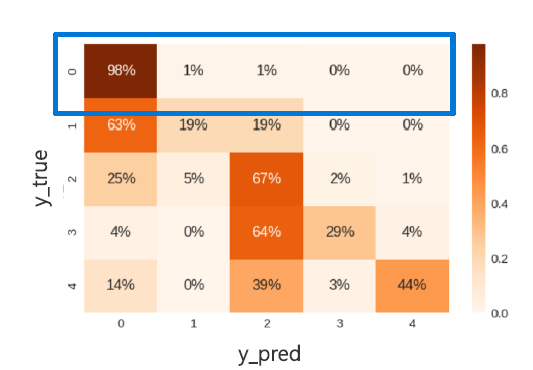
Result: Confusion Matrix
Lung Nodule
Lung Nodule(결절)
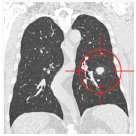
- 폐에서 작은 조직 덩어리
- CT상에서 동그란, 작은 그린자로 나타남
- 약 0.2 인치 (5 millimeters) ~1.2 인치 (30 millimeters)
- 항상 악성은 아니지만, 성장을 통해서 모니터링을 필요로한다.
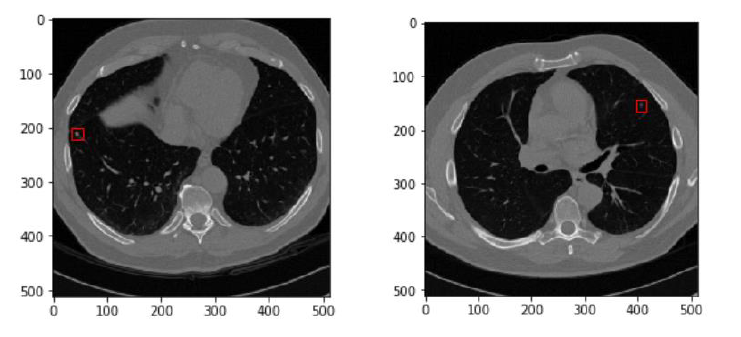
a needle in a haystack: 건초더미에서 바늘찾기!
거의 찾기가 힘들다... 불가능하다...
input output
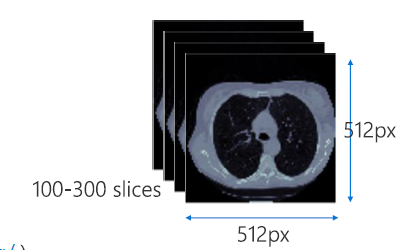
input: Lung CT Scam, 512x512
Output: Detect lung nodule location
Dataset: https://luna16.grand-challenge.org/
Method Overview
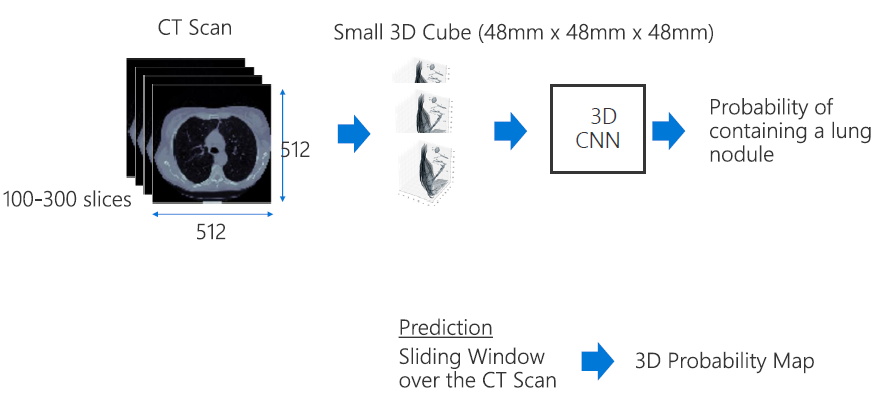
Normalize: Hounsfield Scale
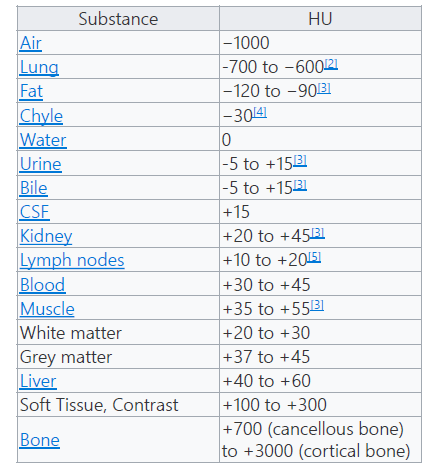
- Hounsfield Scale로
- 0~1로 정규화함
Hounsfield Scale이란?
Hounsfield 단위(HU)는 의료 CT 이미지의 그레이 스케일을 구성합니다. 4096개 값(12비트)의 검은색에서 흰색에 이르는 스케일로 그 범위는 -1024HU ~ 3071HU(0 또한 값에 포함됨)입니다. 이는 다음과 같이 정의됩니다.
-1024HU는 검은색이며 공기(폐 내부)를 나타냅니다. 0HU는 물(인체는 주로 물로 구성되어 있으므로 여기에서 피크가 큼)을 나타냅니다. 3071HU는 흰색이며 인체에서 가장 밀도가 높은 조직인 치아 에나멜을 나타냅니다. 다른 모든 조직은 이 스케일 내에 있습니다. 지방은 약 -100HU, 근육은 약 100HU이며 뼈 폭은 200HU(소주골/하악골)에서 약 2000HU(피질골)입니다.
Extracting Small 3D cube
- 전체 CT scan은 GPU memory에 너무크기에 작은 3D cube영상으로 데이터셋 처리를 해주어야한다!
- positive samples -> lung nodeule 주변
- negative sample
-랜덤으로
-lung nodule 피해서
3D-CNN
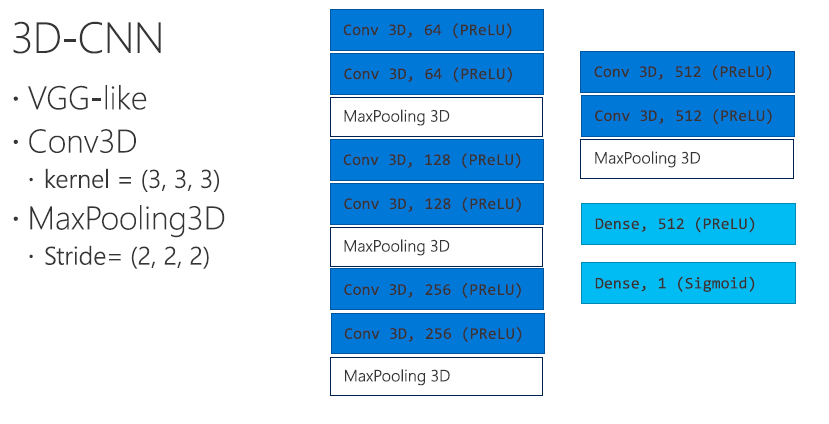
Training
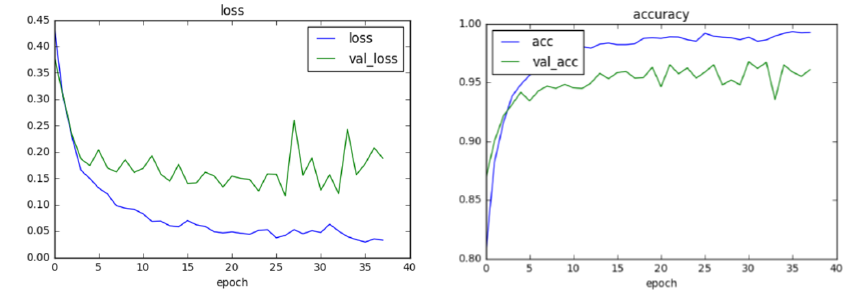
Training: Learning Curve
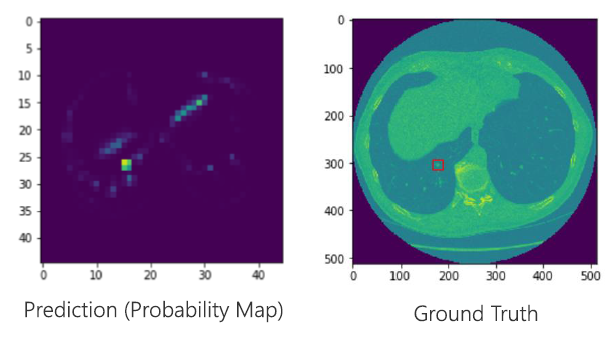
Prediction

Great tutorial on medical image processing using Microsoft's Deep Learning Framework CNTK! Exploring the intersection of technology and healthcare is always exciting. For those intrigued by the possibilities, this link on medical imaging software development provides valuable insights: https://www.cleveroad.com/blog/medical-imaging-software-development/