Code Review : 1st Place Code for AI competition for predicting lymphadenopathy in breast cancer
Code Review
Competition
Data Link
Data
Image Data + Tabular Data
Overview
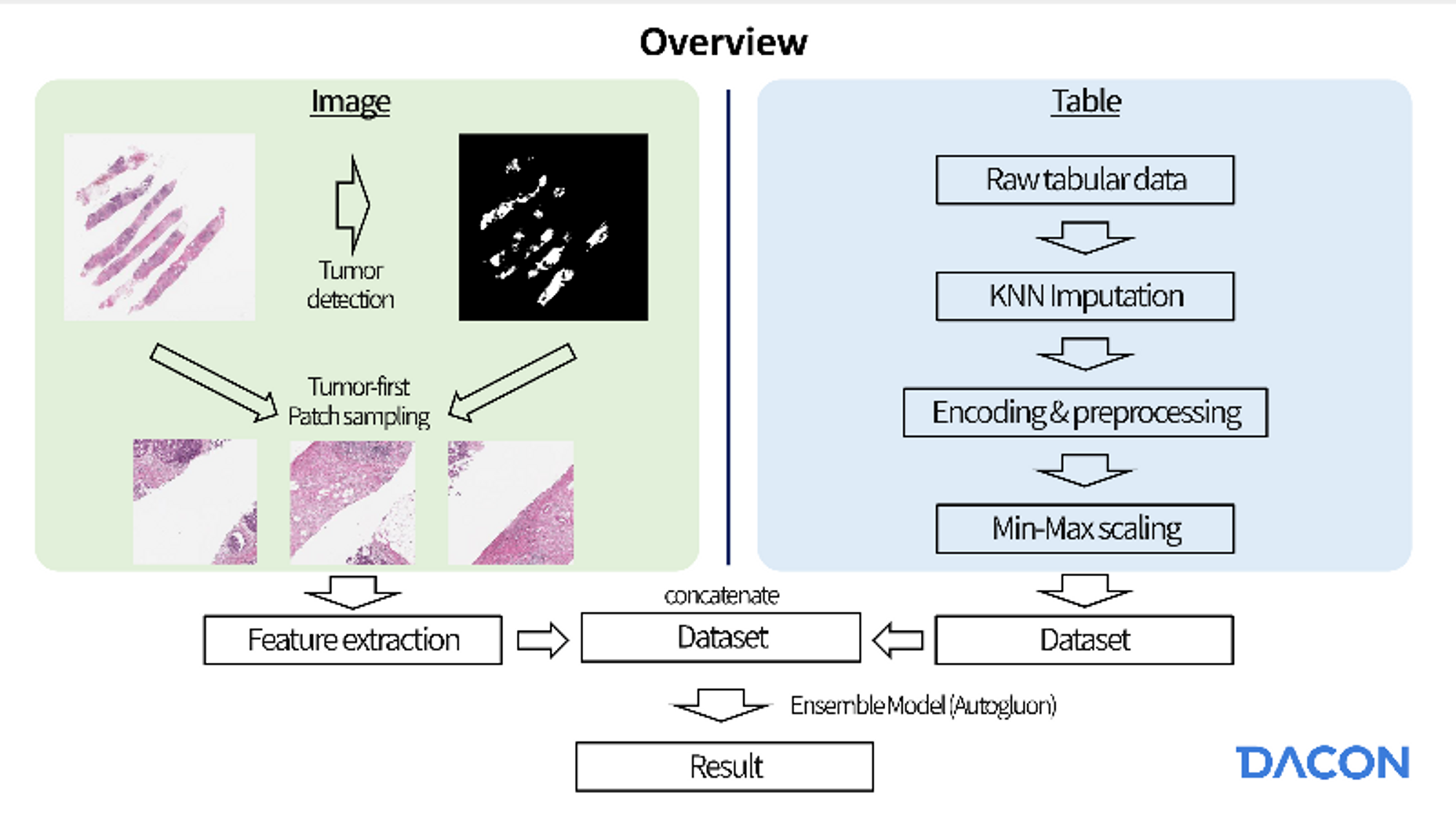
Image Data
IMG Split
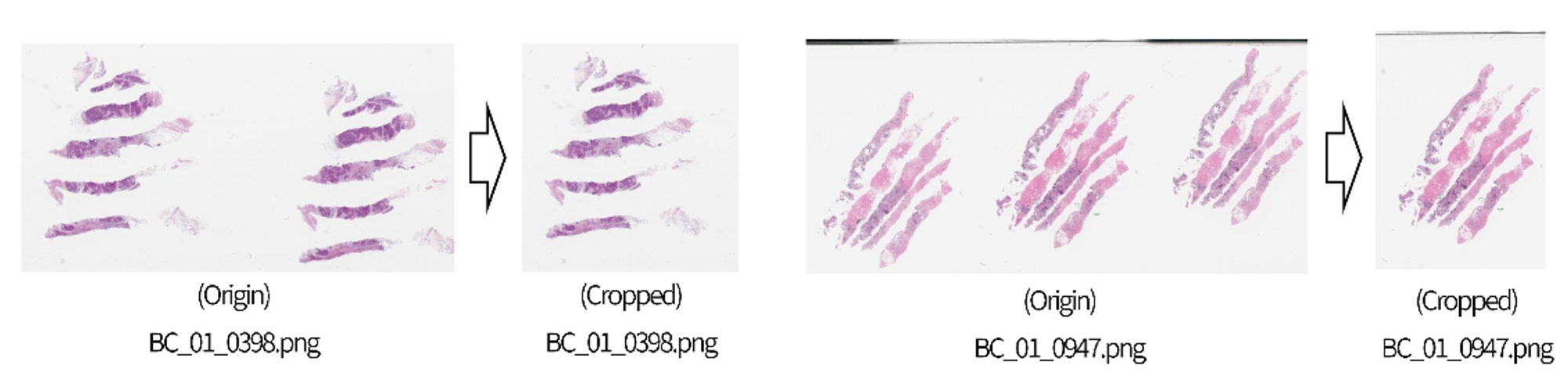
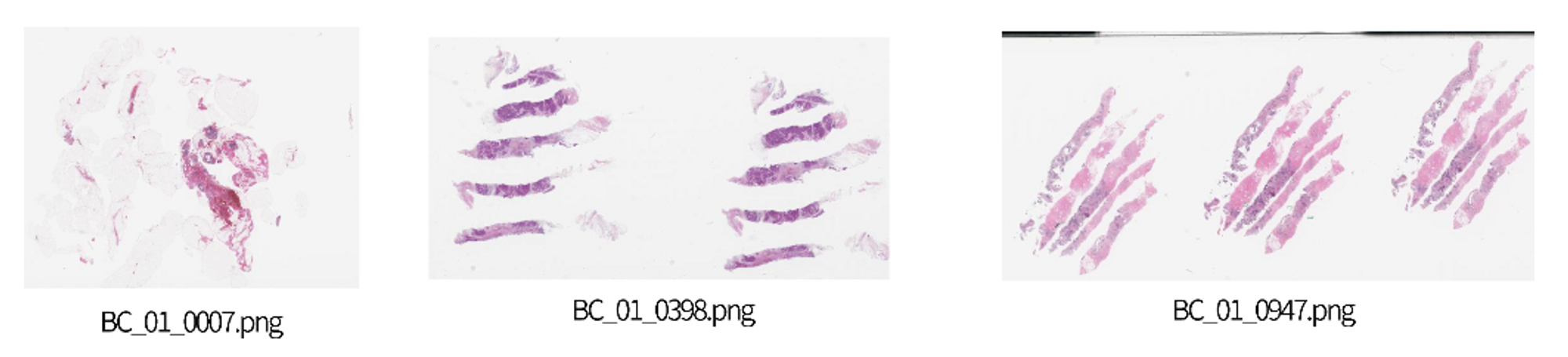
윗 사진과 같이 주어진 이미지 데이터에는 비슷한 조직이 1~4번 반복되어져 있다. 그래서 2등분선과 4등분선을 그어 background에 해당하는 pixel의 개수를 통해 하나의 조직만 학습에 사용될 수 있도록 한다.
count_overlapped_image → 2등분선과 4등분선을 그었을 때, background에 해당하는 pixel의 개수를 반환해주는 함수.
import os
import numpy as np
import pandas as pd
from PIL import Image
from tqdm.auto import tqdm
def count_overlapped_image(image, threshold):
img_size = image.size
image = np.array(image)
mid = image[:, img_size[0]//2] #중앙선
mid_cnt = sum(mid<threshold)
quad = image[:, img_size[0]//4]
quad_cnt = sum(quad<threshold)
return mid_cnt, quad_cnt
# 데이터 로딩
root_dir = "./submission_codes"
csv_dir = os.path.join(root_dir, "data", "clinical_data")
img_dir = os.path.join(root_dir, "data", "image_data")
train = pd.read_csv(os.path.join(csv_dir, 'train.csv'))
# 이미지 파일들의 이름을 가져옴. -> mask path가 있는 것만
train_image_names = []
for i in range(len(train)):
if train['mask_path'].iloc[i] != '-':
train_image_names.append(train['mask_path'].iloc[i][-14:])
각 이미지들이 조직이 몇번 반복된 형태인지 구분하는 방법:
1) 2등분선 기준 30 pixel 미만 존재할 경우 --> 이 경우는 중앙선에 대상이 없다는 것이니까 3번 반복이나 1번 반복은 아님.
1-1) 4등분선 기준 30 pixel 이하 존재하면 4번 반복으로 간주
1-2) 4등분선 기준 30 pixel 초과 존재하면 2번 반복으로 간주
2) 2등분선 기준 30 pixel 이상 존재할 경우,
2-1) 가로 길이와 세로 길이의 비율이 1.3을 넘어갈 경우, 3번 반복으로 간주
2-2) 가로 길이와 세로 길이의 비율이 1.3 이하일 경우, 1번 반복으로 간주
cnt_label = [] #cnt_label은 해당 이미지에서 조직이 몇번 반복되는지 저장.
for file_name in tqdm(train_image_names):
image = Image.open(os.path.join(img_dir, "train_imgs", file_name)).convert('L')
m, q = count_overlapped_image(image, 200)
if m<30:
if q>30:
label = 2
else:
label = 4
else:
if image.size[0]>image.size[1]*1.3:
label = 3
else:
label = 1
cnt_label.append([file_name, label]) annotation mask는 반복된 조직들 중 1개의 조직에만 존재하기 때문에, annotation mask가 존재하는 조직을 골라 crop한다.
cropped_imgs = []
cropped_masks = []
for i in tqdm(range(len(cnt_label)), total=len(cnt_label)):
image = Image.open(os.path.join(img_dir, "train_imgs", train_image_names[i])).convert('RGB') #PIL로 사진 가져오면 rgba로 불러오기 때문에 RGB로 컬러 변환해줘야함.
mask = Image.open(os.path.join(img_dir, "train_masks", train_image_names[i])).convert('L') #mask --> 이미지 내 특정 개체가 어디에 위치해있는지. GrayScale로 변환
img_ary = np.array(image)
mask_ary = np.array(mask)
img_arys = []
mask_arys = []
size = image.size
for j in range(cnt_label[i][1]):
img_arys.append(img_ary[:, j*size[0]//cnt_label[i][1]:(j+1)*size[0]//cnt_label[i][1], :]) #img array에 crop된거 추가
mask_arys.append(mask_ary[:, j*size[0]//cnt_label[i][1]:(j+1)*size[0]//cnt_label[i][1]]) #mask array에 img crop된거에 대한 mask 추가
target_cnt = [] #각 대상 별 0(검정색)의 개수. (crop된 상태니까)
for ary in mask_arys:
target_cnt.append(sum(sum(ary==0)))
idx = target_cnt.index(max(target_cnt))
#crop된 것들 중 가장 검정색이 많은 이미지를 idx로 설정. annotation이 된 대상 선택하는 것.
cropped_img = Image.fromarray(img_arys[idx])
cropped_mask = Image.fromarray(mask_arys[idx])
cropped_imgs.append(cropped_img)
cropped_masks.append(cropped_mask)
# cropped 이미지 저장
save_dir = os.path.join(img_dir, "train_cropped")
if not os.path.exists(save_dir):
os.makedirs(save_dir)
for i, img in enumerate(cropped_imgs):
img.save(os.path.join(save_dir, train_image_names[i]))
print(f'{os.path.join(save_dir, train_image_names[i])} saved.')
print(f'{len(cropped_imgs)} images saved.')
save_dir = os.path.join(img_dir, "mask_cropped")
if not os.path.exists(save_dir):
os.makedirs(save_dir)
for i, img in enumerate(cropped_masks):
img.save(os.path.join(save_dir, train_image_names[i]))
print(f'{os.path.join(save_dir, train_image_names[i])} saved.')
print(f'{len(cropped_masks)} images saved.')- 정리
- 데이터 로딩
- 2등분선, 4등분선을 그어 몇 번 해당 개체가 반복되는지 파악.
- annotation mask는 하나에만 존재하기 때문에 Greyscale로 변환하여 annotation mask 있는 것을 cropped_images, cropped_masks로 저장.
Custom Dataset
Otsu Threshold
- Threshold란?
Thresholding이란, 어떤 임계점(threshold)를 기준으로 두 가지 부류로 나누는 방법을 의미한다.
Binary Image, 검은색과 흰색으로만 표현된 이미지를 만드는 가장 대표적인 방법이다. - Otsu Threshold란?
오츠의 알고리즘은 임계값을 임의로 정해 픽셀을 두 부류로 나누고, 나뉜 두 부류의 명암 분포를 구하는 작업을 반복하여 모든 경우의 수에서 명암 분포가 가장 균일할 때의 임계값을 선택한다.
Class SegmentationInferenceDataset → backround가 제거된 이미지 patch들을 output. (mask 고려 x)
import torch
from torch.utils.data import Dataset
import os
import numpy as np
import pandas as pd
import cv2 as cv
from tqdm.auto import tqdm
from PIL import Image
class SegmentationInferenceDataset(Dataset):
def __init__(self, csv_dir, img_dir, target_value,
img_transform=None,
image_size=300,
stride=150,
file_name=None,
threshold=230,
binary_threshold=False):
self.target_value = target_value
self.csv = pd.read_csv(csv_dir)
self.file_name = file_name
self.img_transform = img_transform
self.patches = []
self.image_size = image_size
total = 0
cnt = 0
self.image = Image.open(os.path.join(img_dir, file_name)).convert('RGB')
gray_img = np.array(Image.open(os.path.join(img_dir, file_name)).convert('L')) #회색으로 이미지 불러오고 numpy array로 변환
if binary_threshold=='otsu':
_, otsu_threshold = cv.threshold(255-gray_img, -1, 255, cv.THRESH_BINARY | cv.THRESH_OTSU)
#경계값을 지정하지 않고 OTSU 사용. OTSU로 선택된 경계값은 _임.
otsu_threshold = 255 - gray_img > _
#경계값으로, pixel값이 threshold 이상인 값에 대해서는
#background로 간주해 0으로 설정. 이걸 하는 이유가 backround 찾아내려고.
elif binary_threshold:
otsu_threshold = gray_img < binary_threshold
else:
otsu_threshold = 1
if self.img_transform:
self.image = self.img_transform(self.image)
#transform이 있으면 해당 transfom 진행
for i in range(3):
self.image[i] = self.image[i] * otsu_threshold
#각 채널에 otsu threshold 값 구한 것을 적용 -> background는 검정색으로.
for row in range(0, self.image.shape[1]-image_size, stride):
#image.shape[1] = width, image_size는 패치 사이즈를 의미.
for col in range(0, self.image.shape[2]-image_size, stride):
#image.shape[2] = height, image.shape[0] = size
self.patches.append((row, col))
total += 1
print(f'{total} patches created.')
def __getitem__(self, idx):
r, c = self.patches[idx][0], self.patches[idx][1]
patch = self.image[:, r:r+self.image_size, c:c+self.image_size]
#patch 값을 불러와서 이미지로 만듬.
return patch
def __len__(self):
return len(self.patches)- SegmentationInferenceDataset Class 정리
Threshold(OTSU)로 background를 찾아서 검정색(0)으로 만들고, image_size 값에 맞게 패치로 자름.
그리고 해당 패치들을 데이터로 전달함.
class SegmentationDataset → bacground가 제거된 patch와 해당 patch들을 augmentation한 patch들 output. (mask 고려)
class SegmentationDataset(Dataset):
def __init__(self, csv_dir, img_dir, target_value,
img_transform=None,
mask_transform=None,
image_size=300,
stride=150,
mask_only=False,
file_names=None,
threshold=230,
cnt_threshold=10,
test=None,
binary_threshold=False):
self.target_value = target_value
self.csv = pd.read_csv(csv_dir)
if file_names:
self.img_file_names = file_names[0]
self.mask_file_names = file_names[1]
else:
if mask_only:
self.img_file_names = []
self.mask_file_names = []
for i in range(len(self.csv)):
if self.csv['mask_path'].iloc[i] != '-':
self.img_file_names.append(self.csv['img_path'].iloc[i])
self.mask_file_names.append(self.csv['mask_path'].iloc[i])
else:
self.img_file_names = list(self.csv['img_path'])
self.mask_file_names = list(self.csv['mask_path'])
self.img_transform = img_transform
self.mask_transform = mask_transform
self.patches = []
self.labels = []
self.label_patches = []
if test:
self.img_file_names = self.img_file_names[:test]
self.mask_file_names = self.mask_file_names[:test]
total = 0
cnt = 0
for idx, (img, msk) in tqdm(enumerate(zip(self.img_file_names, self.mask_file_names)), total=len(self.img_file_names), desc='extracting patch...'):
self.image = Image.open(os.path.join(img_dir, img)).convert('RGB')
gray_img = np.array(Image.open(os.path.join(img_dir, img)).convert('L'))
if binary_threshold=='ostu': #threshold 계산해서 background black.
_, otsu_threshold = cv.threshold(255-gray_img, -1, 255, cv.THRESH_BINARY | cv.THRESH_OTSU)
otsu_threshold = 255 - gray_img > _
elif binary_threshold:
otsu_threshold = gray_img < binary_threshold
else:
otsu_threshold = 1
if self.img_transform: #transfrom 할거 있으면 하기
self.image = self.img_transform(self.image)
gray_img = self.img_transform(gray_img)
self.mask = Image.open(os.path.join(img_dir, msk)).convert('L') #mask 불러오기
if self.mask_transform: #mask transform 있으면 하기
self.mask = self.mask_transform(self.mask)
for i in range(3): #이미지의 각 채널 threshold로 backround 제거.
self.image[i] = self.image[i] * otsu_threshold
gray_img[0] = gray_img[0] * otsu_threshold #gray도 threshold로 background 제거. gray는 채널이 하나니까.
for row in range(0, self.image.shape[1]-image_size, stride):
for col in range(0, self.image.shape[2]-image_size, stride):
patch = self.image[:, row:row+image_size, col:col+image_size] #이미지를 패치로 자르기.
gray_scale = gray_img[:, row:row+image_size, col:col+image_size] #그레이 이미지를 패치로 자르기.
total += 1
if binary_threshold and (gray_scale>threshold/255).sum()<=cnt_threshold: #여기서 threshold는 우리가 input으로 지정한 threshold. 255로 나누는 이유는 scaling때문
cnt += 1 #binary threshold가 정해져 있고, 그 경계값을 넘지 못한다면, 해당 패치는 pass. cnt는 패스하는 패치 count하는 것.
continue
elif not binary_threshold and (gray_scale<threshold/255).sum()<=cnt_threshold:
cnt += 1 #binary threshold가 없으면, gray_scale<threshold/255이면 해당 패치는 pass.
continue
self.patches.append(patch)
label_patch = self.mask[:, row:row+image_size, col:col+image_size]==0 #이렇게 해서 background가 어딘지 확인
label_patch = label_patch.float() #True False를 0과 1로 바꿈.
self.label_patches.append(label_patch)
if (self.mask[:, row:row+image_size, col:col+image_size]==0).sum().item()>0:
self.labels.append(1)
else:
self.labels.append(0)
print(f'{total} patches created, {cnt} patches were passed. (under {threshold})')
#이렇게 해서 backround가 제거된 패치를 넣는다. 패치에 background로 판별되는 픽셀이 너무 많으면 해당 패치는 pass한다.
def data_augmentation(self, methods, label_tranform=False, label_only=False): #augmentation 진행하고 extend로 patch와 label 늘림.
self.augmented_patches = []
self.augemented_labels = []
self.augmented_label_patches = []
for method, factor in methods:
for idx, patch in tqdm(enumerate(self.patches), total=len(self.patches)):
if label_only:
if self.labels[idx] != label_only:
continue
if factor:
self.augmented_patches.append(method(patch, factor))
else:
self.augmented_patches.append(method(patch))
if label_tranform:
self.augmented_label_patches.append(method(self.label_patches[idx]))
else:
self.augmented_label_patches.append(self.label_patches[idx])
self.augemented_labels.append(self.labels[idx])
self.patches.extend(self.augmented_patches)
self.labels.extend(self.augemented_labels)
self.label_patches.extend(self.augmented_label_patches)
print(f'{len(self.augmented_patches)} is added. total dataset size is {len(self.patches)}.')
def __getitem__(self, idx):
return self.patches[idx], self.labels[idx], self.label_patches[idx]
def __len__(self):
return len(self.patches)- SegmentationDataset Class 정리
Threshold(OTSU)로 image와 mask의 background를 찾아서 검정색(0)으로 만들고, image_size 값에 맞게 패치로 자름.
그리고 해당 패치들을 augmentation 진행함.
patch, label, 그리고 label patch를 output으로.
class MILDataset → background가 제거된 patch들을 tumor 임계값에 따라 tumor, nt_tumor으로 나눈다. 원하는 데이터 수에 맞게 데이터를 제공한다.
class MILDataset(Dataset):
def __init__(self, csv_dir, img_dir, mask_dir, target_value,
img_transform=None,
mask_transform=None,
image_size=300,
stride=150,
threshold=230,
cnt_threshold=10,
tumor_threshold=100,
test=None,
binary_threshold=False,
max_width=3000,
n_patches=100,
test_dataset=False):
self.target_value = target_value
self.csv = pd.read_csv(csv_dir)
if test_dataset:
self.img_file_names = [i[12:] for i in self.csv['img_path']]
else:
self.img_file_names = [i[13:] for i in self.csv['img_path']]
self.test_dataset = test_dataset
if test_dataset==False:
self.labels = [i for i in self.csv['N_category']]
else:
self.labels = None
self.image_size = image_size
self.img_transform = img_transform
self.mask_transform = mask_transform
self.patches = []
if test:
self.img_file_names = self.img_file_names[:test]
total = 0
cnt = 0
for idx, img in tqdm(enumerate(self.img_file_names), total=len(self.img_file_names), desc='extracting patch...'):
self.image = Image.open(os.path.join(img_dir, img)).convert('RGB') #이미지 읽기
gray_img = np.array(Image.open(os.path.join(img_dir, img)).convert('L')) #이미지 회색으로 읽기
mask = Image.open(os.path.join(mask_dir, img)).convert('L') #마스크 읽기
if binary_threshold=='ostu': #threshold 정하기
_, otsu_threshold = cv.threshold(255-gray_img, -1, 255, cv.THRESH_BINARY | cv.THRESH_OTSU)
otsu_threshold = 255 - gray_img > _
elif binary_threshold:
otsu_threshold = gray_img < binary_threshold
else:
otsu_threshold = 1
if self.img_transform:
self.image = self.img_transform(self.image) #transform 할거 있으면 하기
gray_img = self.mask_transform(gray_img) #transform 할거 있으면 하기
if self.mask_transform:
self.mask = self.mask_transform(mask) #mask도 transform 할거 있으면 하기
for i in range(3): #background 지우고, 마스크와 이미지 패치로 잘라서 저장.
self.image[i] = self.image[i] * otsu_threshold
gray_img[0] = gray_img[0] * otsu_threshold
t_pat = []
nt_pat = []
for row in range(0, self.image.shape[1]-image_size, stride): #tumor threshold에 따라 tumor patch와 ntumor patch로 나누기. pat에 image, gray_img, mask까지 저장.
for col in range(0, min(self.image.shape[2]-image_size, max_width), stride):
if (self.mask[:, row:row+image_size, col:col+image_size]).sum()<tumor_threshold:
t_pat.append((self.image[:, row:row+image_size, col:col+image_size].unsqueeze(0), (gray_img[:, row:row+image_size, col:col+image_size]>threshold/255).sum(), (self.mask[:, row:row+image_size, col:col+image_size]).sum()))
else:
nt_pat.append((self.image[:, row:row+image_size, col:col+image_size].unsqueeze(0), (gray_img[:, row:row+image_size, col:col+image_size]>threshold/255).sum(), (self.mask[:, row:row+image_size, col:col+image_size]).sum()))
t_pat.sort(key=lambda x: x[1]) #gray_img에 따라 순서 정렬
nt_pat.sort(key=lambda x: x[1]) #위와 동일
t_pat = [i[0] for i in t_pat] #t_pat에 이미지 데이터만 담기.
nt_pat = [i[0] for i in nt_pat] #nt_pat에 이미지 데이터만 담기.
if (len(t_pat)+len(nt_pat))<n_patches: #생성한 패치보다 원하는 패치 수가 더 많다면, 생성한 패치 전부 다 랜덤하게 넣기.
patch = t_pat + nt_pat
indices = [i for i in range(len(patch))]
sampled_indices = np.random.choice(indices, n_patches-len(patch), replace=True)
for p in sampled_indices:
patch.append(patch[p])
elif len(t_pat)>n_patches: #생성한 t_pat 패치가 원하는 패치 수가 더 크다면, 원하는 패치만큼 t_pat에서 가져온다.
# indices = [i for i in range(len(t_pat))]
# sampled_indices = np.random.choice(indices, n_patches, replace=False)
patch = t_pat[:n_patches]
else: #나머지 경우, -> 1)t_pat+nt_pat이 n_patches보다 작거나 같고, t_pat이 n_patches보다 작거나 같은 경우에는 -> 최대한 t_pat을 넣고, 나머지 남은 것을 nt_pat으로 채운다.
# indices = [i for i in range(len(nt_pat))]
# sampled_indices = np.random.choice(indices, n_patches-len(t_pat), replace=False)
patch = t_pat + nt_pat[:n_patches-len(t_pat)]
self.patches.append(torch.cat(patch))
print(f'{total} patches created, {cnt} patches were passed. (under {threshold})')
def __getitem__(self, idx):
if self.test_dataset:
return self.patches[idx]
else:
return self.patches[idx], self.labels[idx]
def __len__(self):
return len(self.patches)- 원하는 크기의 데이터셋 만들기
- 생성된 패치의 수보다 원하는 데이터의 크기가 더 많을 경우:
생성된 패치를 모두 랜덤하게 가져온다. - 생성된 t_pat 패치의 수가 원하는 데이터의 크기보다 더 많을 경우:
원하는만큼 t_patch에서 가져온다. - 이외의 경우(1.t_pat + nt_pat이 n_patches보다 작거나 같은 경우, 2. t_pat이 n_patches보다 작거나 같은 경우):
최대한 t_pat을 가져오고, 남은 만큼 nt_pat을 가져온다.
- MILDataset Class 정리
Threshold(OTSU)로 image와 mask의 background를 찾아서 검정색(0)으로 만들고, image_size 값에 맞게 패치로 자름.
해당 patch들을 tumor 임계값으로 t_pat과 nt_pat으로 구분하고, 각 patch에 mage, gray)img, label을 저장한다.
원하는 크기의 데이터셋만큼 랜덤하게 output으로.
Models
EfficientNet
Image Classification Task에 대해서 2019년 이전보다 훨씬 적은 파라미터 수로 더욱 좋은 성능을 냄.
기존 연구들에서는 ConvNet의 성능을 올리기 위해 scaling up 시도를 많이 했다.
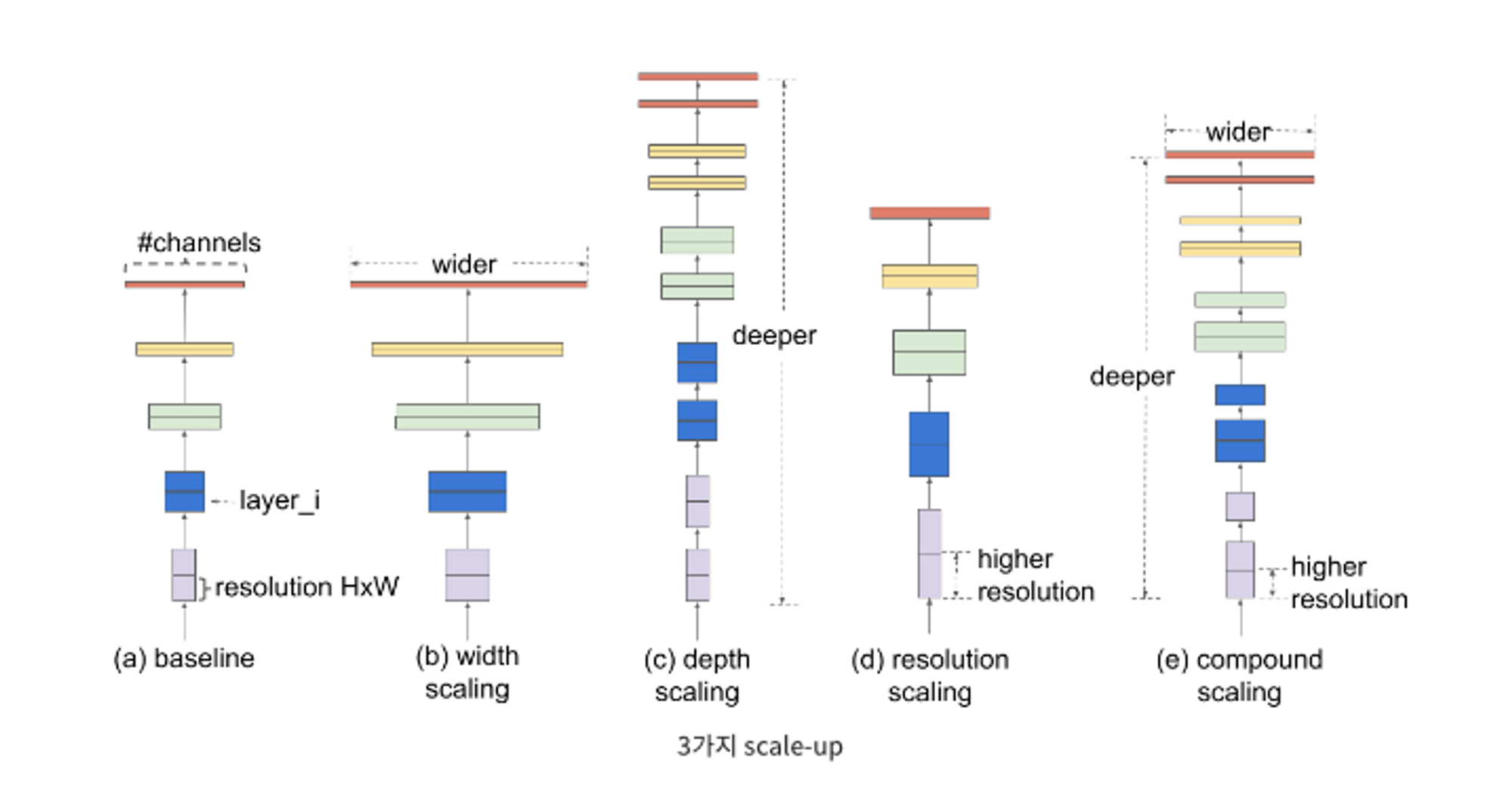
Scale-up 방법은 3가지가 있다.
1) 망의 depth를 늘리는 것 → Layer의 갯수를 늘리는 것
2) Channel Width를 늘리는 것 → filter의 갯수(channel의 갯수)를 늘리는 것.
3) 입력 이미지의 해상도를 올리는 것
EfficientNet은 세가지 방법에 대한 최적의 조합을 AutoML을 통해 찾은 모델이다. 3가지를 효율적으로 조절할 수 있는 Compound Scaling 방법을 제안하는 모델이다.
Unet
인코딩 단계에서 차원 축소를 거치면서 이미지 객체에 대한 자세한 위치 정보를 잃게 되고, 디코딩 단계에서도 저차원의 정보만을 이용하기 때문에 위치 정보 손실을 해결하지 못하는 문제가 발생한다. Unet은 이러한 문제를 Skip Connection으로 해결한 모델이다. 인코더의 레이어와 디코더의 레이어를 직접 연결(Skip Connection)하여 저차원 뿐만 아니라 고차원 정보도 이용하여 이미지의 특징을 추출함과 동시에 정확한 위치 파악도 가능하게 한 모델.
신경망 구조를 스킵 연결을 평행하게 두고, 가운데를 기준으로 좌우가 대칭이 되도록 레이어를 배치했다.
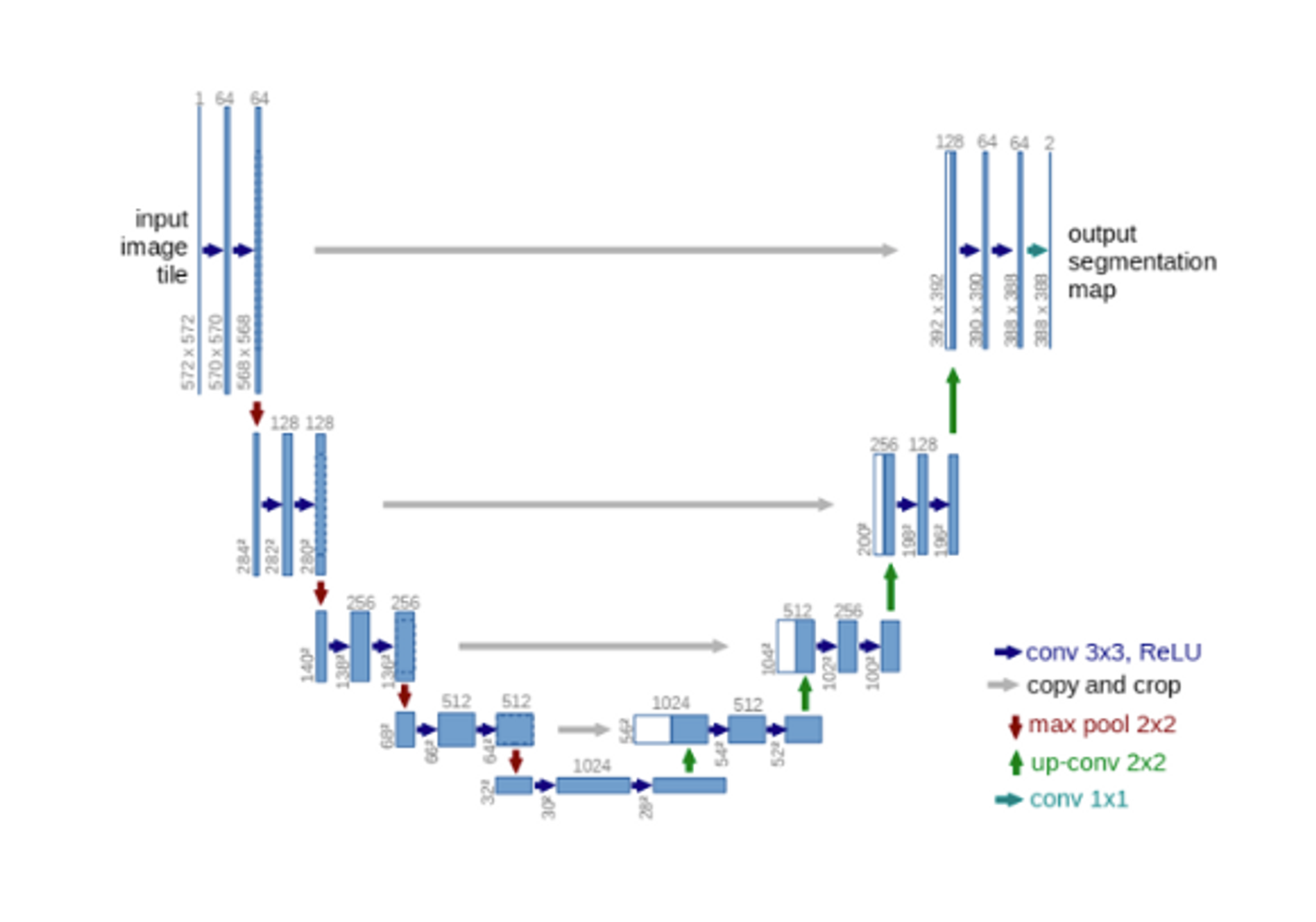
import torch
import torch.nn as nn
import torch.nn.functional as F
from torchvision.models import efficientnet_b3, EfficientNet_B3_Weights
import pretrainedmodels
import segmentation_models_pytorch as smp
from sklearn.decomposition import PCA
from sklearn.manifold import TSNE
class EfficientNetB3(nn.Module):
def __init__(self, resnet=False): #resnet 또는 efficientnet 사용
super(EfficientNetB3, self).__init__()
if resnet:
self.model = pretrainedmodels.__dict__['inceptionresnetv2'](num_classes=1000, pretrained='imagenet')
else:
self.model = efficientnet_b3(pretrained=True, weights=EfficientNet_B3_Weights.DEFAULT)
def forward(self, x):
out = F.softmax(self.model(x)) #Pretrained model 들어간 이후 Fully Connected Layer
out_sum = torch.sum(out, dim=0)
out_sum = out_sum.unsqueeze(0)
return out_sum
class SegmentationUnetModel(nn.Module): #Unet 활용하여 annotation mask 생성.
def __init__(self, encoder):
super(SegmentationUnetModel, self).__init__()
self.model = smp.Unet(
encoder_name=encoder,
encoder_weights='imagenet',
in_channels=3,
classes=1
)
def forward(self, x):
out = self.model(x)
return outSegmentation Training
import torch
import torch.nn as nn
from torch import optim as optim
from torch.utils.data import DataLoader
from torchvision.transforms import transforms
from torchvision.transforms.functional import hflip, vflip, adjust_hue, adjust_brightness, adjust_contrast, adjust_saturation, rotate
import os
import numpy as np
import pandas as pd
import custom_dataset
import utils
import models
import ssl
from tqdm.auto import tqdm
# 2가지 모델로 pipeline을 구성했음.
# 1) timm-efficientnet-b3 encoder based Unet, 2) inceptionresnetv2 encoder based Unet
# 각 모델로 학습을 시키기 위해서 args에 'encoder'를 바꾸면 됨.
# 1) timm-efficientnet-b3
# encoder -> timm-efficientnet-b3
# 2) inceptionresnetv2
# encoder -> inceptionresnetv2
# 모델 다운로드 과정에서 발생하는 오류 해결
ssl._create_default_https_context = ssl._create_unverified_context
# hyperparameters
args = {
'batch_size' : 16,
'learning_rate' : 1e-3,
'target_value' : 0,
'cnt_threshold' : 10000,
'binary_threshold' : 240,
'patch_size' : 320,
'stride' : 160,
'random_seed' : 10,
'test_size' : 0.15,
'out_dim' : 2,
'n_epochs' : 10,
'device' : 'cuda' if torch.cuda.is_available() else 'cpu',
'test' : None,
'encoder' : 'timm-efficientnet-b3',
'save' : True
}
# random seed 세팅
utils.set_seeds(seed=args['random_seed'])
# 경로 설정
root_dir = "./submission_codes"
csv_dir = os.path.join(root_dir, "data", "clinical_data")
img_dir = os.path.join(root_dir, "data", "image_data")
model_dir = os.path.join(root_dir, "data", "saved_models")
file_names = os.listdir(os.path.join(img_dir, "train_cropped"))
file_names.sort()
# train/validation dataset에 들어갈 이미지 random choice
split_indices = np.random.choice(len(file_names),
int(len(file_names)*args['test_size']),
replace=False)
split_indices.sort()
train_file_names = []
val_file_names = []
for i in range(len(file_names)):
if i in split_indices:
val_file_names.append(file_names[i])
else:
train_file_names.append(file_names[i])class SegmentationDataset으로 train_dataset과 val_dataset을 생성한다.
# dataset 로딩
#torchvision의 transforms로 Tensor으로 변환 및 normalize
img_transform = transforms.Compose([transforms.ToTensor(),
transforms.Normalize(0.5, 0.5)])
mask_transform = transforms.ToTensor()
train_dataset = custom_dataset.SegmentationDataset( #backround가 제거된 patch들을 output으로 줌.
csv_dir=os.path.join(csv_dir, "train.csv"),
img_dir=img_dir,
target_value=args['target_value'],
img_transform=img_transform,
mask_transform=mask_transform,
mask_only=True,
file_names=[[os.path.join("train_cropped", i) for i in train_file_names],
[os.path.join("mask_cropped", i) for i in train_file_names]],
threshold=1,
cnt_threshold=args['cnt_threshold'],
test=args['test'],
binary_threshold=args['binary_threshold'],
image_size=args['patch_size'],
stride=args['stride']
)
val_dataset = custom_dataset.SegmentationDataset( #background가 제거된 patch들을 output으로 줌.
csv_dir=os.path.join(csv_dir, "train.csv"),
img_dir=img_dir,
target_value=args['target_value'],
img_transform=img_transform,
mask_transform=mask_transform,
mask_only=True,
file_names=[[os.path.join("train_cropped", i) for i in val_file_names],
[os.path.join("mask_cropped", i) for i in val_file_names]],
threshold=1,
cnt_threshold=args['cnt_threshold'],
test=args['test'],
binary_threshold=args['binary_threshold'],
image_size=args['patch_size'],
stride=args['stride']
)hue data augmentation과 horizontal flip을 적용
- hue augmentation
Hue augmentation randomly alters the color channels of an input image, causing a model to consider alternative color schemes for objects and scenes in input images. This technique is useful to ensure a model is not memorizing a given object or scene's colors. While output image colors can appear odd, even bizarre, to human interpretation, hue augmentation helps a model consider the edges and shape of objects rather than only the colors.
- Background가 제거된 patch와 hue augmentation까지 진행된 patch
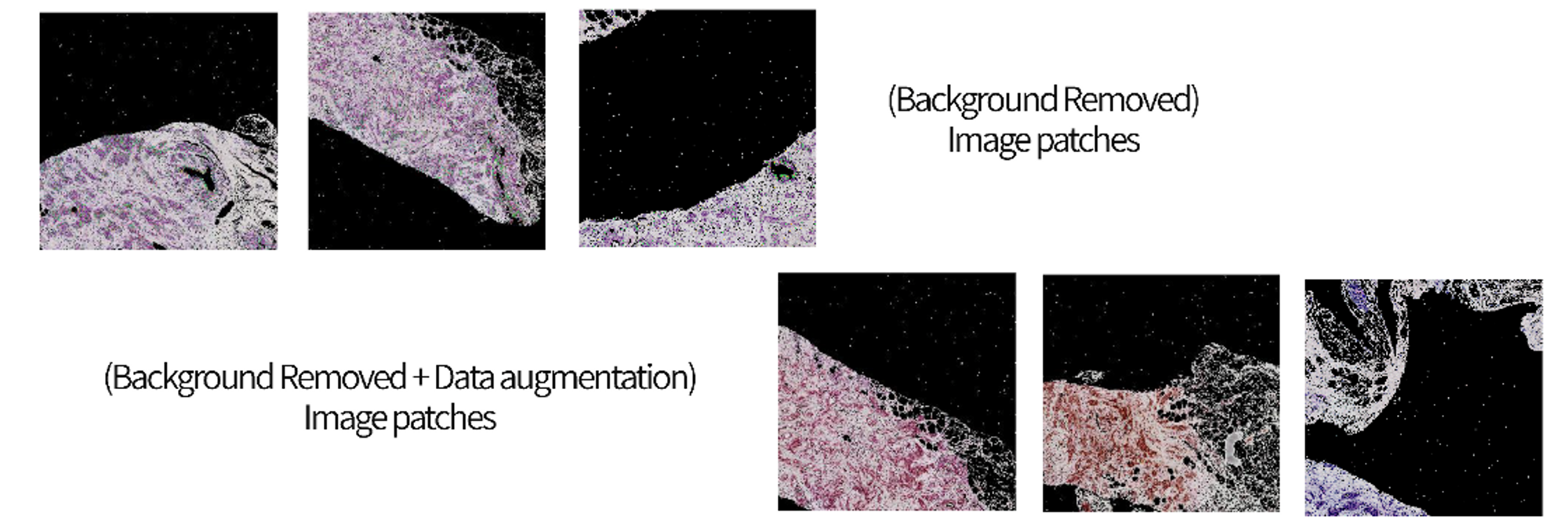
# data augmentation
DA_methods = [(adjust_hue, 0.1), (adjust_hue, -0.1), (adjust_hue, 0.2), (adjust_hue, -0.2)] #alters the color channels of an input image,
#causing a model to consider alternative color schemes
#for objects and scenes in input images
train_dataset.data_augmentation(DA_methods)
DA_methods = [(hflip, None)] #horizontal flip
train_dataset.data_augmentation(DA_methods, label_tranform=True, label_only=1)
# dataloader 생성
train_dataloader = DataLoader(train_dataset,
batch_size=args['batch_size'],
shuffle=True,
drop_last=True)
val_dataloader = DataLoader(val_dataset,
batch_size=args['batch_size'],
shuffle=False,
drop_last=False)# 모델 및 optimizer, loss function 정의
model = models.SegmentationUnetModel(args['encoder']).to(args['device'])
optimizer = optim.Adam(model.parameters(), lr=args['learning_rate'])
scheduler = optim.lr_scheduler.CosineAnnealingLR(optimizer, T_max=100, eta_min=0)
criterion = utils.dice_loss
for epoch in range(args['n_epochs']):
model.train()
total_loss = 0
n_iter = 0
for batch_id, (x, y, y_patch) in tqdm(enumerate(train_dataloader), total=len(train_dataloader), desc=f'{epoch+1} training...'):
model.train()
x = x.to(args['device'])
y = y.to(args['device'])
y_patch = y_patch.to(args['device'])
preds = model(x)
loss = criterion(preds, y_patch)
optimizer.zero_grad()
loss.backward()
optimizer.step()
scheduler.step()
total_loss += loss
n_iter += 1
if (batch_id+1)%(len(train_dataloader)//3)==0:
with torch.no_grad():
model.eval()
union, intersection, wrong, miss = 0, 0, 0, 0
for x, y, y_patch in val_dataloader:
x = x.to(args['device'])
y = y.to(args['device'])
y_patch = y_patch.to(args['device'])
pred = model(x) > 0.5
u, i, w, m = utils.segmentation_metric(pred, y_patch)
union += u
intersection += i
wrong += w
miss += m
print(f'correct : {intersection/union}, wrong prediction : {wrong/union}, missed : {miss/union}')
save_dir = os.path.join(model_dir, args['encoder'])
if not os.path.exists(save_dir):
os.makedirs(save_dir)
if args['save']:
torch.save(model, os.path.join(save_dir, f"model_{epoch+1}_{(batch_id+1)//(len(train_dataloader)//3)}.pt"))
print(f'train loss : {total_loss/n_iter}')Annotation Mask Generating
- Mask Image
Segmentation을 수행할 때 주로 사용된다. Mask는 원본 이미지에서 사용자가 관심이 있는 영역인 ROI를 255 픽셀값으로 채우고, 그 이외의 영역은 0픽셀값으로 채운다. 이렇게 하여 흑백 이미지, 즉 Binary Image를 얻는다.

- annotations to semantic segmentation mask images
For many tasks that process annotation data like training machine learning algorithms or measuring inter observer agreement a mask image representation where pixel values encode ground truth information is more useful.
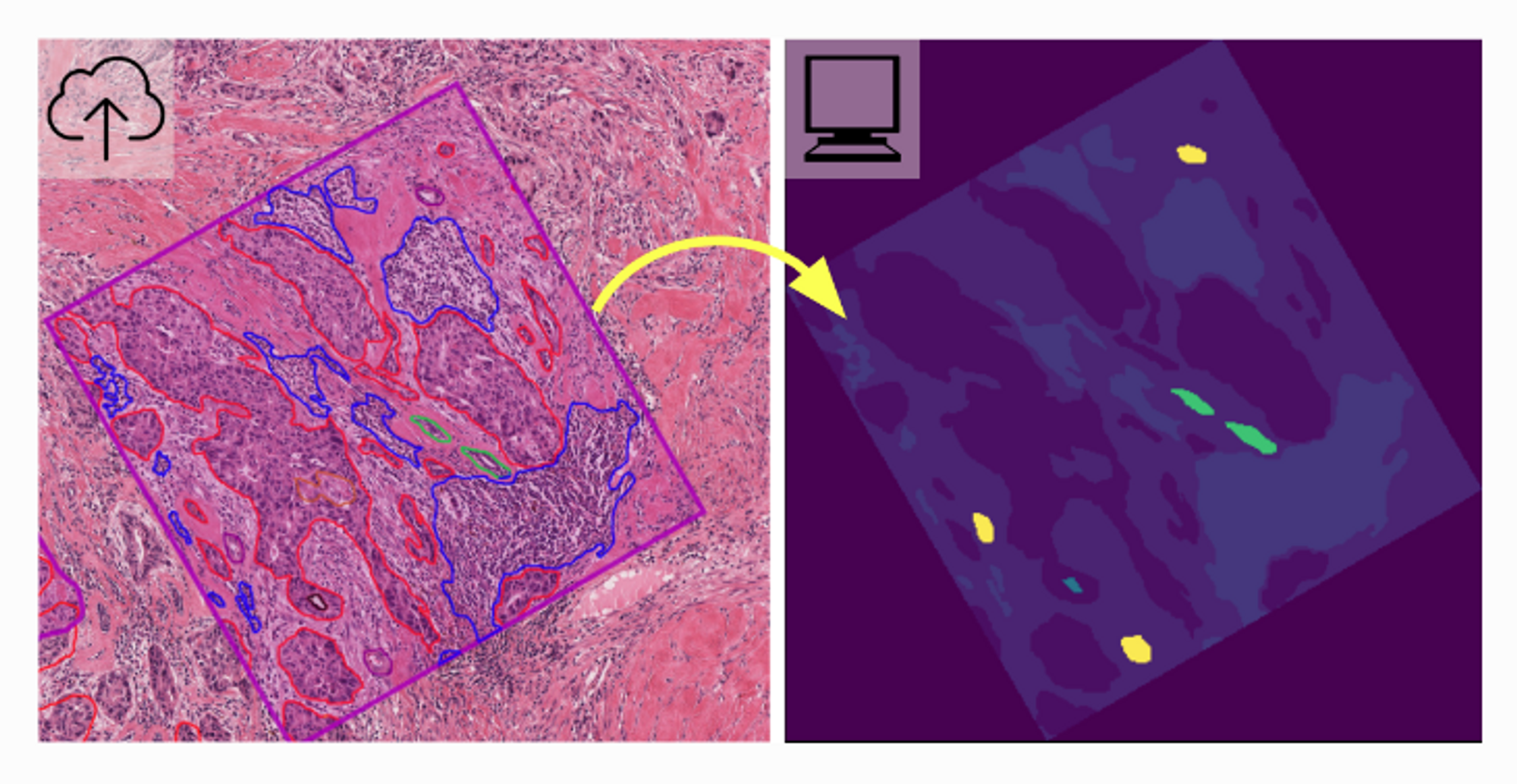
- Annotation을 mask image로 변환시킨 것

import torch
import torch.nn as nn
from torch import optim as optim
from torch.utils.data import DataLoader
from torchvision.transforms import transforms
import os
import numpy as np
import pandas as pd
import custom_dataset
import utils
import models
from tqdm.auto import tqdm
from PIL import Image
import warnings
warnings.filterwarnings("ignore")
# train dataset과 test dataset 모두에서 annotation mask를 만들어야 함.
# train dataset일 경우, inference_dataset variable을 "train",
# test dataset일 경우, inference_dataset variable을 "test"로 세팅하면 됨.
# 실험에 활용된 annotation mask는 computer resource 이슈로 inference는 100~300 정도로 끊어 생성했음.
inference_dataset = "test"
if inference_dataset == "train":
img_dir_name = "train_imgs"
slicing = 13
save_dir_name = "train_generated_mask"
csv_name = "train.csv"
elif inference_dataset == 'test':
img_dir_name = "test_imgs"
slicing = 12
save_dir_name = "test_generated_mask"
csv_name = "test.csv"
else:
raise ValueError('Wrong dataset variable!')
args = {
'batch_size' : 64,
'learning_rate' : 1e-3,
'target_value' : 0,
'cnt_threshold' : -1,
'binary_threshold' : 240,
'patch_size' : 320,
'stride' : 50,
'random_seed' : 10,
'test_size' : 0.15,
'n_epochs' : 10,
'device' : 'cuda' if torch.cuda.is_available() else 'cpu',
'encoder' : 'timm-efficientnet-b3',
}
# random seed 세팅
utils.set_seeds(seed=args['random_seed'])
# 경로 설정
root_dir = "./submission_codes"
csv_dir = os.path.join(root_dir, "data", "clinical_data")
# train set과 test set에 대하여 모두 inference를 진행해야 함.
img_dir = os.path.join(root_dir, "data", "image_data", img_dir_name)
save_dir = os.path.join(root_dir, "data", "image_data", save_dir_name)
if not os.path.exists(save_dir):
os.makedirs(save_dir)
train = pd.read_csv(os.path.join(csv_dir, csv_name))
img_path = [i[slicing:] for i in train['img_path']]
# model 로딩
model1 = torch.load(os.path.join(root_dir, f"/data/saved_models/timm-efficientnet-b3/model_6_3.pt").to(args['device']))
model2 = torch.load(os.path.join(root_dir, f"/data/saved_models/inceptionresnetv2/model_6_2.pt").to(args['device']))
model1.eval()
model2.eval()
img_transform = transforms.Compose([transforms.ToTensor(),
transforms.Normalize(0.5, 0.5)])
mask_transform = transforms.ToTensor()
for img_idx, file_name in enumerate(img_path):
dataset = custom_dataset.SegmentationInferenceDataset(
csv_dir=os.path.join(csv_dir, csv_name),
img_dir=img_dir,
target_value=args['target_value'],
img_transform=img_transform,
file_name=file_name,
binary_threshold=args['binary_threshold'],
image_size=args['patch_size'],
stride=args['stride']
)
dataloader = DataLoader(dataset,
batch_size=args['batch_size'],
shuffle=False,
drop_last=False)
preds = []
with torch.no_grad():
for x in tqdm(dataloader, desc=f'{img_idx+1} inference...', total=len(dataloader)):
x = x.to(args['device'])
pred1 = nn.Sigmoid()(model1(x))
pred2 = nn.Sigmoid()(model2(x))
pred = pred1 / 2 + pred2 / 2
preds.append(pred.cpu())
preds = torch.cat(preds)
col = (dataset.image.shape[1]-args['patch_size'])//args['stride'] + 1
row = (dataset.image.shape[2]-args['patch_size'])//args['stride'] + 1
new_img = torch.zeros(dataset.image.shape[1:])
cnt = torch.zeros(dataset.image.shape[1:])
cnt += 1e-4
idx = 0
for r in range(0, dataset.image.shape[1]-args['patch_size'], args['stride']):
for c in range(0, dataset.image.shape[2]-args['patch_size'], args['stride']):
new_img[r:r+args['patch_size'], c:c+args['patch_size']] += preds[idx].squeeze(0)
cnt[r:r+args['patch_size'], c:c+args['patch_size']] += 1
idx += 1
new_img /= cnt
new_img = (new_img > 0.5) #Binary
new_img = np.array(new_img)
image = Image.fromarray(new_img)
image.save(os.path.join(save_dir, file_name)) #train_generated_mask 또는 test_generated_mask에 저장됨
print(os.path.join(save_dir, file_name))Image Data - Feature Extraction
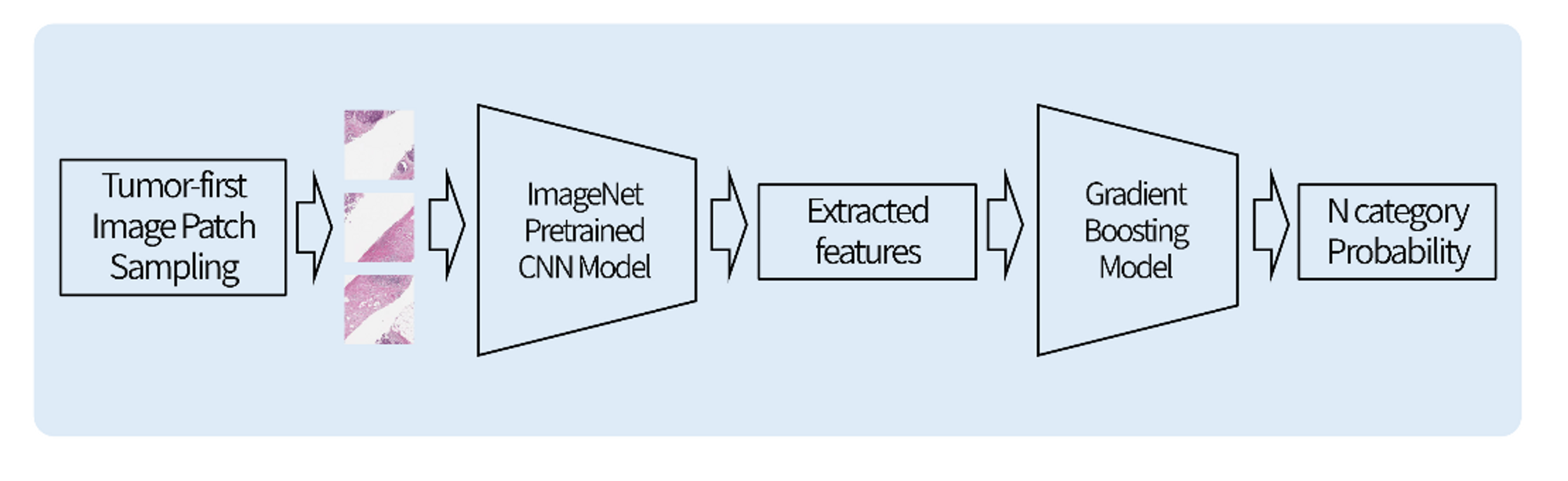
Image Feature Extraction
class MILDataset으로 데이터셋을 생성한다.
모델을 통과시켜 얻은 hidden_feature를 dataframe으로 저장한다.
import torch
import torch.nn as nn
import torch.nn.functional as F
from torch import optim as optim
from torchvision.transforms import transforms
import os
import numpy as np
import pandas as pd
import custom_dataset
import utils
import models
from tqdm.auto import tqdm
# n_patch는 (96, 120)을 모두 활용했음. 아래 n_patches variable을 수정하면 됨.
# inference dataset을 train, test로 세팅하여 코드 실행.
inference_dataset = "test"
n_patches = 96
if inference_dataset == 'train':
test_dataset = False
csv_name = "train.csv"
img_name = "train_imgs"
mask_name = "train_generated_mask"
elif inference_dataset == 'test':
test_dataset = True
csv_name = "test.csv"
img_name = "test_imgs"
mask_name = "test_generated_mask"
else:
raise ValueError('Wrong dataset variable!')
utils.set_seeds(seed=42)
args = {
'target_value' : 0,
'binary_threshold' : 240,
'tumor_threshold' : 3000,
'patch_size' : 300,
'stride' : 150,
'device' : 'cuda' if torch.cuda.is_available() else 'cpu',
'test' : 10,
'save' : True,
'max_width' : 3500,
'n_patches' : n_patches,
}
# 경로 설정
root_dir = "./submission_codes"
data_dir = os.path.join(root_dir, "data")
csv_dir = os.path.join(data_dir, "clinical_data")
img_dir = os.path.join(data_dir, "image_data")
dataset = custom_dataset.MILDataset(
csv_dir = os.path.join(csv_dir, csv_name),
img_dir = os.path.join(img_dir, img_name),
mask_dir = os.path.join(img_dir, mask_name),
target_value=args['target_value'],
img_transform=transforms.Compose([transforms.ToTensor(),
transforms.Normalize(0.5, 0.5)]),
mask_transform=transforms.ToTensor(),
image_size=args['patch_size'],
stride=args['stride'],
threshold=1,
tumor_threshold=args['tumor_threshold'],
test=args['test'],
binary_threshold=args['binary_threshold'],
max_width=args['max_width'],
n_patches=args['n_patches'],
test_dataset=test_dataset
)
def extract_features(resnet, dataset, args, image_features):
model = models.EfficientNetB3(resnet=resnet).to(args['device'])
hidden_features_sum = []
if inference_dataset == 'train':
train_labels = dataset.labels
with torch.no_grad():
for x, y in tqdm(dataset, total=len(dataset)):
x = x.to(args['device'])
s = model(x)
hidden_features_sum.append(s.cpu()) #모델을 통과시켜 얻은 값을 hidden_features_sum에 append함. GPU에 있던 Tensor을 CPU로 옮김.
else:
with torch.no_grad():
for x in tqdm(dataset, total=len(dataset)):
x = x.to(args['device'])
s = model(x)
hidden_features_sum.append(s.cpu()) #모델을 통과시켜 얻은 값을 hidden_features_sum에 append함. GPU에 있던 Tensor을 CPU로 옮김.
hidden_features_sum = np.array(torch.cat(hidden_features_sum)) #hidden features들을 concatenate함.
print(hidden_features_sum.shape, resnet)
if args['save']: #hidden_features_sum을 train_df_sum, test_df_sum으로 저장. label도 train_label, test_label로 저장.
df_sum = pd.DataFrame(hidden_features_sum)
save_dir = os.path.join(img_dir, image_features)
if not os.path.exists(save_dir):
os.makedirs(save_dir)
df_sum.to_csv(os.path.join(save_dir, f"{inference_dataset}_df_sum.csv"), index=False)
if inference_dataset == 'train':
label_df = pd.DataFrame(train_labels)
label_df.to_csv(os.path.join(save_dir, f"{inference_dataset}_label.csv"), index=False)
print(args['n_patches'])
resnet = True
if resnet:
save_name = f"{args['n_patches']}_inceptionresnetv2"
else:
save_name = f"{args['n_patches']}_efficientnetb3"
extract_features(resnet, dataset, args, save_name) #여기서 실행.
resnet = False
if resnet:
save_name = f"{args['n_patches']}_inceptionresnetv2"
else:
save_name = f"{args['n_patches']}_efficientnetb3"
extract_features(resnet, dataset, args, save_name)#여기서 실행.Image Classification
- Stratified KFold
데이터가 편항되어 있을 경우(몰려있을 경우) 단순 k-겹 교차검증을 사용하면 성능 평가가 잘 되지 않을 수 있다. 따라서 이러한 경우에는 stratified k-fold cross-validation을 사용한다. 주로 분류 문제에서 사용한다.
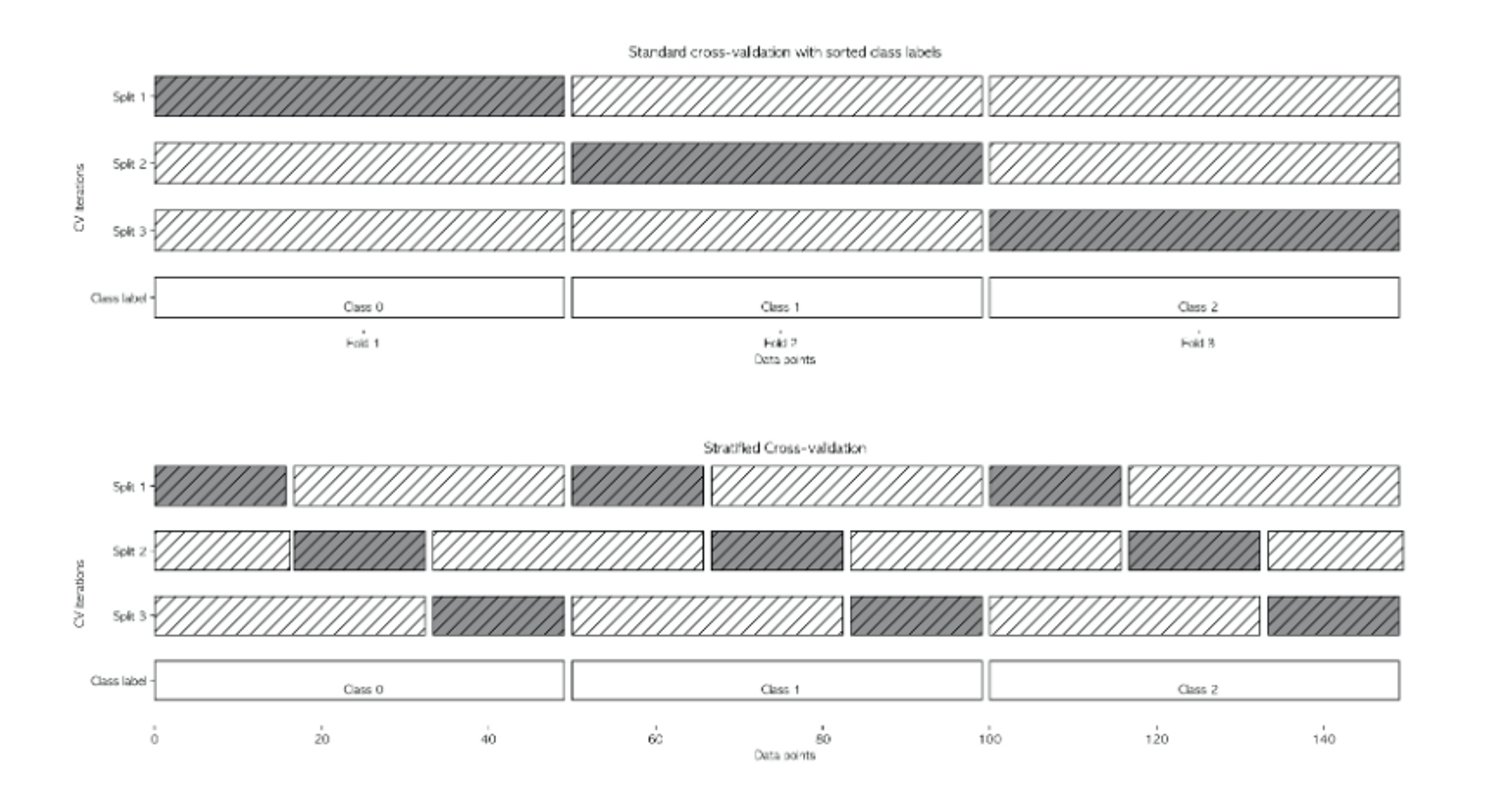
import os
import pandas as pd
import numpy as np
import torch
import random
from tqdm.auto import tqdm
from sklearn.metrics import f1_score
from sklearn.model_selection import StratifiedKFold
from autogluon.tabular import TabularPredictor
import utils
import warnings
warnings.simplefilter(action='ignore', category=FutureWarning)
def main(train, test, label, n_splits, random_seed):
# dataset preprocessing
label = label['0']
train['label'] = label
# train & inference
train_predictions = pd.DataFrame(np.zeros((train.shape[0], 2)), columns=[0, 1]) #train prediction 미리 만들어놓기. 0으로.
test_predictions = pd.DataFrame(np.zeros((test.shape[0], 2)), columns=[0, 1]) #test prediction 미리 만들어놓기. 0으로.
kfold = StratifiedKFold(n_splits=n_splits,
random_state=random_seed,
shuffle=True)
kfold.get_n_splits(train, label)
train_indices = []
val_indices = []
for train_ids, val_ids in kfold.split(train, label):
train_indices.append(train_ids)
val_indices.append(val_ids)
for i in range(n_splits):
train_ids = train_indices[i]
val_ids = val_indices[i]
x_train = train.iloc[train_ids]
x_val = train.iloc[val_ids]
gluon_clf = TabularPredictor(label='label',
path=f'image_train_{i}_fold',
verbosity=0
).fit(x_train, presets=['best_quality'])
train_proba = gluon_clf.predict_proba(x_val)
train_predictions.iloc[val_ids] += train_proba
gluon_clf = TabularPredictor(label='label',
path=f'image_train_full',
verbosity=0
).fit(train, presets=['best_quality'])
test_proba = gluon_clf.predict_proba(test)
test_predictions += test_proba
return train_predictions, test_predictions #Feature Extration 된 것을 autogluon으로 N Category Classification함.
# hyperparameters
random_seed = 42
n_splits = 5
root_dir = "./submission_codes"
data_dir = os.path.join(root_dir, "data", "image_data")
save_dir = os.path.join(root_dir, "data", "clinical_data")
ensemble_list = ['96_efficientnetb3',
'96_inceptionresnetv2',
'120_efficientnetb3',
'120_inceptionresnetv2']
utils.set_seeds(seed=random_seed)
train_ensemble_result = pd.DataFrame(np.zeros((1000, 2)), columns=[0, 1])
test_ensemble_result = pd.DataFrame(np.zeros((1000, 2)), columns=[0, 1])
for k in tqdm(ensemble_list, desc="training...", total=len(ensemble_list)):
train = pd.read_csv(os.path.join(data_dir, k, 'train_df_sum.csv'))
test = pd.read_csv(os.path.join(data_dir, k, 'test_df_sum.csv'))
label = pd.read_csv(os.path.join(data_dir, k, 'train_label.csv'))
train_prediction, test_prediction = main(train=train,
test=test,
label=label,
n_splits=n_splits,
random_seed=random_seed) #train이랑 test에서 predict한 결과값이 나옴.
train_ensemble_result += train_prediction #train은 train에 더함
test_prediction += test_prediction #test는 test에 더한
train_ensemble_result /= len(ensemble_list) #ensemble된거를 len(ensemble)의 수로 나눠줌.
test_ensemble_result /= len(ensemble_list) #ensemble된거를 len(ensemble)의 수로 나눠줌.
# save result
train_ensemble_result.to_csv(os.path.join(save_dir, "train_image.csv"), index=False)
test_ensemble_result.to_csv(os.path.join(save_dir, "test_image.csv"), index=False)Tabular Data
Impute
- Null값 처리
1) 각 칼럼별 Null값 개수를 구하고, Null 값이 너무 많으면 drop한다.
2) Null값들은 KNN Imputation으로 처리한다. - KNN(K-Nearest Neighbor) Inputation
'feature similarity'를 이용해 가장 닮은(근접한) 데이터를 K개를 찾는 방식이다. KDTree를 생성한 후 이를 이용해 가장 가까운 이웃(NN)을 찾는다.
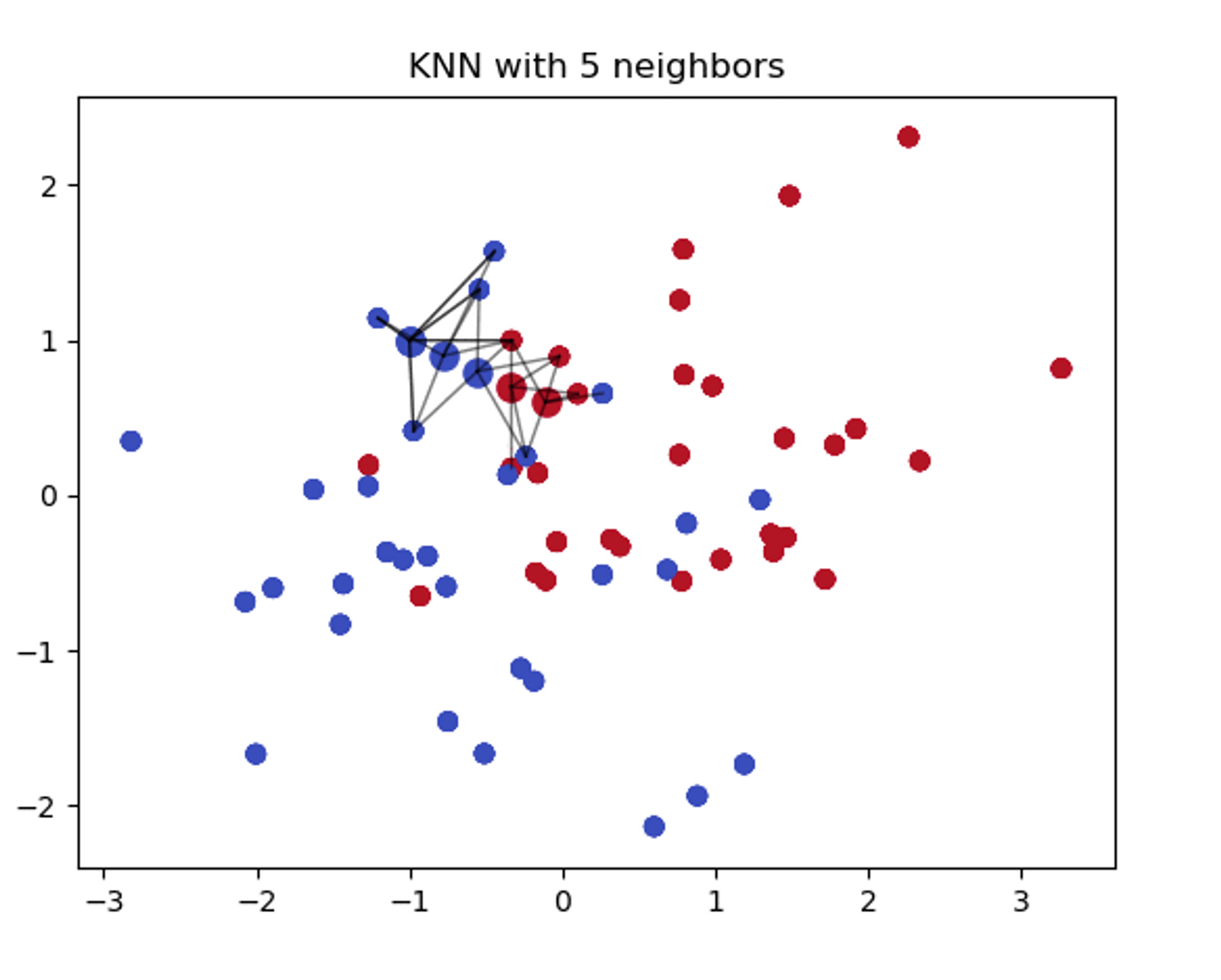
import pandas as pd
import numpy as np
from sklearn.impute import KNNImputer, SimpleImputer
def check_validity(df, origin): #데이터셋의 각 칼럼별 null값 갯수 구하기.
nan_cnt = sum(df.isnull().sum())
if nan_cnt:
raise ValueError('Nan values still exist!')
if set(df.columns) != set(origin.columns):
raise ValueError('Some columns are shifted!')
def knn_impute(train, test):
float_columns = ['나이', '진단명', '암의 위치', '암의 개수',
'암의 장경', 'NG', 'HG', 'HG_score_1', 'HG_score_2', 'HG_score_3',
'DCIS_or_LCIS_여부', 'DCIS_or_LCIS_type', 'T_category', 'ER',
'ER_Allred_score', 'PR', 'PR_Allred_score', 'KI-67_LI_percent', 'HER2',
'HER2_IHC', 'HER2_SISH', 'HER2_SISH_ratio', 'BRCA_mutation'] #numerical columns
str_columns = ['img_path', 'mask_path', '수술연월일', 'ID'] #categorical columns
knn_imputer = KNNImputer() #KNN Imputer 사용
knn_imputer.fit(train[float_columns]) #train데이터로 knn fit.
#float_columns에만 knn imputation 적용
train_knn_imputed = pd.DataFrame(knn_imputer.transform(train[float_columns]), columns=float_columns)
test_knn_imputed = pd.DataFrame(knn_imputer.transform(test[float_columns]), columns=float_columns)
for col in str_columns:
train_knn_imputed[col] = train[col] #numerical column이 knn imputation된 df에 categorical 추가.
train_knn_imputed['N_category'] = train['N_category'] #그리고 N_Category도 df에 추가.
for col in str_columns:
if col == 'mask_path':
continue
test_knn_imputed[col] = test[col] #mask path를 제외하고 categorical data를 knn imputing이 된 test df에 추가.
check_validity(train_knn_imputed, train)
check_validity(test_knn_imputed, test)
return train_knn_imputed, test_knn_imputed, knn_imputerImputation
데이터를 불러와 KNN Imputation을 진행한다.
import os
import pandas as pd
import numpy as np
import utils
import impute
utils.set_seeds(seed=42)
# data directory
root_dir = "C://Users/rlawo/Desktop/Dacon/lymphatic/submission_codes/"
data_dir = os.path.join(root_dir, "data")
save_dir = os.path.join(data_dir, "clinical_data")
# load data
train = pd.read_csv(os.path.join(data_dir, "clinical_data", "train.csv"))
test = pd.read_csv(os.path.join(data_dir, "clinical_data", "test.csv"))
print(f'train.csv has {sum(train.isnull().sum())} Nan values.')
print(f'test.csv has {sum(test.isnull().sum())} Nan values.')
# impute methods > KNN impute
train_impute = train.copy()
test_impute = test.copy()
train_imputed, test_imputed, imputer = impute.knn_impute(train_impute, test_impute)
impute_save_dir = os.path.join(save_dir, "knn_impute")
if not os.path.exists(impute_save_dir):
os.makedirs(impute_save_dir)
train_imputed.to_csv(os.path.join(impute_save_dir, "train_imputed.csv"), index=False)
test_imputed.to_csv(os.path.join(impute_save_dir, "test_imputed.csv"), index=False)Preprocessing
- Date Encoding
’수술연월일’은
1) year
2) month
3) year&month(year*12 + month)
총 3개의 column으로 encoding. - Scaling
1) 암의 장경 - outlier에 의한 noise를 제거하기 위해 median 값인 15를 기준으로 0, 1로 변환
2) Min-Max Scaling 진행.
import os
import pandas as pd
import numpy as np
import utils
from tqdm.auto import tqdm
from sklearn.preprocessing import MinMaxScaler
def date_encoding(csv):
year = []
year_month = []
month = []
for i in range(len(csv)):
date = csv['수술연월일'].iloc[i]
year.append(int(date[2:4]))
year_month.append(int(date[2:4])*12 + int(date[5:7]))
month.append(int(date[5:7]))
csv['year'] = pd.Series(year)
csv['year&month'] = pd.Series(year_month)
csv['month'] = pd.Series(month)
csv = csv.drop('수술연월일', axis=1)
return csv
def scaling(csv, scaler, test=False):
scaling_columns = ['나이', 'year', 'year&month','month', '진단명',
'암의 위치', '암의 개수 cat', 'NG', 'HG',
'HG_score_1', 'HG_score_2', 'HG_score_3', 'DCIS_or_LCIS_여부',
'T_category', 'ER', 'PR', 'HER2', 'HER2_IHC', 'HER2_SISH',
'암의 장경 cat', 'ER_Allred_score', 'PR_Allred_score', 'KI-67_LI_percent']
csv = date_encoding(csv)
csv.drop('DCIS_or_LCIS_type', axis=1, inplace=True)
csv.drop('HER2_SISH_ratio', axis=1, inplace=True)
csv.drop('BRCA_mutation', axis=1, inplace=True)
cnt_cat = csv['암의 개수'] == 2
csv['암의 개수 cat'] = pd.Series([int(i) for i in cnt_cat])
length_cat = csv['암의 장경'] >= 15
csv['암의 장경 cat'] = pd.Series([int(i) for i in length_cat])
csv.drop('암의 개수', axis=1, inplace=True)
csv.drop('암의 장경', axis=1, inplace=True)
if test==False: #test는 train data로부터 fit된 스케일링을 적용해야하니까.
scaler.fit(csv[scaling_columns])
scaled = pd.DataFrame(scaler.transform(csv[scaling_columns]), columns=scaling_columns)
if test==False: #test는 N_Category가 없을테니까.
scaled['N_category'] = csv['N_category'] #scaling된 N_Category를 원래 상태로 복귀시킨다.
return scaled, scaler
else:
return scaled
def preprocessing(file_dir):
# load data
train = pd.read_csv(os.path.join(file_dir, "train_imputed.csv"))
test = pd.read_csv(os.path.join(file_dir, "test_imputed.csv"))
# preprocessing & scaling
# scaling methods : min-max scaling
# min-max scaling
mm_scaler = MinMaxScaler()
train_mm_scaled, train_mm_scaler = scaling(train, mm_scaler, test=False)
test_mm_scaled = scaling(test, train_mm_scaler, test=True)
train_mm_scaled.to_csv(os.path.join(file_dir, "train_mm_scaled.csv"), index=False)
test_mm_scaled.to_csv(os.path.join(file_dir, "test_mm_scaled.csv"), index=False)
utils.set_seeds(seed=42)
# data directory
root_dir = "./submission_codes"
data_dir = os.path.join(root_dir, "data", "clinical_data")
preprocessing(os.path.join(data_dir, 'knn_impute')) #preprocessing된 데이터는 train_mm_scaled, test_mm_scaled로 저장된다.Classification
이미지로부터 얻은 각 class별 확률값과 preprocessing된 tabular 데이터를 concatenate한다.
Auto Gluon으로 classification을 한다.
import os
import pandas as pd
import numpy as np
import torch
import random
from tqdm.auto import tqdm
from sklearn.metrics import f1_score
from sklearn.model_selection import StratifiedKFold
from autogluon.tabular import TabularPredictor
import utils
import warnings
warnings.simplefilter(action='ignore', category=FutureWarning)
# hyperparameters
random_seed = 42
root_dir = "./submission_codes/"
data_dir = os.path.join(root_dir, "data", "clinical_data")
utils.set_seeds(seed=random_seed)
train_image_pred = pd.read_csv(os.path.join(data_dir, "train_image.csv"))
test_image_pred = pd.read_csv(os.path.join(data_dir, "test_image.csv"))
preds = []
probas = []
train = pd.read_csv(os.path.join(data_dir, 'knn_impute', 'train_mm_scaled.csv'))
test = pd.read_csv(os.path.join(data_dir, 'knn_impute', 'test_mm_scaled.csv'))
train['image'] = train_image_pred['1'] #두 데이터를 concatenate
test['image'] = test_image_pred['1'] #두 데이터를 concatenate
label = train['N_category']
gluon_clf = TabularPredictor(label='N_category',
path=f'classification_model',
).fit(train)
predictions = gluon_clf.predict(test)
# load & fill submission file
submission = pd.read_csv(os.path.join(data_dir, "sample_submission.csv"))
submission['N_category'] = pd.Series(predictions)
# check validity of submission file
nan_cnt = sum(submission.isnull().sum())
if nan_cnt:
assert ValueError(f'Nan value detected! : {nan_cnt}')
# save submission file
submission.to_csv(os.path.join(root_dir, "submission.csv"), index=False)