(Ubuntu 18.04.6 LTS)
2022.03.18.
C++, VS code 사용
프로그래머스 자율주행 데브코스 3기
histogram stretch
이전의 대조는 특정값을 기준으로 분포를 나누는 형태로 주어졌다. 그 값은 주로 평균값이나 밝기의 중간값인 128인 경우가 많았다.
dst = src + (src - m) * alpha
dst = src + (128 - m)histogram stretch는 histogram 상 분포에서 최대,최소값을 이용하여 밝기를 조절한다.
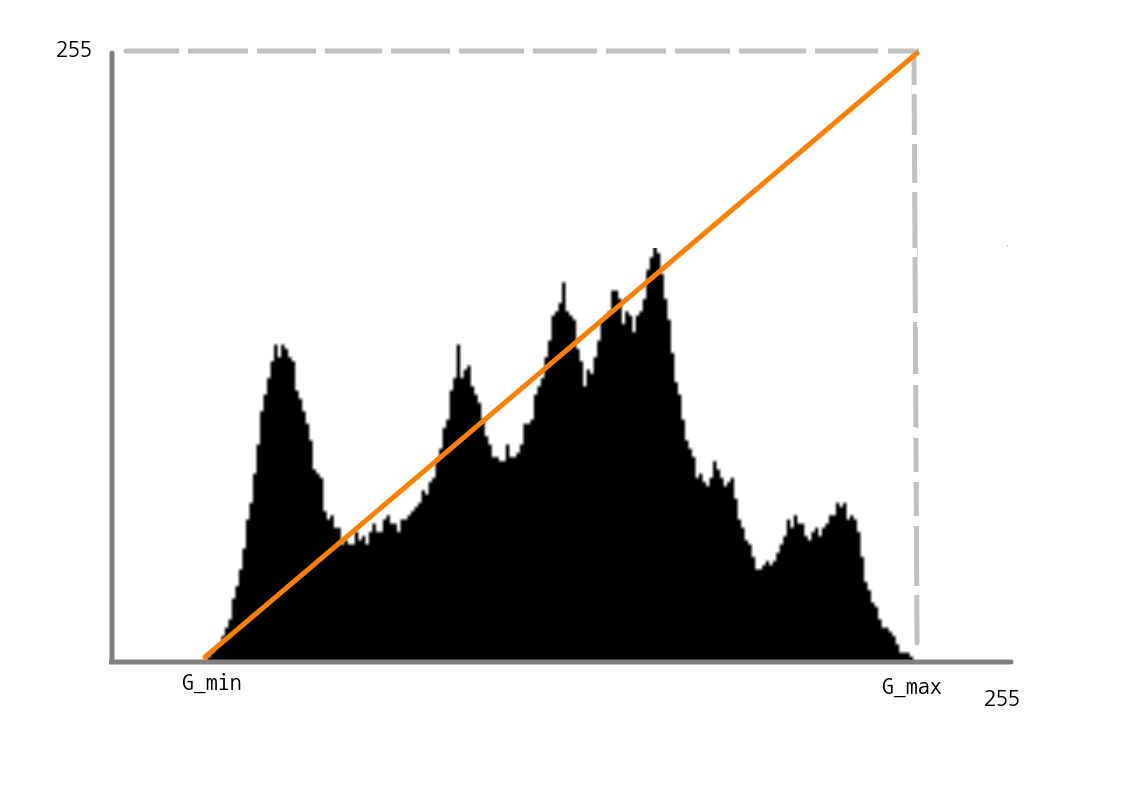
적용되는 공식은 아래와 같다.
dst = 255/(G_max - G_min) * (src - G_min)openCV에서 최대 최소값을 구하는 함수는 cv::minmaxLoc이다.
void cv::minMaxLoc ( InputArray src,
double * minVal,
double * maxVal = 0,
Point * minLoc = 0,
Point * maxLoc = 0,
InputArray mask = noArray()
) minVal, maxVal에는 각각의 값을 담을 double형 변수의 주소를 넣으면 된다.
Point도 추가적으로 넣으면 해당 위치를 Point로 받을 수 있다.
cv::normalize를 이용해서도 histogram stretch를 수행할 수 있다.
void cv::normalize ( InputArray src,
InputOutputArray dst,
double alpha = 1,
double beta = 0,
int norm_type = NORM_L2,
int dtype = -1,
InputArray mask = noArray()
) norm_type을 NORM_MINMAX로 설정한 경우 alpha는 최소, beta는 최대값으로 사용할 수 있다.
// 예시 코드
#include <iostream>
#include "opencv2/opencv.hpp"
int main()
{
cv::Mat src = cv::imread("./resources/lenna.bmp",cv::IMREAD_COLOR);
if (src.empty()){
std::cerr << "Image load failed!" << std::endl;
return -1;
}
double gmin,gmax;
cv::minMaxLoc(src,&gmin,&gmax);
cv::Mat dst = (src-gmin)*255/(gmax-gmin);
cv::Mat dst2,src2;
cv::cvtColor(src,src2,cv::COLOR_BGR2GRAY);
cv::normalize(src2,dst2,0,255,cv::NORM_MINMAX);
cv::imshow("image",src);
cv::imshow("dst",dst);
cv::imshow("image2",src2);
cv::imshow("dst2",dst2);
cv::waitKey();
cv::destroyAllWindows();
return 0;
}하지만 최대값, 최소값 주변의 pixel 수가 적거나 noise가 발생하였을 때 단순한 최대 최소값만으로는 수행에 어려움이 있다. 일정 비율을 넘었을 때 그 값을 기준으로 histogram stretch를 수행할 수 있으며 그 코드는 아래와 같다.
#include "opencv2/opencv.hpp"
#include <iostream>
void histogram_stretching(const cv::Mat& src, cv::Mat& dst);
void histogram_stretching_mod(const cv::Mat& src, cv::Mat& dst);
int main()
{
cv::Mat src = cv::imread("./resources/lenna.bmp", cv::IMREAD_GRAYSCALE);
if (src.empty()) {
std::cerr << "Image load failed!" << std::endl;
return -1;
}
cv::Mat dst1, dst2;
histogram_stretching(src, dst1);
histogram_stretching_mod(src, dst2);
cv::imshow("src", src);
cv::imshow("dst1", dst1);
cv::imshow("dst2", dst2);
cv::waitKey();
}
void histogram_stretching(const cv::Mat& src, cv::Mat& dst)
{
double gmin, gmax;
cv::minMaxLoc(src, &gmin, &gmax);
dst = (src - gmin) * 255 / (gmax - gmin);
}
void histogram_stretching_mod(const cv::Mat& src, cv::Mat& dst)
{
int hist[256] = {0,};
for(int y=0;y<src.rows;y++){
for(int x=0;x<src.cols;x++){
hist[src.at<uchar>(cv::Point(y,x))]+=1;
}
}
int gmin, gmax;
int ratio = int(src.cols * src.rows * 0.01);
for (int i = 0, s = 0; i < 255; i++) {
s += hist[i];
if (s>=ratio){
gmin = i;
break;
}
}
for (int i = 255, s = 0; i >= 0; i--) {
s += hist[i];
if (s>=ratio){
gmax = i;
break;
}
}
dst = (src-gmin)/(gmax-gmin)*255;
}
가장 왼쪽이 기존 사진, 두번째가 일반적인 histogram stretch, 3번째 사진이 1%에서 histogram stretch를 한 것으로, 조금 더 선명도가 높은 것을 볼 수 있다.
histogram equalization
histogram equalization은 전체 구간에서 균일한 분포로 나타나도록 변경하는 기법으로, 히스토그램을 구하고, 정규화한 후 누적 분포 함수를 구한뒤 최대값을 곱한 값을 반올림한다. 아래의 위키 사이트의 내용을 보고 작성하였다.
https://en.wikipedia.org/wiki/Histogram_equalization
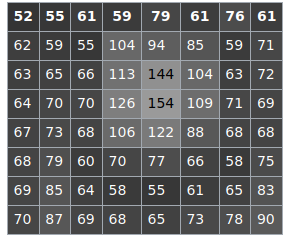
이미지의 명암값들을 텍스트 파일에 넣었다. 이후 파이썬을 이용해서 개수를 구해보았다.
# python 3
f = open("counti.txt",'r')
hist = [0]*256
while True:
line = f.readline()
if not line: break
hist[int(line)]+=1
hist_sum = float(sum(hist))
cdf = 0
for idx, count in enumerate(hist):
if (count!=0):
norm_hist=count/hist_sum
cdf += norm_hist
dist_val = int(round(cdf*255,0))
print(f'{idx:3}: count: {count}, norm_hist = {norm_hist:.3f}, cdf = {cdf:.3f}, dst_val = {dist_val}')
f.close결과값은 아래와 같다. count는 개수, norm_hist는 전체 개수로 나눈 값, cdf는 cumulative distribution function이다. 이를 255 범위에서 평활화하기 위해서는 cdf에 255를 곱한 후 반올림해주었다.
52: count: 1, norm_hist = 0.016, cdf = 0.016, dst_val = 4
55: count: 3, norm_hist = 0.047, cdf = 0.062, dst_val = 16
58: count: 2, norm_hist = 0.031, cdf = 0.094, dst_val = 24
59: count: 3, norm_hist = 0.047, cdf = 0.141, dst_val = 36
60: count: 1, norm_hist = 0.016, cdf = 0.156, dst_val = 40
61: count: 4, norm_hist = 0.062, cdf = 0.219, dst_val = 56
62: count: 1, norm_hist = 0.016, cdf = 0.234, dst_val = 60
63: count: 2, norm_hist = 0.031, cdf = 0.266, dst_val = 68
64: count: 2, norm_hist = 0.031, cdf = 0.297, dst_val = 76
65: count: 3, norm_hist = 0.047, cdf = 0.344, dst_val = 88
66: count: 2, norm_hist = 0.031, cdf = 0.375, dst_val = 96
67: count: 1, norm_hist = 0.016, cdf = 0.391, dst_val = 100
68: count: 5, norm_hist = 0.078, cdf = 0.469, dst_val = 120
69: count: 3, norm_hist = 0.047, cdf = 0.516, dst_val = 131
70: count: 4, norm_hist = 0.062, cdf = 0.578, dst_val = 147
71: count: 2, norm_hist = 0.031, cdf = 0.609, dst_val = 155
72: count: 1, norm_hist = 0.016, cdf = 0.625, dst_val = 159
73: count: 2, norm_hist = 0.031, cdf = 0.656, dst_val = 167
75: count: 1, norm_hist = 0.016, cdf = 0.672, dst_val = 171
76: count: 1, norm_hist = 0.016, cdf = 0.688, dst_val = 175
77: count: 1, norm_hist = 0.016, cdf = 0.703, dst_val = 179
78: count: 1, norm_hist = 0.016, cdf = 0.719, dst_val = 183
79: count: 2, norm_hist = 0.031, cdf = 0.750, dst_val = 191
83: count: 1, norm_hist = 0.016, cdf = 0.766, dst_val = 195
85: count: 2, norm_hist = 0.031, cdf = 0.797, dst_val = 203
87: count: 1, norm_hist = 0.016, cdf = 0.812, dst_val = 207
88: count: 1, norm_hist = 0.016, cdf = 0.828, dst_val = 211
90: count: 1, norm_hist = 0.016, cdf = 0.844, dst_val = 215
94: count: 1, norm_hist = 0.016, cdf = 0.859, dst_val = 219
104: count: 2, norm_hist = 0.031, cdf = 0.891, dst_val = 227
106: count: 1, norm_hist = 0.016, cdf = 0.906, dst_val = 231
109: count: 1, norm_hist = 0.016, cdf = 0.922, dst_val = 235
113: count: 1, norm_hist = 0.016, cdf = 0.938, dst_val = 239
122: count: 1, norm_hist = 0.016, cdf = 0.953, dst_val = 243
126: count: 1, norm_hist = 0.016, cdf = 0.969, dst_val = 247
144: count: 1, norm_hist = 0.016, cdf = 0.984, dst_val = 251
154: count: 1, norm_hist = 0.016, cdf = 1.000, dst_val = 255각각의 값에 따른 테이블이 만들어진 것으로 해석할 수 있으며 예를 들어 원본의 144 값은 평활화 후 251로 주어질 수 있다. 변경 후 밝기 값은 균일하게 분포하게된다.
filtering
필터링은 필요한 정보만 통과시키는 방법으로, 공간적 필터링을 다루는 영상 처리에서는 mask, kernel이라고 부르는 행렬을 이용하여 blur, edge detection 등의 필터링을 수행한다. OpenCV에서는 cv::filter2D 함수를 이용하여 수행할 수 있다.
void cv::filter2D ( InputArray src,
OutputArray dst,
int ddepth,
InputArray kernel,
Point anchor = Point(-1,-1),
double delta = 0,
int borderType = BORDER_DEFAULT
) ddepth는 데이터의 depth를 의미하며, -1로 둘 경우 소스와 동일하게 처리된다. 종류는 아래의 링크에서 확인할 수 있다.
https://docs.opencv.org/3.4/d4/d86/group__imgproc__filter.html#filter_depths
kernel은 필터링을 하기 위한 행렬이다.
anchor는 고정점 위치인데, Point(-1,-1)일 경우 필터의 중앙을 고정점으로 사용한다.
delta는 추가적으로 더할 값이고, borderType은 가장자리에서 처리 방식을 결정한다. border type에 대한 정보는 아래에서 더 확인할 수 있다.
https://docs.opencv.org/3.4/d2/de8/group__core__array.html#gga209f2f4869e304c82d07739337eae7c5a697c1b011884a7c2bdc0e5caf7955661
// 예제 코드: 엠보싱.
#include <iostream>
#include "opencv2/opencv.hpp"
int main()
{
cv::Mat src = cv::imread("./resources/lenna.bmp",cv::IMREAD_GRAYSCALE);
float data[] = {-1,-1,0,-1,0,1,1,1,1};
cv::Mat kernel(3,3,CV_32FC1,data);
cv::Mat dst;
if (src.empty()){
std::cerr << "Image load failed!" << std::endl;
return -1;
}
cv::filter2D(src,dst,-1,kernel);
cv::imshow("image",src);
cv::imshow("dst",dst);
cv::waitKey();
cv::destroyAllWindows();
return 0;
}예제 코드는 엠보싱을 구현하기 위한 필터 마스크를 생성한 것이며 엠보싱에 대한 정보는 아래 링크에서 확인 가능하다.
https://en.wikipedia.org/wiki/Image_embossing
코드 실행 결과는 아래와 같다.
