라이브러리 불러오기
import pandas as pd
import numpy as np
# 데이터 분포 확인을 위한 plt 라이브러리 import
import matplotlib.pyplot as plt
import seaborn as sns
#sklearn 에서 제공하는 데이터 셋 중 하나인 diabetes 불러오기
from sklearn.datasets import load_diabetes
#회귀분석 라이브러리 import
from sklearn.linear_model import LinearRegression
lr = LinearRegression()데이터 가져오기
이 sklearn 데이터 세트에는 인구 통계 및 임상 측정을 포함하여 당뇨병 환자 442명에 대한 정보가 포함되어 있다.
- 나이
- 섹스
- 체질량지수(BMI)
- 평균 혈압
- 6가지 혈청 측정(예: 총 콜레스테롤, 저밀도 지질단백질(LDL) 콜레스테롤, 고밀도 지질단백질(HDL) 콜레스테롤).
- 당뇨병 질환 진행(HbA1c)의 정량적 측정.
diabetes = load_diabetes()자료구조 확인 - sklearn.utils._bunch.Bunch .
# key 와 value 값으로 나뉘어 저장되어 있고, dictionary 구조와 유사합니다.
type(diabetes), diabetes
# key 확인. 각각의 key 값에 값이 존재.
diabetes.keys()dict_keys(['data', 'target', 'frame', 'DESCR', 'feature_names', 'data_filename', 'target_filename', 'data_module'])
데이터 프레임만들어주기
- concat사용해도 괜찮음.
import pandas as pd
import numpy as np
from sklearn import datasets
# 데이터 로드
diabetes = datasets.load_diabetes()
# 데이터와 타겟을 데이터프레임으로 변환
data_df = pd.DataFrame(data=diabetes.data, columns=diabetes.feature_names) # feature 데이터
target_df = pd.DataFrame(data=diabetes.target, columns=['target']) # target 데이터
# 두 데이터프레임을 가로로 합치기
df = pd.concat([data_df, target_df], axis=1)
print(df.head())- np.c_ 사용하기
# np.c_ 는 두 배열을 가로 방향으로 합치는 함수입니다.
df= pd.DataFrame(data=np.c_[diabetes.data, diabetes.target], columns=diabetes.feature_names + ['target'])
# 총 442 명에 대한 나이, 성별, bmi(체질량지수).. 등을 가져왔습니다.
df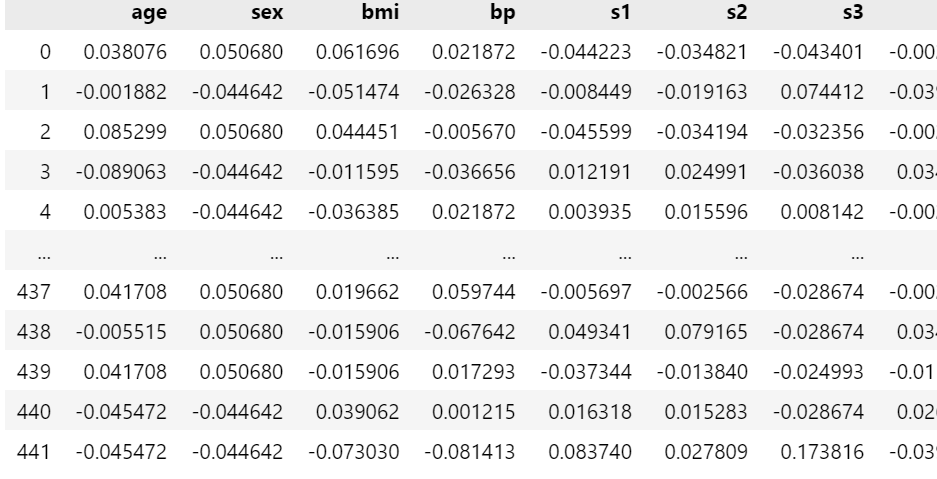
히스토 그램으로 데이터 분포 살펴보기
df.hist(bins=20, figsize=(12, 6), color='blue')
plt.tight_layout() #자동으로 레이아웃 맞춰주는 함수
plt.show()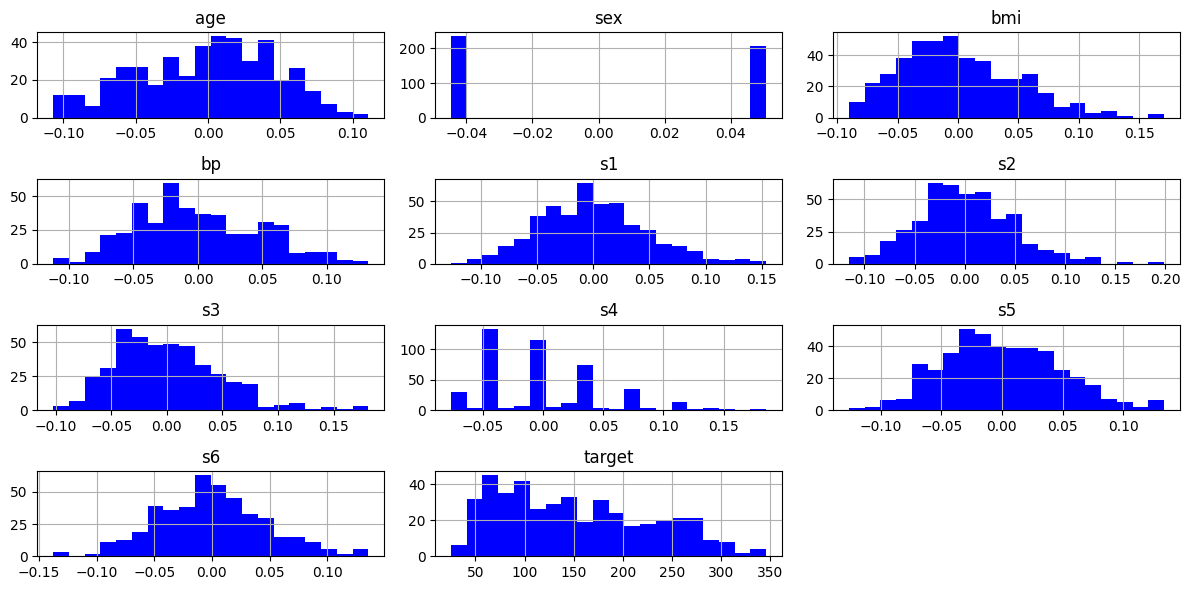
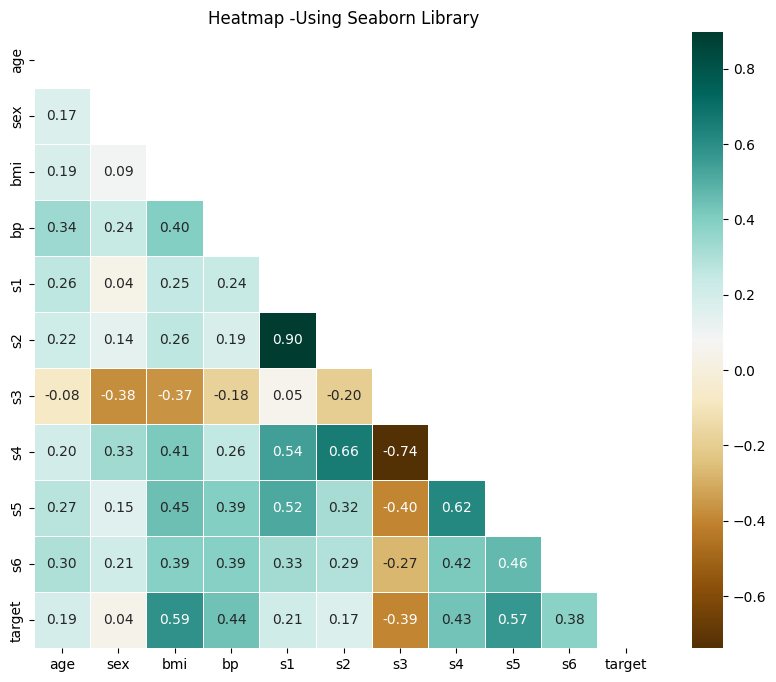
특성간 상관관계
# 삼각형 형식으로 보여주기
corr_matrix = df.corr()
plt.figure(figsize=(10, 8))
mask = np.triu(np.ones_like(corr_matrix, dtype=bool))
sns.heatmap(corr_matrix,annot=True, cmap='BrBG', linewidths=0.5, mask=mask, fmt=".2f")
plt.title("Heatmap -Using Seaborn Library")
plt.show()
-> bp와 당뇨병의 상관관계는 0.44로 높은 편은 아니지만 어느정도의 상관관계를 보이는것 같음.
종속변수와 독립변수 관계 파악하기(둘다 연속형)
sns.scatterplot(x='bp', y='target', data=df, color='green', marker='*')
plt.xlabel("BP")
plt.ylabel("Progression of Diabetes") # 당뇨병 진행 정도
plt.title("Relationship between Progression of Diabetes & BP")
plt.show()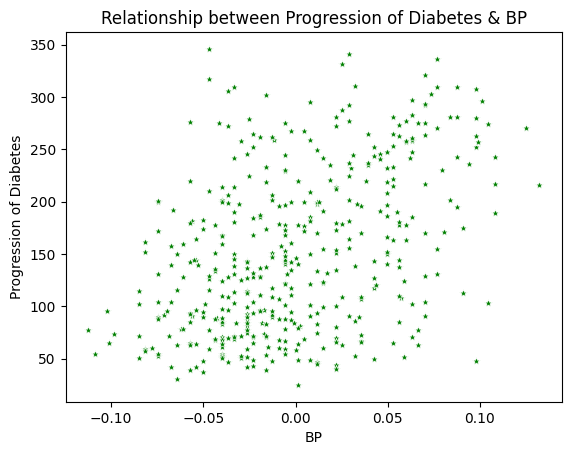
# Diabetes(당뇨병) 데이터셋 로드
diabetes = load_diabetes()
X = df.bp.values
y = df.target.values
X
X.shape(442,)
-> 2차원 배열로 바꿔줄 필요가 있음
# 단순선형회귀 모델 선언
model = LinearRegression()
# 1차원 -> 2차원 변경
X = X.reshape(-1, 1)
X.shapeX.shape
모델 학습하기
# 모델 학습. FIT 을 사용.
model.fit(X, y)
회귀식 만들기
회귀식: y= 152.133 + 714.738*x
coef 가 기울기를, intercept_가 Y 절편을 의미.
model.coef_[0], model.intercept_(np.float64(714.7382594960411), np.float64(152.13348416289597))
회귀선 추가하기
plt.scatter(X, y, alpha=0.5, color='green', marker='*')
plt.plot(X, model.predict(X), color='red', linewidth=2)
plt.xlabel("BP")
plt.ylabel("progression of diabetes") # 당뇨병 진행 정도
plt.title("Relationship between Progression of Diabetes & BP")
plt.show()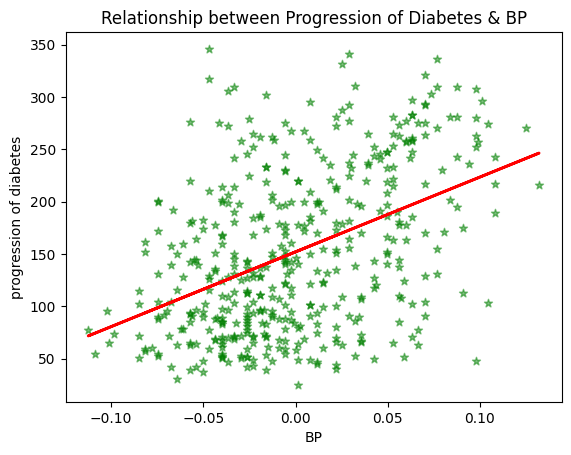
결과 해석하기
- 결정계수 R-squared 확인, 모형의 적합도 Prob(F-statistic) 확인, P>|t| 확인
import statsmodels.api as sm
results = sm.OLS(y, sm.add_constant(X)).fit()
results.summary()
# 메시지 Standard Errors assume that the covariance matrix of the errors is correctly specified.
# 메시지 해석 데이터 관측치의 부족으로 첨도 테스트에 문제가 있다는 경고(common notice 정도로 이해해주세요)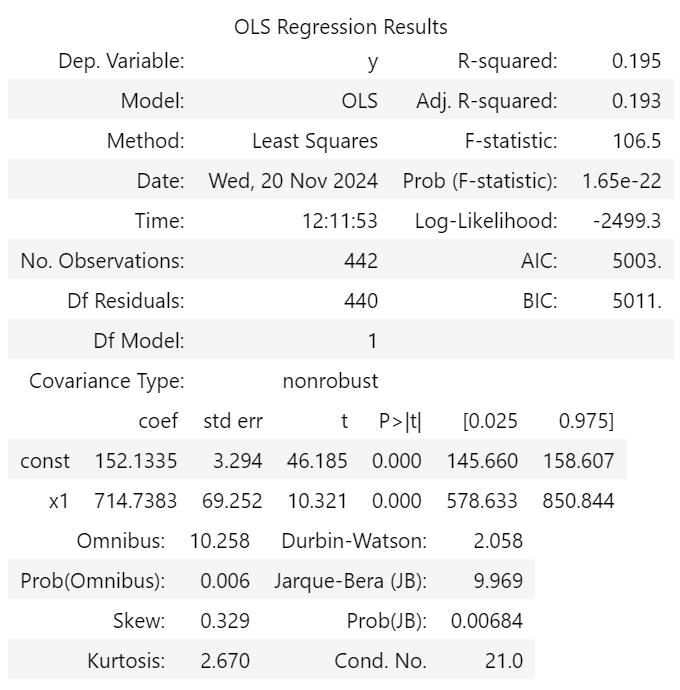
A. R제곱
값이 0.195정도로 이는 19%만큼의 설명력을 가진다고 판단할 수 있다.
0에 가까울 수록 예측값을 믿을 수 없고 1에 가까울 수록 믿을 수 있다고 판단한다.
B. Prob(F-statistic)
도출된 회귀식이 회귀분석 모델 전체에 대해 통계적으로 의미가 있는지 파악.
-> F-statistic의 p-value 값은 Prob(F-statistic)으로 표현되는데,
이는 1.65e-22로 0.05보다 작기에 이 회귀식은 회귀분석 모델 전체에 대해 통계적으로 의미가 있다고 볼 수 있다.
C. P>|t|
각 변수가 종속변수에 미치는 영향이 유의한지 파악.
x1과 const에 대한 p-value가 0.000으로 표기 되어 있기에 0.05보다 작으므로 target을 설명하는데 유의하다고 볼 수 있다.