https://arxiv.org/pdf/1505.04597.pdf
Introduction
기존 전형적인 CNN은 한 이미지 당 하나의 class label을 결과로 보여주는 분류 작업에서 많이 이용됨. 하지만, biomedical task에서는 단순 이미지 분류 뿐 아니라, 각 pixel level의 class label이 할당되어야 할 필요가 있다(segmentation).
각 pixel의 class label을 예측하기 위해 학습 과정에서 해당 픽셀의 주변 patch를 입력으로 제공하여 ISBI segmentation challenge 2012에서 우승한 기존 연구도 있지만, 아래 두 가지 단점이 존재했다.
- network가 각 패치별로 실행되어야 하기 때문에 느리고, 겹치는 patch로 인한 중복 발생
- patch의 크기에 따라 localization accuracy와 use of context 간 trade-off가 존재한다. patch가 클수록 max pooling layer가 더 많이 필요하여 localization accuracy는 줄어들지만, patch가 작으면 네트워크가 적은 context만을 볼 수 있음.
따라서 저자들은 아래와 같이 FCN 구조를 변형하여 문제에 접근하였다.
Network Architecture
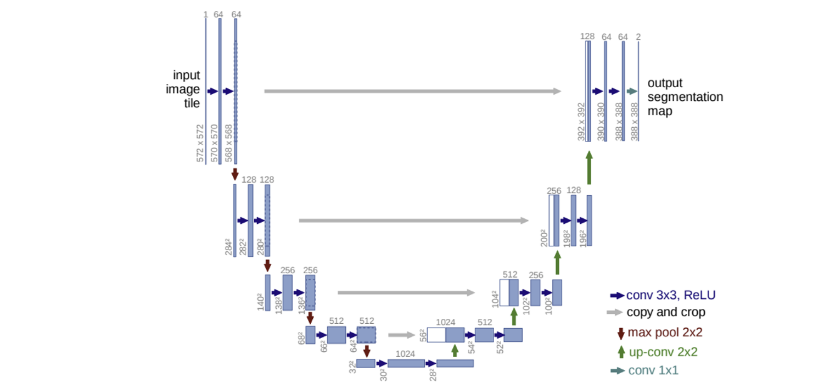
왼쪽은 contracting path, 오른쪽은 expansive path이다.
Contracting path
- 각 contracting block에서는 두 번의 3x3 convolution과 각각 ReLU로 활성화 된다.
- 다음 block으로 넘어갈 땐 stride 2의 2x2 max pooling으로 downsampling 되고, channel 수는 두 배가 된다.
Expansive path
- contracting path와 마찬가지로 각 expanding block에서는 3x3 convolution을 두 번 반복하고(각각 ReLU 활성화), 다음 block으로 넘어갈 때 2x2 Up-conv로 feature map은 upsampling되고, channel은 반으로 줄어든다.
- 각 단계 별 feature map은 대칭인 contracting path의 feature map과 concatenation 된다(contracting path의 feature map이 크기 때문에 테두리 부분은 crop)
- 마지막 layer에서는 각 feature vector를 원하는 class 수로 mapping하기 위 1x1 convolution 연산
FCN의 main idea는 pooling layer들을 upsampling operators로 대체하여 contracting network를 보강하는 것. localizing을 위해 contracting path로부터 나온 고해상도 feature들이 upsampled output과 결합된다.
Upsampling part에서는 많은 feature channel이 존재하기 때문에 network가 context information을 고해상도 층으로 전파할 수 있다. 그 결과 expansive path와 contracting path는 유사해지고, 결합하여 U 모양의 구조가 나오게 된다.
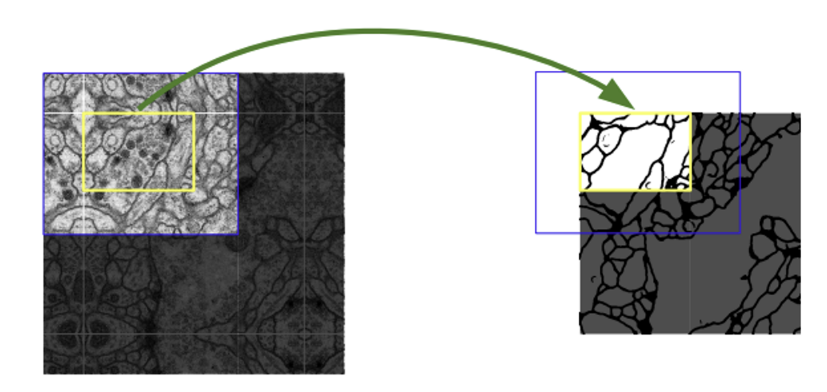
위 구조를 유심히 보면, input image의 크기는 572 X 572이지만, 결과 segmentation map은 388 X 388이다. 이는 각 layer에서 padding 없이 convolution을 통과했기 때문이다.
따라서 파란 부분을 input으로 하면 노란 부분을 output으로 받는다. 그리고 다음 tile에 대한 결과를 얻으려면 이전 input인 파란 부분을 일부 포함하는 input이 들어와야 한다. 이를 Overlap-title 기법이라고 한다.
input에 비해 border region이 잘린 output이 나오기에, 부족한 부분은 mirroring extrapolation을 이용하여 채워진다.
Training
Energy function은 마지막 feature map에서 pixel-wise의 softmax와 cross entropy를 결합하여 계산된다.
이후 픽셀 고유의 weight를 곱해주어 Loss 값이 결정된다.
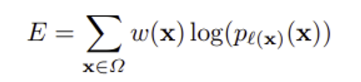
파라미터 최적화에서는 momentum 0.99, 초기화에서는 gaussian distribution를 사용하였다.
이 때 weight map은 각 ground truth segmentation에 대해 미리 계산되어 훈련 데이터 셋에서 특정 클래스의 빈도 차이를 보상해주고, 접촉하는 세포들 사이의 작은 경계를 구분하도록 학습된다.
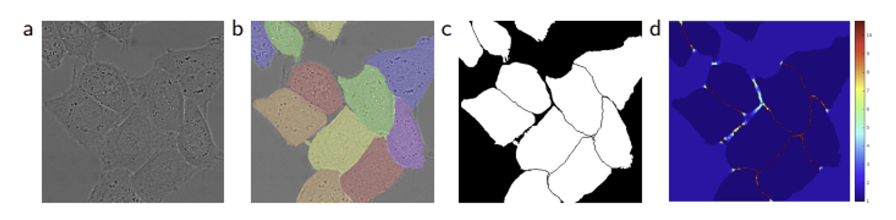
이렇게 세포 사이의 경계에 대한 가중치를 더 크게 주는 이유는 cell segmentation task에서는 동일한 class인 touching objects를 분리 해내는 것이 하나의 큰 과제이기 때문이다. a는 raw image, b는 ground truth segmentation, c는 생성된 segmentation mask, d는 pixel-wise loss weight map을 그린 것이다.
d를 보면 세포 간 경계의 weight가 높은 것을 확인할 수 있고, 이에 따라 c처럼 세포 별 경계 분리가 잘 된 segmentation mask를 얻을 수 있어야 한다.
weight map은 아래와 같이 계산된다.
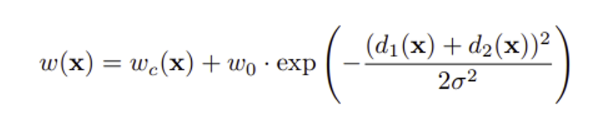
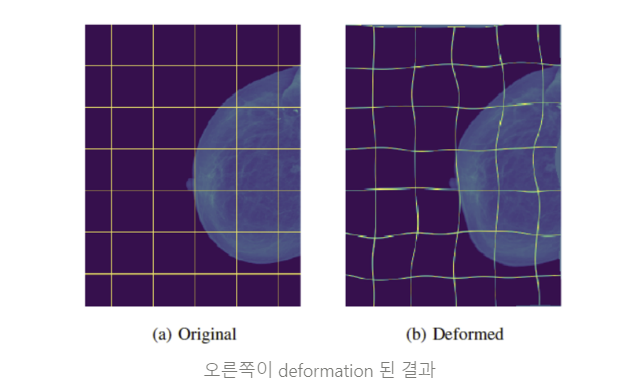
Data Augmentation
Shift, rotation, gray value variations 등의 전형적인 augmentation 뿐 아니라 elastic deformation 같은 기법도 추가로 활용했는데, 이것이 few annotated images에서 segmentation network의 핵심이 된다고 한다.
Results
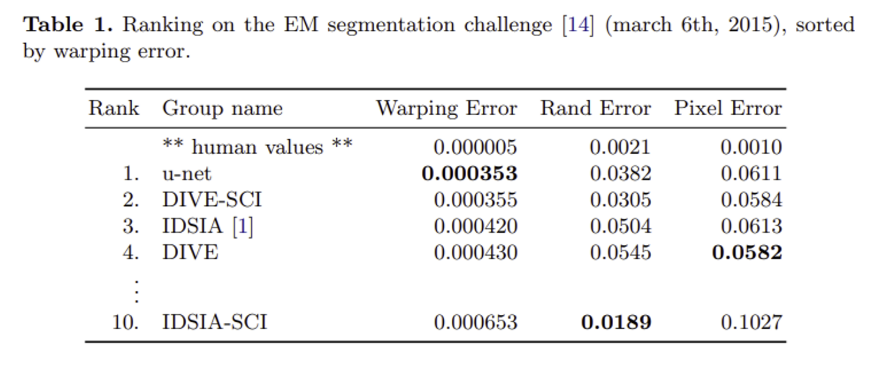
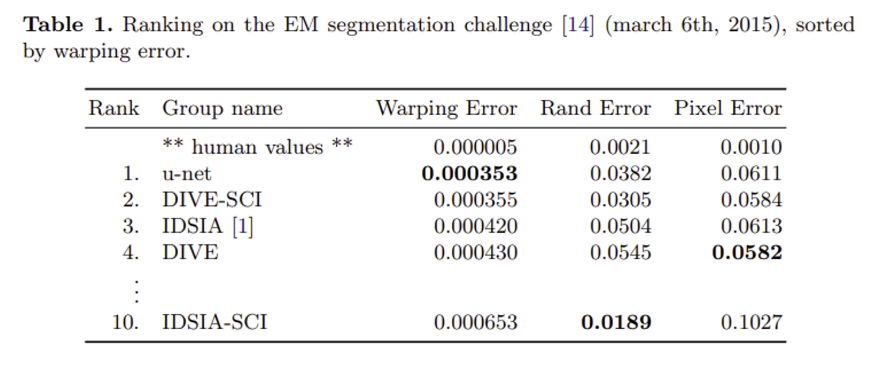
위와 같은 방법으로 EM segmentation challenge(training dataset은 512x512 이미지 30장이었다고 한다..)에서 우승하였고, ISBI cell tracking challenge 2015에서도 다른 SOTA 모델에 비해 좋은 결과를 낼 수 있었다.