DTI 공부를 시작할 때는 논문, 코드, 책 으로 하지말고 데이터를 먼저 explore 하면서 익숙해지는게 먼저라고 한다. 그래서 단백질 정보데이터들을 찾을 수 있는 RCSB PDB 사이트를 알아보았다.
PDB는 Protein Data Bank 로 단백질 구조에 대한 정보 및 시각적으로도 확인 할 수 있다.
PDB 파일을 notepad 에서 열어보면 다음과 같은 정보를 얻을 수 있다.
대표적인 Record Type(6문자)
REMARK : 0-999 여러 기록들 기재, ex) 465-missing residues
ATOM : 단백질 구성 원자
HETATM : 리간드 원자
SEQRES : 아미노산 서열
CONECT : 원자 간 결합 ex) CONECT 18679(기준) 18680 18683 18711
찾아보니 이와 관련된 LAIDD 강의가 있는 것 같아 수강하기로 했다. 2강 밖에 없어서 금방 들을 수 있을 것 같다.

1강
실습환경 구축
실습은 linux 로 진행되어 ubuntu WSL을 설치하였다.
--> sudo apt update --> upgrade
ubuntu 설치 --> anaconda3 설치 --> pymol 설치
완전복잡하다! 나도 내가 어떻게 했는지 모르겠다.
막혔던 부분 :
- WSL2로 업그레이드
- GUI(Xming)
중요 : 재부팅시 display 안되는 문제 해결
Xming 실행 확인
오류해결링크
모를 땐 검색!!
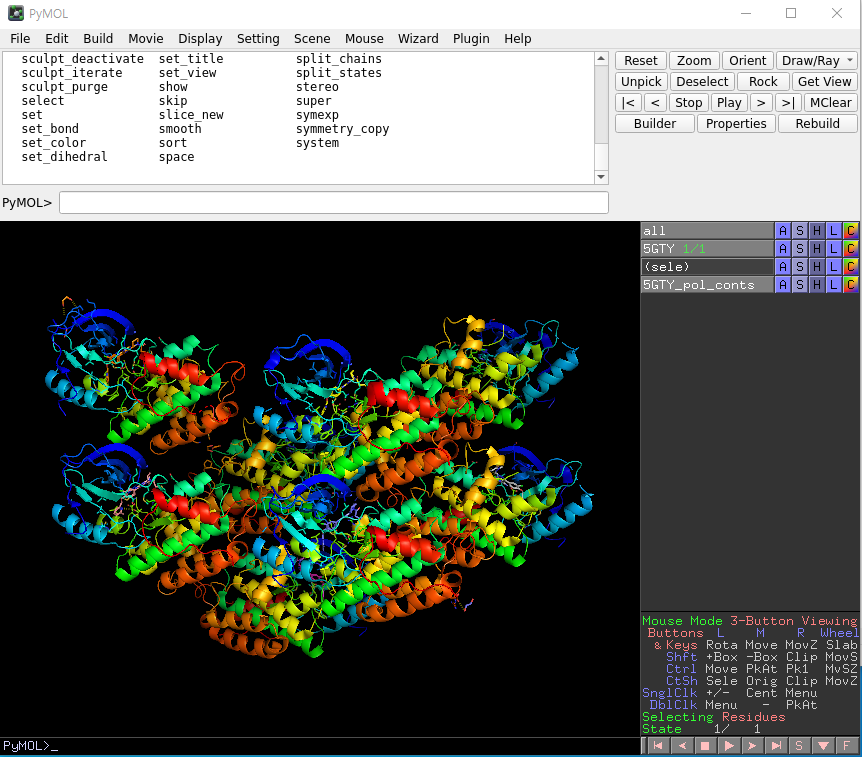
위 그림은 pdb 파일을 pymol에서 불러온 'EGFR' 이다.
window에 설치 되어있는 파일을 linux 환경으로 불러오기
그냥 explorer.exe . 해서 파일로 옯기기
실습파일 :
1. pdb2seq : seq 추출
2. pdb_coor : 좌표 추출(거리계산)
3. protein_ligand : protein ligand 분리(도킹 계산)
1. pdb2seq
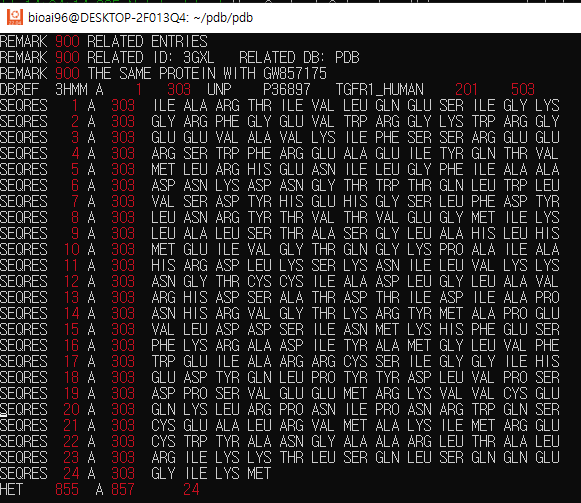
SEQRES 파싱
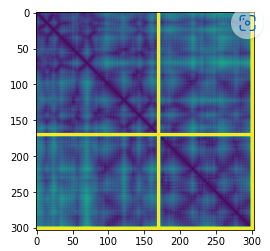
2. pdb_coor
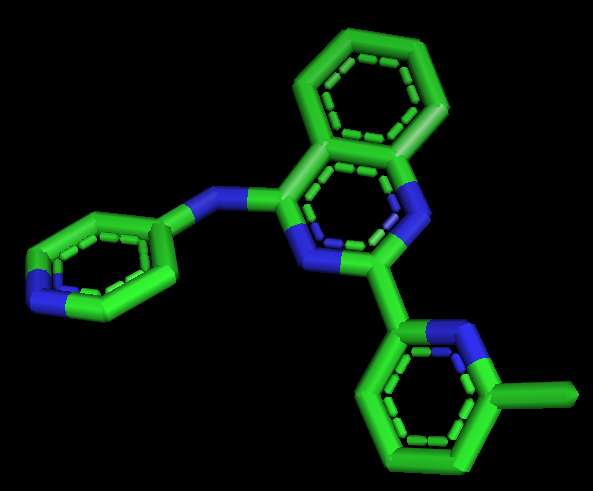
3. protein_ligand
protein 과 ligand 분리 --> Vi에서 하는 방법, 코드로 하는 방법 존재
2강
pdb 수집 및 분석 자동화
UniProtKB
정보: 단백질 기능, 이름, 세포에서의 위치, 질병, 변이, 발현, 상호작용, 단백질 서열, 구조, 패밀리, 도메인 등
PBI(protein bioinformatics) toolkit
기능 : pdb 파일 목록 다운로드, pdb 파일 다운로드, chain 분리, 구조 정렬, ligand 분리, protein fix, ligand fix
다음 과정은 단백질과 분자 간 도킹을 분석할 수 있는 프로그램에 관련된 내용이다.
후에 필요하면 수강을 해야겠다.
