https://arxiv.org/abs/2307.08919
CVPR 2024
1. INTRODUCTION
- Deep neural networks의 뛰어난 classification 성능은 large scale labeled 데이터로 학습될 때 가능
- Medical image 분야에서는 대규모 label 데이터셋 구축이 시간과 비용 측면에서 어려움
- Label이 없는 image 데이터는 건강 기록 데이터베이스에서 쉽게 얻을 수 있음
- 최근 연구는 small labeled 데이터와 large unlabel 데이터를 활용하는 method 개발에 집중
- 두 가지 주요 접근법
- Semi-supervised learning
- 두 개의 loss term을 사용하는 공동 학습
- Self-supervised learning
- Unlabel 데이터로 representation learning 후 labeled 데이터로 classifier 학습
- Semi-supervised learning
"Which recent semi- or self-supervised methods are likely to be most effective?"
- 성능은 hyperparameters에 민감하지만 현재 벤치마크들은 현실적이지 않은 설정 사용
- 현실적인 상황에서 hyperparameter tuning의 효과에 대한 의문 제기
- 연구 목표: 자원이 제한된 상황에서 semi/self-supervised method 비교
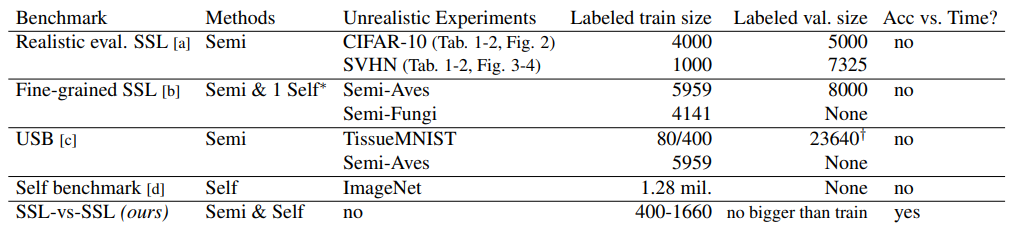
- 아래 테이블과 같이, 4개의 데이터셋을 사용하여 실험, 다양한 해상도와 클래스 수 포함
Contributions
- Semi-supervised 및 self-supervised method의 체계적인 비교 제공
- 현실적인 실험 프로토콜 채택, 과도하게 큰 validation set 사용을 피함
- 현실적인 크기의 validation set에서도 hyperparameter tuning이 가능하고 필요함을 보여줌
2. BACKGROUND AND METHODS
Unified Problem Formulation
- Chen et al. [21]의 연구를 따라 supervised, semi-supervised, self-supervised learning을 통합하여 문제를 정의
- Labeled set : feature-label 쌍 로 구성되며 ,
- Unlabeled set : feature만 포함된 set
- Objective: 를 -dimensional simplex 에 mapping하는 Network 학습
- : Backbone network
- : Final linear-softmax classifier
Unified Objective
- 다음의 unified Objective function을 최적화
- : Labeled-set loss (e.g., multi-class cross-entropy)
- : Unlabeled-set loss
- : 각 loss term의 가중치
Learning Paradigms
Supervised Learning
- , labeled set만 사용
Semi-supervised Learning
- and , labeled와 unlabeled set 모두 사용
Self-supervised Learning
- 아래 두 단계로 나뉨
- Pretraining: , representation 학습에 집중
- Fine-tuning: , classifier 학습에 집중
- 아래 두 단계로 나뉨
Evaluated Methods
- 16개의 method를 평가함
- Supervised Methods: labeled data만 사용
- Sup: multi-class cross-entropy
- MixUp: mixup data augmentation
- SupCon: supervised contrastive learning
- Semi-supervised Methods: labeled와 unlabeled data를 동시에 사용
- Pseudo Label, Mean Teacher, MixMatch, FixMatch, FlexMatch, CoMatch
- Self-supervised Methods: labeled data 없이 학습
- SimCLR, MOCO (v2), SwAV, BYOL, SimSiam, DINO, Barlow Twins
- Supervised Methods: labeled data만 사용
3. RELATED WORK
Comparison to Oliver et al.
- Oliver et al. [62]는 큰 validation set을 사용하는 문제를 지적하며, 실험에서는 training set 크기보다 크지 않은 validation set을 사용
- Oliver et al.의 정의보다 큰 validation set을 사용하여 실제 적용 상황을 반영
- "1000 trials of Gaussian Process-based black-box optimization" 같은 자원 집약적 프로세스를 요구
Other semi-supervised benchmarks
- 다양한 semi-supervised learning 알고리즘을 비교
- 다양한 초기화와 unlabeled data의 내용이 semi-supervised method 성능에 미치는 영향 연구
- 다양한 self-supervised learning method를 포함하지 않아 비교의 한계 존재
Prior self-supervised benchmarks
- 여러 self-supervised benchmarks가 제안됨
- Goyal et al. [32]는 9가지 task을 다루는 benchmark 제안
- Ericsson et al. [27]은 40개 downstream task에서 13개의 self-supervised 모델 비교
- Da Costa et al. [23]는 다양한 downstream task과 데이터셋에 쉽게 적용 가능한 self-supervised method의 라이브러리 제안
Self-supervision for medical images
- Medical image classification를 위한 self-supervised method 평가 연구와 보완적 관계
- Multi-stage transfer learning pipelines 설계에 중점을 둠
Combining semi- and self-supervision
- 최근 연구에서는 semi-supervised와 self-supervised 학습 아이디어의 융합을 탐구
4. DATASETS AND TASKS
Datasets and Task
- 2D image로 구성된 네 개의 open-access medical image classification 데이터셋을 사용
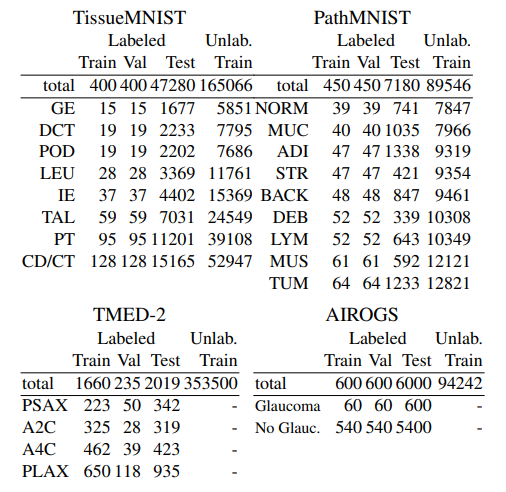
- PathMNIST와 TissueMNIST: MedMNIST collection에서 선택된 데이터셋, 28x28 해상도
- TMED-2: 112x112 해상도의 초음파 심장 image 데이터셋
- AIROGS: 384x384 해상도의 망막 image 데이터셋, 녹내장 여부를 결정하는 binary classification 과제
Data Splitting Strategy
- 실제 SSL 애플리케이션 조건을 반영하여 데이터 분할 전략을 수립
- Labeling 된 training과 validation set은 클래스의 자연 분포를 유지
- 실질적으로 더 큰 validation set을 사용하여 더 현실적인 평가 수행
PathMNIST
- 대장암 조직 슬라이드의 패치로 구성된 28x28 image 데이터셋, 클래스 불균형이 적음
- 총 900개의 image를 label링하여 training과 validation에 고르게 분할
TissueMNIST
- 신장 피질 세포 image로 구성된 28x28 image 데이터셋, 클래스 불균형 존재
- 총 800개의 image를 label링하여 training과 validation에 고르게 분할
TMED-2
- 초음파 심장 스캔의 112x112 2D 그레이스케일 image 데이터셋
- 다양한 뷰 타입을 classification하는 과제, 불균형한 데이터셋
AIROGS
- 망막 image로 녹내장 여부를 결정하는 binary classification 과제
- 1200개의 image를 label링하여 training과 validation에 고르게 분할, 클래스 불균형 존재 (9:1)
5. EXPERIMENTAL DESIGN
Performance Metric
- Balanced accuracy를 주요 성능 지표로 사용
- 개의 클래스가 있는 task에서 개의 example에 대한 실제 label을 , 예측된 label을 으로 표기
- 클래스 의 true positives 수를 로, 해당 클래스의 총 example 수를 로 표기
- Balanced accuracy (BA)를 다음과 같이 계산
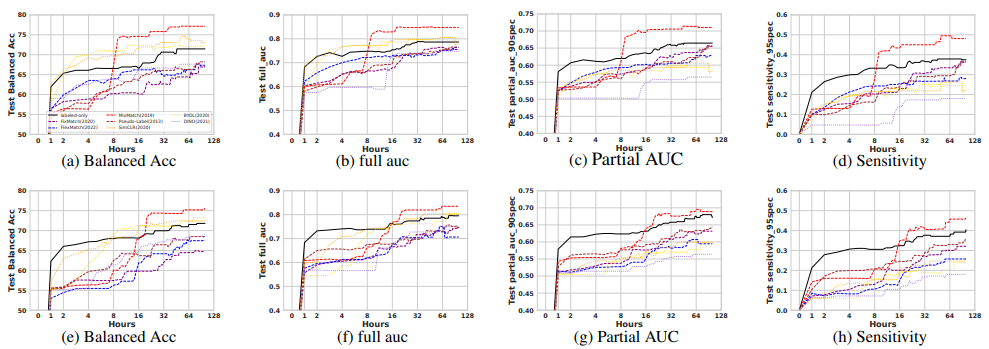
- AIROGS에서는 AUROC, partial AUROC, sensitivity-at-95%-specificity도 추가로 기록
Architectures
- CNN backbone 사용
- ResNet-18 [36]을 TissueMNIST와 PathMNIST에, WideResNet-28-2 [81]을 TMED-2에 사용
- AIROGS에서는 ResNet-18과 50을 실험
Training with Early Stopping
- 각 training phase에서 Adam optimizer와 cosine learning rate schedule을 사용하여 minibatch gradient descent 수행
- 최대 200 epochs 동안 학습, epoch마다 balanced accuracy를 validation set에서 기록
Hyperparameters
- Hyperparameters는 learning rate, weight decay, 등을 포함
Unified Procedure for Training and Hyperparameter Tuning
- 고정된 시간 내에 각 알고리즘을 평가
- NVIDIA A100 GPU 한 대를 사용하여 random search 수행
- 80,000 unlabeled example 당 약 25시간의 학습 시간 할당
Self-supervised Classification Phase
- Self-supervised method은 label 정보를 사용하지 않음
- 비교를 위해 각 self-SL method에 대해 추가적인 classification layer를 도입
Data Augmentation
- 모든 semi-supervised 및 supervised method에서 random flip 및 crop을 사용
- MixMatch는 MixUp [85], RandAugment [22]를 사용
- 각 self-supervised method 및 SupCon에서도 동일한 SimCLR augmentation을 사용
Multiple Trials
- 5개의 별도 trial (각기 다른 random seeds)에서 mean balanced accuracy 기록
6. RESULTS & ANALYSIS
Performance Metric
- 각 알고리즘의 test set balanced accuracy (5번의 시도 평균)를 측정함
- AIROGS 데이터셋에서는 AUROC, partial AUROC, sensitivity-at-95%-specificity도 추가로 측정
Hyperparameter Tuning
- Hyperparameter tuning은 실용적인 크기의 validation set에서도 효과적임
- 모든 알고리즘이 시간이 지남에 따라 성능이 향상됨
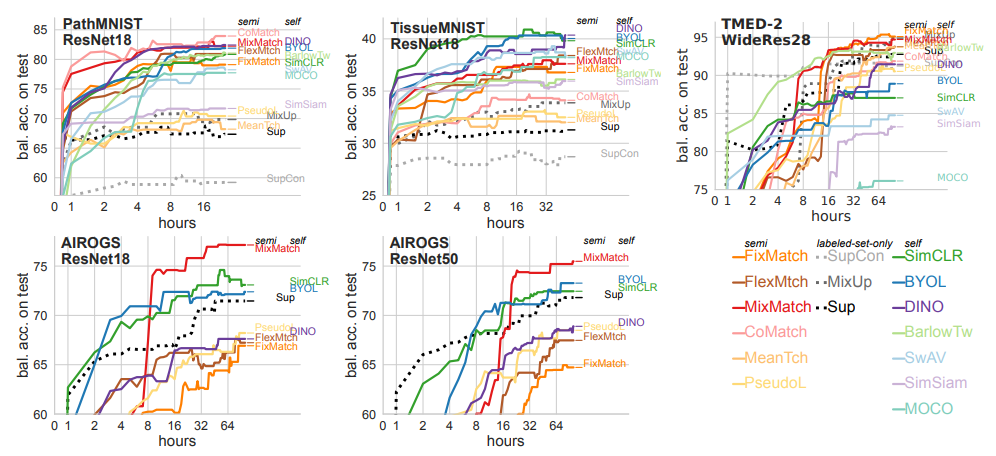
- 모든 알고리즘이 시간이 지남에 따라 성능이 향상됨
Best SSL Methods
- 데이터셋별로 최고 성능을 보이는 method
- Path: CoMatch, MixMatch, DINO, BYOL
- Tissue: DINO, BYOL, SimCLR
- TMED-2: MixMatch, FixMatch
- AIROGS: MixMatch, SimCLR, BYOL
Pretraining vs. From Scratch
- ImageNet에서 pretrained weights를 사용하는 것이 약간의 성능 향상이 있음
- 수렴 속도 향상 및 trial 간 변동성 감소
ResNet-18 vs. ResNet-50
- AIROGS에서 두 아키텍처의 성능이 유사함
- ResNet-50이 ResNet-18보다 실질적으로 더 나은 성능을 보이지 않음
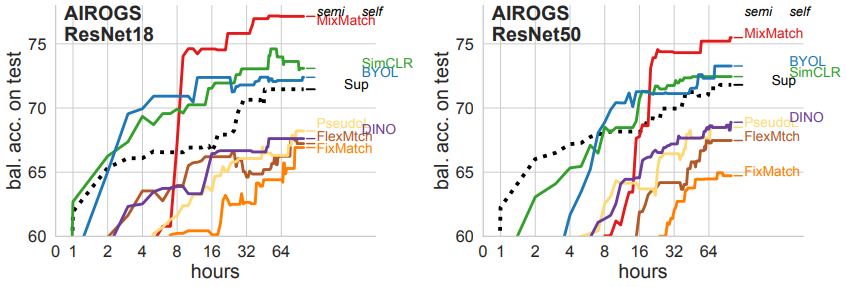
- ResNet-50이 ResNet-18보다 실질적으로 더 나은 성능을 보이지 않음
Tuning vs. Transferring from Another Dataset
- 제한된 label 데이터를 최대한 활용하기 위해 전체 label 셋을 학습에 사용하고 validation set을 사용하지 않는 method이 있음
- 사전 설정된 hyperparameters를 사용하여 성능을 평가
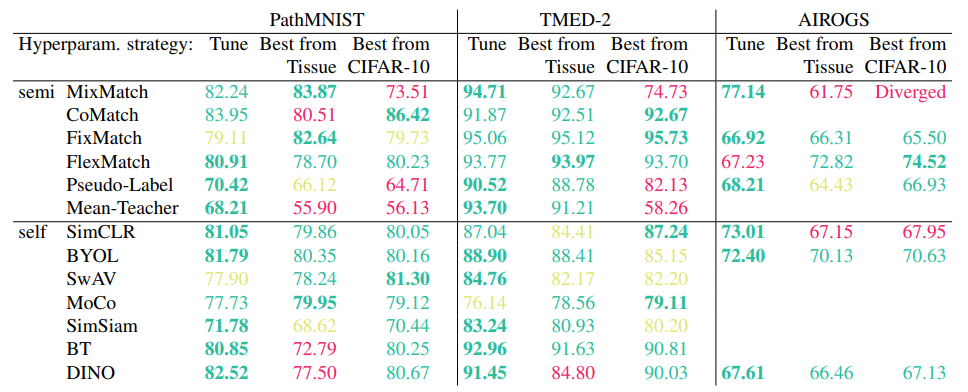
- 사전 설정된 hyperparameters를 사용하여 성능을 평가
7. DISCUSSION & CONCLUSION
Discussion & Conclusion
Benchmark Contribution
- 본 논문에서는 실용적인 benchmark를 제공하여 unlabeled data가 classification task에 기여할 수 있는 이점을 정량화하고, 이를 달성하는 데 도움이 되는 method들을 제시
- 경제적이고 현실적인 method으로 semi-supervised, self-supervised, supervised baselines의 학습 및 hyperparameter 선택 method를 통합하여 제공
Limitations
- 적은 수의 labeled examples (30-1000개)에 초점을 맞추었음
- 더 희귀한 label 상황 (예: zero-shot, few-shot)이나 더 세밀한 클래스가 있는 경우 다른 benchmark도 참조할 필요가 있음
- Labeled와 unlabeled 데이터의 분포가 크게 다른 상황을 구체적으로 연구하지는 않음
