[DB : Data] 의료 영상 데이터 (Medical Imaging Data)

▽ [DB : Data] 의료 영상 데이터 (Medical Imaging Data)
목 차
📂 1. DICOM 파일 구조 (Header vs Pixel Data)
🧩 2. 3D/4D 의료 영상의 데이터 처리 방식
🖥️ 3. PACS (Picture Archiving and Communication System)

의료 영상 데이터 구조.
📂 1. DICOM 파일 구조 (Header vs Pixel Data)
(1) DICOM이란?
-
Digital Imaging and Communications in Medicine
-
의료 영상 표준 포맷 : 전 세계 모든 병원/장비/AI 연구에서 사용.
-
특징 :
- 단순 이미지 파일(X-ray.jpg)이 아님.
- 의료 메타데이터 + 실제 영상 데이터를 함께 저장.
- 환자 진료 이력 추적, 장비 호환, 데이터 공유 가능.


(2) DICOM 구조.
DICOM 파일은 크게 두 부분으로 구성됩니다.
-
-
Header(메타데이터)
- (0010,0010) = 환자 이름, (0028,0010) = 행 개수 같은 코드(tag)로 저장
- 환자 정보: 환자 ID, 이름, 성별, 나이
- 검사 정보: 검사 날짜, 시간, 병원/의사 이름
- 장비 정보: CT/MRI 제조사, 파라미터 (Slice Thickness, kVp, TR/TE 등)
- 영상 정보: 해상도(512x512), 픽셀 크기, 이미지 위치
- 표준 태그(Tag): (Group, Element) 형태 예: (0010,0010) = Patient Name
👉 마치 "영상 설명서" 역할.
-
-
- Pixel Data (영상 데이터)
- 실제 영상 픽셀 값 (Grayscale Intensity).
- CT: HU(Hounsfield Unit, 조직 밀도 단위) 저장.
- MRI: MR 신호 강도 저장.
- X-ray: 픽셀 밝기 값 저장.
- Pixel Data (영상 데이터)

(3) 예시.
-
DICOM Tag (0010,0010) → Patient Name = 홍길동
-
DICOM Tag (0028,0010) → Rows = 512
-
DICOM Tag (0028,0011) → Columns = 512
-
Pixel Data → 512×512 행렬(숫자값: 조직 밀도).

(4) AI에서 중요한 이유.
- 단순히 '이미지 픽셀'만 쓰는 것이 아니라,
- 환자 메타데이터 (나이, 성별, 검사 조건)
- 영상 파라미터 (슬라이스 두께, 픽셀 크기)도 함께 분석해야 정확한 AI 모델 생성 가능.

🧩 2. 3D/4D 의료 영상의 데이터 처리 방식.
(1) 2D vs 3D vs 4D
-
2D : 단일 슬라이스 ( ex: 한 장의 X-ray)
-
3D : 다수의 슬라이스를 모아 '볼륨 데이터' 생성 (ex : CT/MRI)
- 픽셀(pixel)이 2D 한 점이라면, 복셀(voxel)은 3D 한 점.
-
4D : 시간축까지 포함된 데이터(ex: 심장 MRI 시퀸스, 초음파 동영상)
👉 쉽게 말해,
2D = 사진
3D = 입체 모형
4D = 움직이는 입체 모형(영상)

(2) 3D 데이터 처리.
-
3D 의료 영상은 'Voxel(Volume + Pixel)' 단위로 저장됨.
- Pixel: 2D 이미지에서 한 점.
- Voxel: 3D 공간에서 한 점 (x,y,z 좌표 + 값).
- 처리 방식 :
- Resampling: 서로 다른 슬라이스 두께/해상도를 표준화.
- Segmentation: 종양/장기 영역을 3D로 구분.
- Visualization: 2D 슬라이스, MPR(Multi-Planar Reconstruction), 3D Rendering.

(3) 4D 데이터 처리.
-
시간(Time)을 포함하는 3D 시퀸스.
-
Ex:
- 심장 MRI (수축~이완 과정을 시간별 3D Volume으로 기록).
- 초음파 영상(프레임 연속).
-
AI 과제.
-
Spatio-temporal Analysis (시공간 분석).
-
예: 심장 운동 기능 추적, 암 성장률 예측.
-

🖥️ 3. PACS (Picture Archiving and Communication System).
(1) 개념.
-
의료 영상 데이터를 저장, 검색, 공유하는 시스템.
-
모든 병원 영상 센터의 핵심 인프라.
-
CT/MRI 촬영 → PACS 서버 저장 → 의사/AI 시스템에서 조회.

(2) 구성 요소.
-
-
Image Acquisition (획득)
- CT, MRI, X-ray 장비에서 DICOM 생성.
-
-
-
PACS Server (저장/관리)
-
DICOM 파일 저장소.
-
Database + Storage System.
-
-
-
-
Workstation (조회/분석)
-
의사가 영상 확인.
-
AI 모듈이 접속해 분석 수행.
-
-
-
-
Network/Integration(네트워크 연결)
- HL7/FHIR과 연결 → 환자 진료 기록과 매칭.
-

(3) 실무 포인트.
- 영상이 수천 장, 수 GB → 스토리지 및 압축 기술 중요.
- 병원 내 보안 규제 (HIPAA, GDPR).
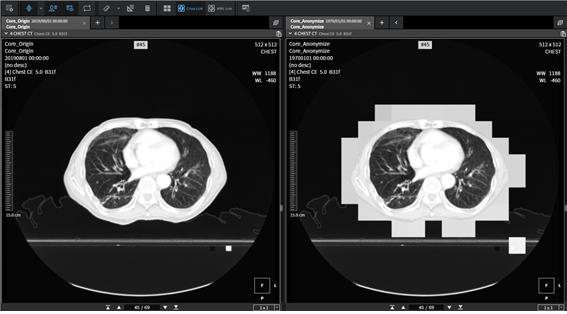
- AI 연구자는 PACS에서 데이터를 가져와 익명화(De-identification) 후 학습에 사용.

의료 영상 데이터 전처리 & 학습 방식.
전처리-모델 학습 및 검증-배포 및 평가 까지의 전체 요약.
데이터 수집 → 익명화/검증(QC) → 물리단위·해상도 정리(Resample/Registration)
→ 강도 표준화(Intensity normalization / windowing / stain normalization) → 메모리 친화적 분할(Patch/Tiling)
→ 균형 잡힌 샘플링 + 고품질 레이블 처리 → 모델 설계(2D/3D/Transformer 등) → 학습(optimizer, LR, AMP, DDP)
→ 검증(lesion-level / patient-level) → 불확실성/해석/모니터링 → 배포(경량화, 규제준수).

1) 🖥️ 데이터 수집·인입(ingest) & 품질검사(QC).
의미.
데이터 파이프라인의 맨 앞단.
- 잘못 수집되거나 품질 낮은 데이터가 들어오면 후속 전처리 * 학습이 모두 무용지물.
- 의료영상에서는 특히 장비 & 프로토콜이 다양해 '데이터 불일치(domain shift)'가 흔함.
핵심 개념
- 메타데이터 : DICOM 헤더(환자,장비,촬영 파라미터)
- 무결성 : 파일 손상, 누락 슬라이스, 잘못된 인코딩 탐지.
- 익명화(De-identification) : PHI(환자식별정보) 제거.
- 기초 QC 지표 : 픽셀 분포, 해상도, 방향(orientation), 슬라이스 간격.
절차 (실무 단계별)
-
- 원본 수집.
- PACS/DICOM export, 연구용 DB, 공개 데이터셋 수급.
- 원본 수집.
-
-
무결성 체크.
- 파일이 정상적으로 열리는지, 읽기 오류 발생 여부 확인.
- ex) 파일 CRC/해시 확인, pydicom으로 읽어보기.
-
-
- 메타데이터 추출 * 정리.
- 필수 태그 추출:
- PixelSpacing, SliceThickness, ImageOrientationPatient,
- ImagePositionPatient, RescaleSlope, RescaleIntercept,
Modality, Manufacturer.
- 필수 태그 추출:
- 메타데이터 추출 * 정리.
-
- 익명화
- DICOM 헤더의 PHI 삭제(병원 규정 준수).
- 이미지 상에 그려진 텍스트(burned-in text)는 OCR로 탐지·블러/마스킹.
- 익명화
-
-
기본 QC 파이프라인 실행.
- 픽셀값 히스토그램(분포), 평균/표준편차, 최소/최대값.
- 해상도(행×열), 채널 수(흑백/RGB), 슬라이스 개수 점검.
- 누락 슬라이스/중복 슬라이스 탐지.
-
-
- 로그 * 메타데이터 저장.
- 수집원, 장비, 프로토콜, QC 결과 저장(추후 분석, 도메인 시프트 조사에 필요).
- 로그 * 메타데이터 저장.
도구/예시.
-
라이브러리: pydicom, SimpleITK, nibabel, dicom-numpy
-
익명화 툴: pydicom으로 태그 삭제, DICOMCleaner 등
-
QC 자동화: custom script + pandas로 메타데이터 테이블 관리
실무 팁 & 함정.
-
Burned-in text: 헤더 익명화만 하면 안 됨—이미지에 환자명이 찍혀있을 수 있음.
-
장비별 표기 차이: 동일 태그가 없거나 다른 위치에 저장되는 경우가 많음—태그 맵핑이 필요.
-
로그 남기기: 어떤 파일를 왜 제외했는지 기록해두지 않으면 나중에 문제 원인 규명이 어려움.
DICOM → 표준화 파일(NIfTI 등)
-
DICOM 헤더에서 PixelSpacing, SliceThickness, ImageOrientationPatient, PatientPosition, RescaleIntercept/RescaleSlope 등 필수 파라미터를 뽑아 둡니다.
-
CT의 경우 픽셀값 → HU 변환(RescaleSlope/Intercept 적용) 확인 필수.
익명화(De-identification)
-
DICOM 태그(PatientName 등) 제거 + burned-in text(이미지에 찍힌 환자명) 탐지/마스킹.
-
연구·배포시 로그에 익명화 버전·해시 남기기.
자동 QC 파이프라인(권장)
-
결측 슬라이스, 이상 해상도, 이미지 방향(좌우/상하 뒤바뀜), 너무 큰 결손영역(비어있는 이미지) 탐지.
-
통계적 이상치 탐지: 픽셀값 분포(히스토그램), 평균/표준편차, 영상 대비비(contrast) 확인.
메타데이터 표준화
- 수집 병원/장비(모델, 소프트웨어 버전)을 컬럼으로 저장 → 이후 도메인시프트 분석에 필수.

2) 🖥️ 공간·물리적 정규화 (Registration / Resampling / Cropping).
의미
서로 다른 장비/프로토콜에서 온 영상들을 "공통의 물리 좌표계와 해상도"로 맞추는 작업.
- 3D 볼륨의 종류나 슬라이스 두께가 다르면, 모델이 학습하기 어려움.
핵심 개념.
-
Voxel spacing: 픽셀이 실제 물리공간에서 차지하는 거리(mm).
-
Resampling: voxel spacing을 표준화(예: 1×1×1 mm)하는 보간(interpolation).
-
Registration: 한 영상(또는 시점)을 다른 영상과 정렬(위치·회전 보정).
- Rigid→Affine→Deformable(Non-rigid).
절차.
-
- 공간정보 확인
- (PixelSpacing, SliceThickness, ImageOrientationPatient, ImagePositionPatient)
- 공간정보 확인
-
- Resample to isotropic voxels (권장: task-dependent, 예: 1mm)
- 보간 방법: 이미지 → linear/bspline, 라벨(마스크) → nearest neighbor.
- Resample to isotropic voxels (권장: task-dependent, 예: 1mm)
-
- Registration (필요 시)
- 같은 환자의 다른 modality(CT↔MRI)나 시간축 데이터 정렬 시 적용.
- 단계: rigid → affine → non-rigid(유연)
- Registration (필요 시)
-
- Cropping/ ROI extraction
- Body mask 생성(예: threshold) → 불필요한 공백 제거 → 연산량 감소.
- Cropping/ ROI extraction
도구.
- SimpleITK, ANTs, elastix, SimpleElastix, niftyreg, VoxelMorph(딥러닝 기반 비지도 정합)
팁 & 함정.
-
라벨 보간 주의: label을 float 보간하면 레이블 값이 깨짐 → 반드시 nearest 사용.
-
Resample 성능저하: 너무 작은 spacing으로 다운샘플링 하면 정보 소실, 너무 촘촘하면 메모리 폭증.
-
Registration 실패 탐지: 유사성 지표(NCC, MI)로 체크.
왜 필요한가?
- 서로 다른 장비와 프로토콜(예: CT slice thickness 1mm vs 5mm)은 비교 불가
→ 모델 성능 저하.
기본 작업.
-
- Resample to isotropic spacing (권장 1mm 혹은 task-dependent)
-
SimpleITK.Resample() 등 사용.
-
주의: CT는 HU 기반 → 보간(method)에 따라 값 왜곡 주의
(선형보간 권장 for intensities; nearest for labels).
-
- Registration(multi-modal/longitudinal).
- rigid → affine → non-rigid 순. ANTs, SimpleElastix, elastix 사용.
-
- Cropping/ROI Extraction.
- 자동화: 바디-구역 마스크(Bounding box by threshold)
→ 불필요한 공백 제거.
실무 팁.
- MRI 상호 비교 시 bias-field(조도 불균형) 보정(N4BiasFieldCorrection) 먼저.

3) 🖥️ 강도 표준화 (Intensity normalization & windowing).
의미.
동일한 조직이라도 장비/설정에 따라 픽셀값(강도)이 달라집니다.
모델이 이런 차이를 “노이즈”로 인식하지 않게 강도 분포를 표준화해야 합니다.
핵심 개념.
CT(HU 기반)
-
HU 단위 그대로 사용하거나, 'task-specific windowing 사용'
- (ex: lung window: -1000~400, bone: 300~2000)
-
이후 min-max (e.g. -1000..400 → 0..1) 또는
z-score(normalize by mean/std of lung region).
※ HU(Hounsfield unit).
CT의 구동원리.
-
- CT는 기본적으로 X-ray라는 방사선을 사용하며, 촬영의 대상이 되는 물체에
X-ray Tube(Source)로 부터 나오는 X선을 투과시켜 검출기(Detector)에서 투과된 수치를 측정.
- CT는 기본적으로 X-ray라는 방사선을 사용하며, 촬영의 대상이 되는 물체에
-
- Source에서 방출한 X선의 양과 Detector에서 검출한 X선의 양의 차이를 전기적 신호로 처리
-
- 물질에 따라 감쇠된 방사선량을 선형 감쇠 계수(linear attenuation coefficient, μ)로서 나타넴.
HU(Hounsfield unit).
-
여기서 Hounsfield unit은 앞서 설명한 선형 감쇠 계수를 위와 같은 수식에 근거하여 선형적 변환을 적용한 값이며,
-
표준 대기압 및 온도(STP)에서 증류된 물(Distilled Water)을 기준(HU=0)으로 삼아 여러가지 물질들에 대해 상대적인 값을 정의해 놓은 것.
-
임상에서의 CT 영상은 HU의 범위가 -1024 ~ +3071까지이며, 512x512 = 262,144개의 픽셀로 구성.
-
CT 영상의 정확도는 환자선량과 픽셀의 크기에 지배적인 영향을 받으며, CT 장치에 따른 영향은 거의 받지 않는다.
-
쉽게 설명하면, X-ray beam이 물체를 통과할 때, beam이 물체에 얼마나 흡수되는지를 나타낸다.
-
주로 사람의 몸에 대한 X-ray 영상을 취득할 때,
각 조직(tissue)의 밀도(density)를 표현하는 단위로 사용된다.
- X-ray beam이 많이 흡수될수록 (즉, density가 높을수록) HU값이 크고,
적게 흡수될수록 (즉, density가 낮을수록) HU값이 작다.


MRI.
- 값이 장비·파라미터에 따라 다름
→ z-score per volume 또는
-> Nyul histogram matching / WhiteStripe 사용.

WSI(병리)
- 염색(stain) 색상 불균일 → stain normalization (Macenko, Reinhard) 적용.

Ultrasound
- 클리핑, log-transform, speckle noise reduction(비선형 필터) 고려.

요약 규칙
"같은 스케일/분포"로 맞춰라 → batch 간 차이 완화.

4) 🖥️ 라벨(Annotation) 품질관리 및 처리.
라벨 유형.
- image-level (진단/양성/음성), box-level, pixel-level (mask), landmark/contour
라벨 노이즈 & 불확실성.
-
다수 의사 레이블이 상이할 때:
- majority voting, soft label(확률 라벨),
- 라벨 가중치(신뢰도 기반) 사용.
-
Inter-rater variability 측정(일반적으로 Cohen’s Kappa, Fleiss’ kappa).
어노테이션 워크플로우.
-
기초 레이블 → 2) 리뷰(2nd reader) → 3) Adjudication(논쟁 샘플에 대해 전문가 패널).
-
라벨 메타데이터(annotator id, timestamp, confidence)를 반드시 보관.
약한 레이블(weak supervision) 활용.
-
Multiple Instance Learning (MIL)
— WSI 같은 경우 image-level label만 있을 때 patch-level MIL 모델 사용. -
Pseudo-labeling & self-training: 모델이 예측한 강한 확신 예측을 라벨로 추가.

5) 🖥️ 데이터 분할(Train/Val/Test) & 샘플링 전략.
환자 단위 분할(patient-wise split):
- 동일 환자(혹은 동일 study)에 속한 여러 이미지가 train/test에 걸쳐지지 않도록 주의.
(데이터 누수 방지).
층화(stratified) 분할:
- 질병 유무, 중증도, 기관(병원)별 층화 권장.
교차검증(cross-validation)
- 소규모 데이터에서는 k-fold 또는 nested CV 사용(하이퍼파라미터 튜닝 시 leakage 주의).
Patch sampling for small lesions
-
Positive patch oversampling:
- lesion이 희소한 경우 positive-containing patches를 더 자주 샘플링.
-
Hard-negative mining:
- 학습 후 오탐(존재하지 않는데 양성으로 예측)을 샘플링해 추가 학습.

6) 🖥️ 데이터 증강(Data Augmentation) — 실제로 성능을 좌우.
Geometry
-
rotation
-
flip(주의:좌우 반전은 좌/우 정보가 중요한 경우 사용 금지)
-
scaling
-
elastic deformation(특히 병리 조직).
Intensity
-
brightness
-
contrast jitter
-
gausian noise
-
gamma transform
Modality-specific.
-
CT: realistic noise modeling (Poisson), random windowing.
-
MRI: simulate bias field, k-space augmentations (고급).
-
WSI: stain perturbation (H&E color jitter), stain augmentation.
Advanced.
-
MixUp / CutMix (classification), RandAugment / AutoAugment 자동탐색.
-
GAN / Diffusion 로 synthetic data 생성(단, 임상 신뢰성 검증 필요).
Test-time augmentation(TTA).
- 평균/투표 기반으로 inference 결합 → 성능 향상.

7) 🖥️ 모델 아키텍처 선택(문제별 요약).
Classification (image-level):
-
ResNet family, EfficientNet, DenseNet, SWIN/Vit (빠르게 뜸)
-
Transfer learning(Imagenet) 초기화 후 fine-tune 추천(특히 데이터 적을 때).
Detection:
- Faster R-CNN, RetinaNet (Focal loss), YOLOv8, DETR 계열.
Segmentation
-
2D: U-Net, Attention U-Net, DeepLabV3+
-
3D: 3D U-Net, V-Net, nnU-Net(자동설정의 강력한 baseline)
-
Transformer 계열: UNETR, Swin-UNET/TransUNet (멀티스케일 학습에 강함)
Registration:
- VoxelMorph (unsupervised), ANTs (classical)
Sequence / Temporal:
- ConvLSTM, 3D+Time CNNs, Video Transformer

8) 🖥️ 2D vs 3D 선택 기준 & 실무 팁.
3D 장점:
- 공간 맥락(종양 볼륨) 고려 → 더 높은 성능 가능
3D 단점:
-
메모리·연산 비용 큼 → 작은 batch size → BN 문제
-
대안: patch-based 3D training, gradient accumulation, GroupNorm / InstanceNorm 사용.
2D slice-based 접근
-
장점: 경량, pretraining(Imagenet 사용) 쉬움.
-
단점: 단편적 맥락 손실 → post-processing으로 3D 연결 필요.
권장
-
작은 병변(예: 폐 결절): 3D 패치 학습(+sliding-window inference)
-
전역/전신 구조 문제: 2D 큰 field-of-view approaches or hybrid (2.5D stacks).

9) 🖥️ 손실함수 / 불균형 처리.
Classification: Cross-Entropy, Focal Loss(심한 클래스 불균형), Label Smoothing
Segmentation: Dice Loss, Soft Dice, BCE + Dice, Tversky/Focal-Tversky(for small lesions)
Detection: Focal Loss(포지티브 희박), IoU loss variants
Sampling: class-weighted sampling, over/undersampling, focal loss 같이 사용 권장.

10) 🖥️ 학습 설정 & 최적화 팁.
Optimizer:
- AdamW (weight decay 분리), SGD+momentum (큰 데이터셋), Ranger (AdamW + Lookahead) 실험적.
LR 스케줄러:
- CosineAnnealing, OneCycleLR, ReduceLROnPlateau
Mixed Precision:
- AMP (automatic mixed precision)로 메모리·속도 최적화.
Distributed Training:
- PyTorch DDP 권장(DDP는 DataParallel보다 안정적).
BatchNorm 문제(작은 batch):
- GroupNorm/InstanceNorm 사용.
Checkpointing:
- 주기적(가장 좋은 val metric) + 최근 N개 보관, 체크포인트에 RNG 상태·epoch 저장.
추천 시작 레시피(예: 2D U-Net segmentation):
-
Patch size: 256×256
-
Batch size: 8~32 (GPU 메모리에 맞게)
-
Optimizer: AdamW lr=1e-4 weight_decay=1e-5
-
Scheduler: CosineAnnealingWarmRestarts or ReduceLROnPlateau
-
Epochs: 200~400 (early stopping on val Dice)
-
Augmentation: geometric + intensity + elastic.

11) 🖥️ 검증·평가(Validation & Metrics).
데이터 분리: 환자 단위로 Train/Val/Test. 시간순 분할 검토(시계열 환자 패턴 있을 경우).
Metrics:
-
Classification: AUC-ROC, AUPRC(불균형일 때 중요), Accuracy, F1
-
Segmentation: Dice coefficient, IoU, Hausdorff distance(경계 품질)
-
Detection: mAP, FROC (lesion-level sensitivity at specific FP per image)
Lesion-level vs Pixel-level 평가
- 병변 검출에서는 lesion-level sensitivity (어떤 lesion을 잡았는지가 중요) — 단순 pixel IoU가 충분하지 않을 수 있음.
Calibration
-
모델 확률의 해석 가능성: Calibration curve, Expected Calibration Error(EC).
-
온도 스케일링(Temperature scaling)으로 보정.
통계적 유의성
-
부트스트랩(bootstrap)으로 CI(신뢰구간) 계산.
-
모델 비교 시 paired tests (DeLong test for ROC 등).

12) 🖥️ 불확실성(uncertainty) & 안전성.
불확실성 추정
-
Epistemic(모델 불확실): Deep Ensembles, MC-Dropout
-
Aleatoric(데이터 불확실): Predictive variance (heteroscedastic loss)
OOD(Out-of-distribution) 탐지:
- Mahalanobis distance, ODIN, density-estimation methods.
의료적 안전장치
- ‘작동 범위(operational envelope)’ 명시: 어떤 환자·장비에서 모델이 유효한지 문서화.

13) 🖥️ 해석가능성(Explainability).
도구:
- Grad-CAM/Grad-CAM++,
- Integrated Gradients,
- SmoothGrad,
- SHAP(특징기반)
임상의와의 소통:
- Saliency map을 단순히 보여주는 것만으로는 부족
→ 임상 사용성 관점에서 false-positive 원인/false-negative 영향 해석 필요.
