U-Net: Convolutional Networks for Biomedical Image Segmentation 논문을 기반으로 작성되었습니다.
[Background info]
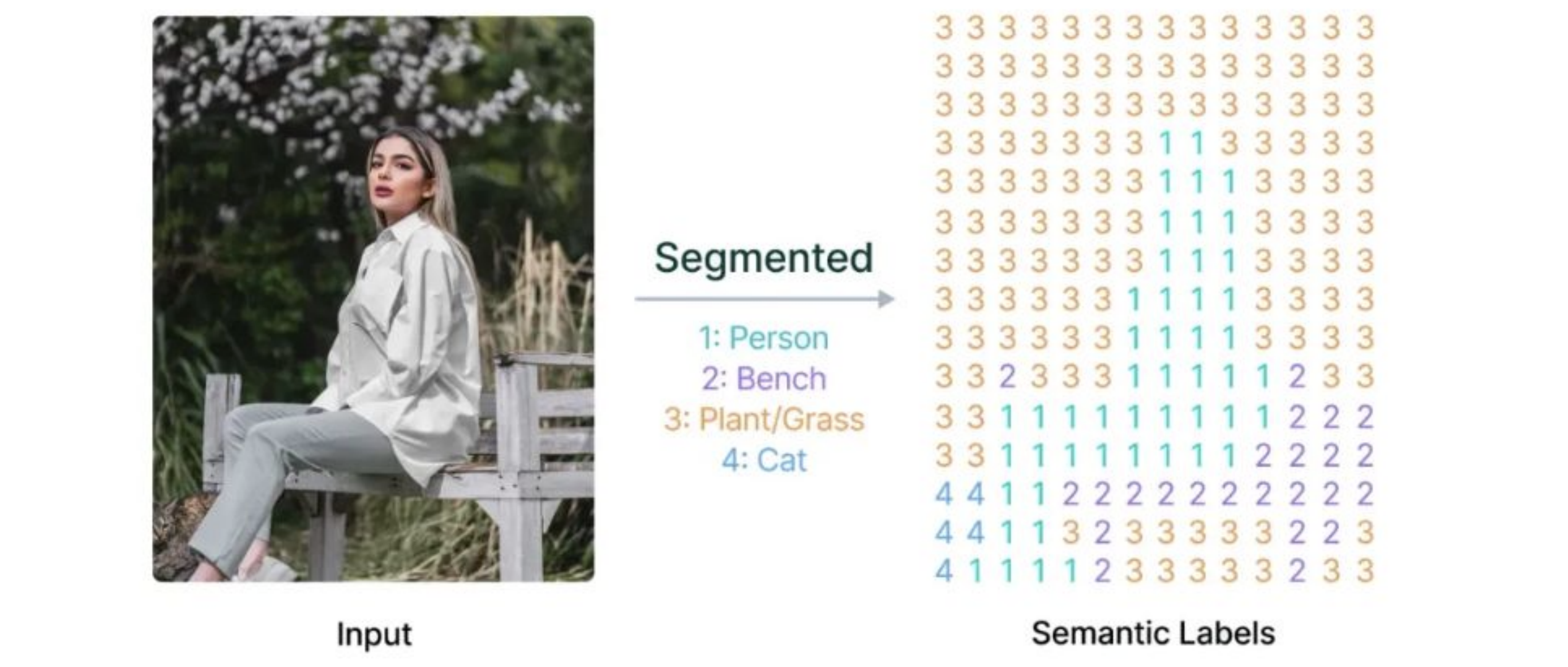
Segmentation
> 입력 이미지의 모든 픽셀에 대해 Classification해, 각 픽셀이 어느 클래스에 속하는지 라벨 할당 = 분할
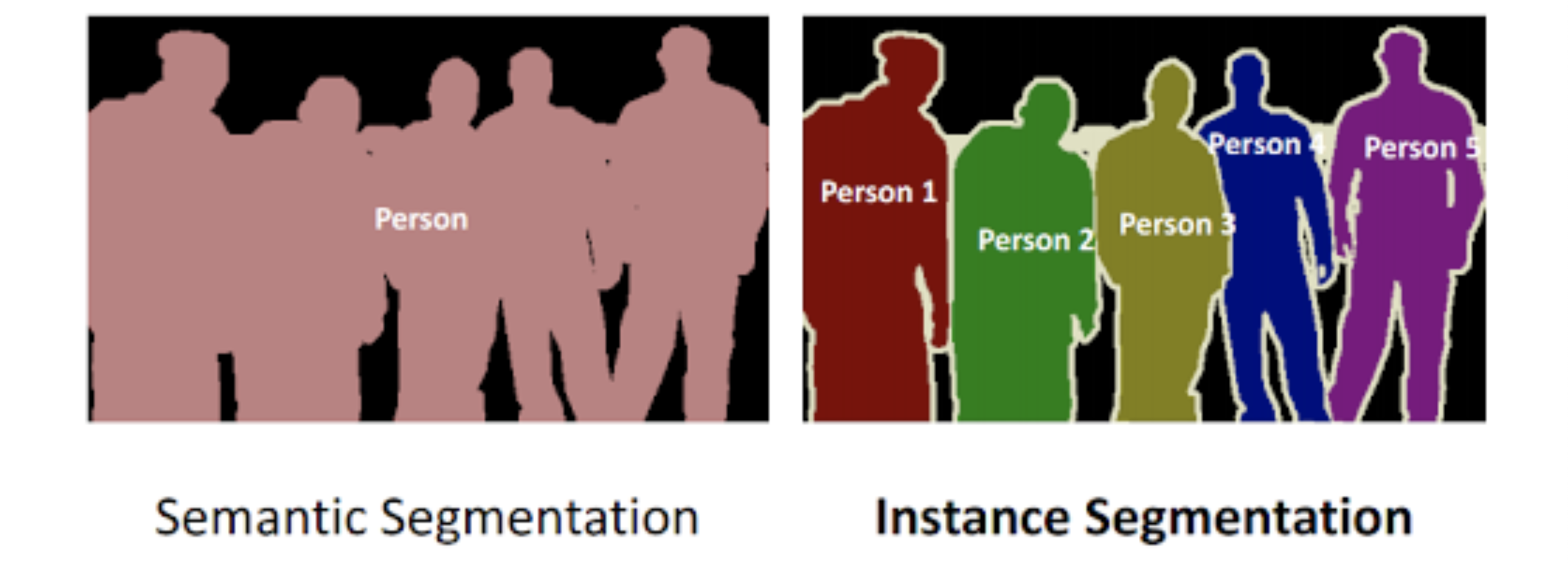
Semantic vs Instance (Segmentation)
- Semantic segmentation: 같은 클래스 내부에서 객체 구분 불가능
- Instance segmentation: 같은 클래스 내부에서 객체 구분 가능
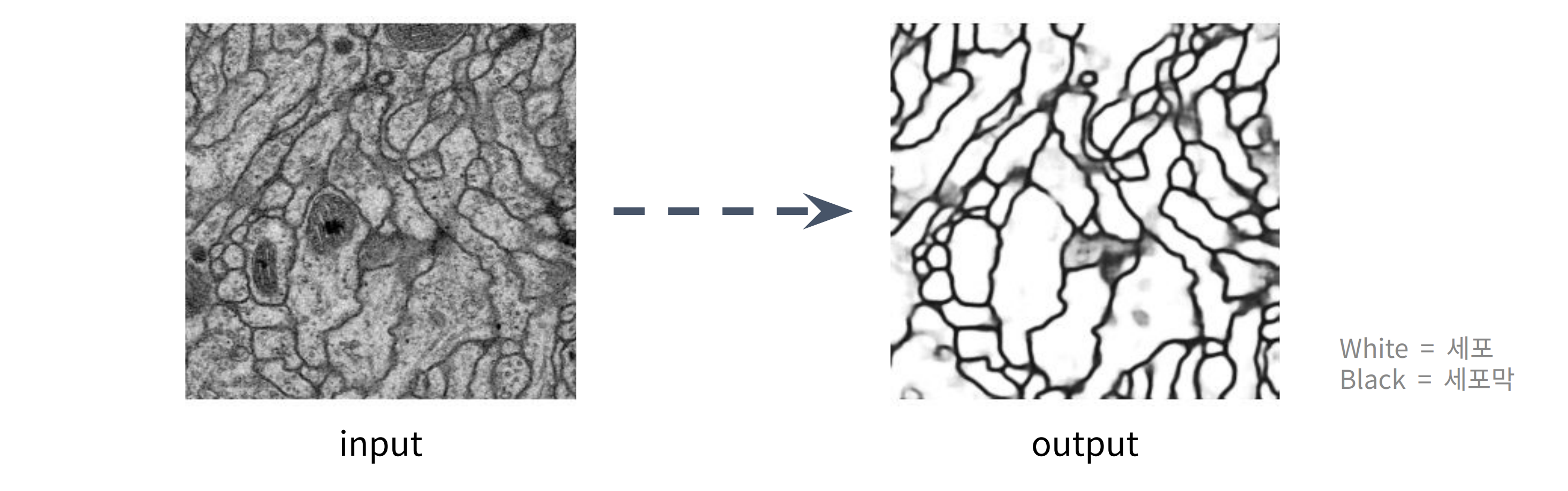
Biomedical Image Segmentation
- 의료 또는 생물학 연구에서 얻은 영상(X-ray, CT, MRI, 현미경 사진 등) 내의 특정 구조물(종양, 세포 등)을 픽셀 단위로 정확하게 식별하고 경계를 나누는 기술
- U-Net에선 주로 세포 구조를 분할
- 현미경 영상과 같은 미세 구조를 다루는 영상에서 픽셀 단위의 Segmentation이 중요
1. Introduction
Before U-Net
CNN + Sliding-Window
- VGGNet처럼 conv layer들을 거친 후 최하단에 Fully-Connected Layer를 놓아 Classification 하는 전통 CNN 모델을 픽셀 단위로 적용
- 이미지의 한 픽셀 주변값(patch)을 입력으로 받아, patch 중앙의 한 픽셀 클래스를 출력(부여)하는 방식
- 모든 픽셀마다 네트워크가 독립적으로 수행돼 속도가 느리고, 각 패치마다 겹치는 부분이 많아 중복으로 인한 낭비가 심함
- 공간 context를 확보하기 위해 patch 크기를 늘리면 더 많은 max pooling을 거쳐야하므로 localization 정확도가 떨어짐 (trade-off)
FCN: Fully Convolutional Network
- 기존 방식을 완전히 개편하여 FC Layer를 없애고 모두 1x1 conv layer로 대체해, 이미지 전체를 입력으로 받아 전체 공간 특징맵(segmentation)을 출력하는 모델
- Semantic Segmentation을 위해 제안됨
- 이 Base model을 수정, 확장하여 제안된 모델이 U-Net
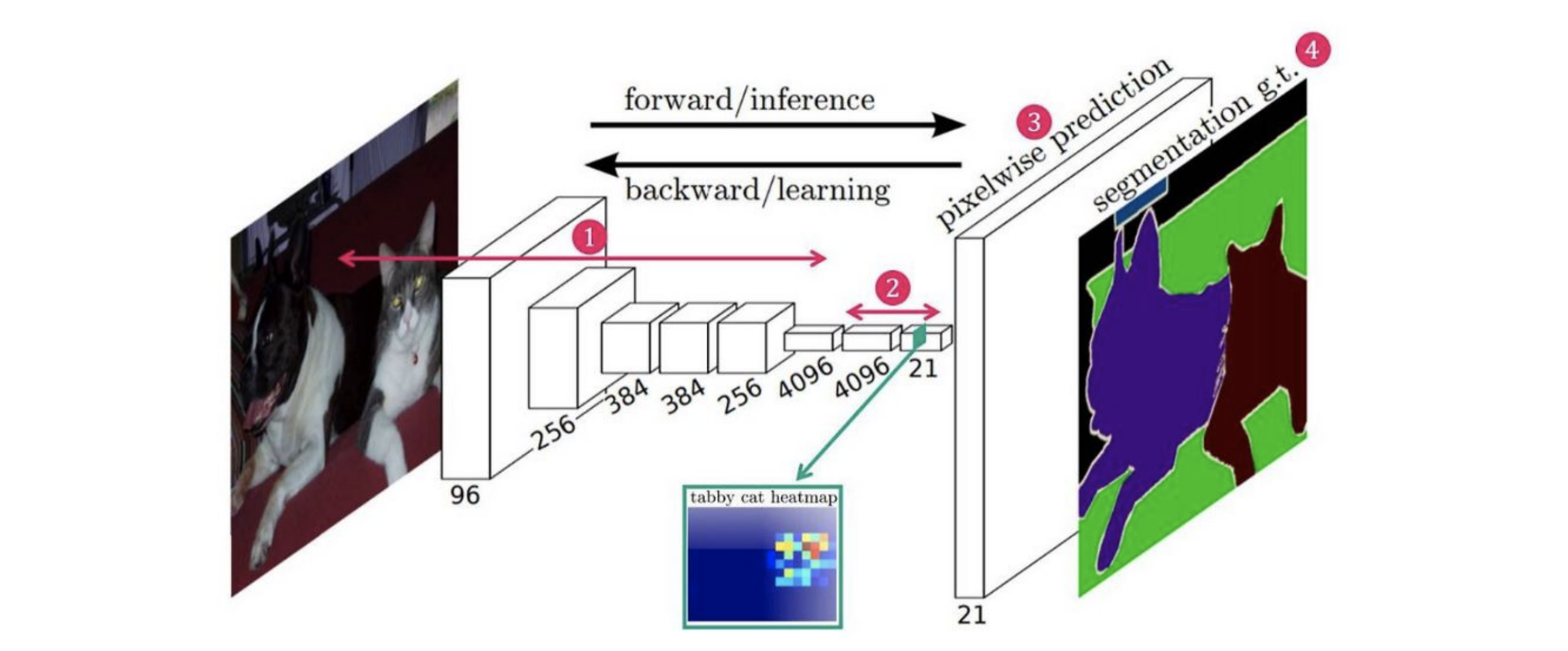
Idea of U-Net
- FCN보다 업샘플링 과정을 더 많이 가져가, U자형 대칭 인코더-디코더 구조
- 고해상도 특징의 연결을 위한 Skip Connection
- Transposed Convolution(Up-Convolution)을 통한 업샘플링
- Overlap-Tile, Mirroring
- Weighted Loss, Data augmentation
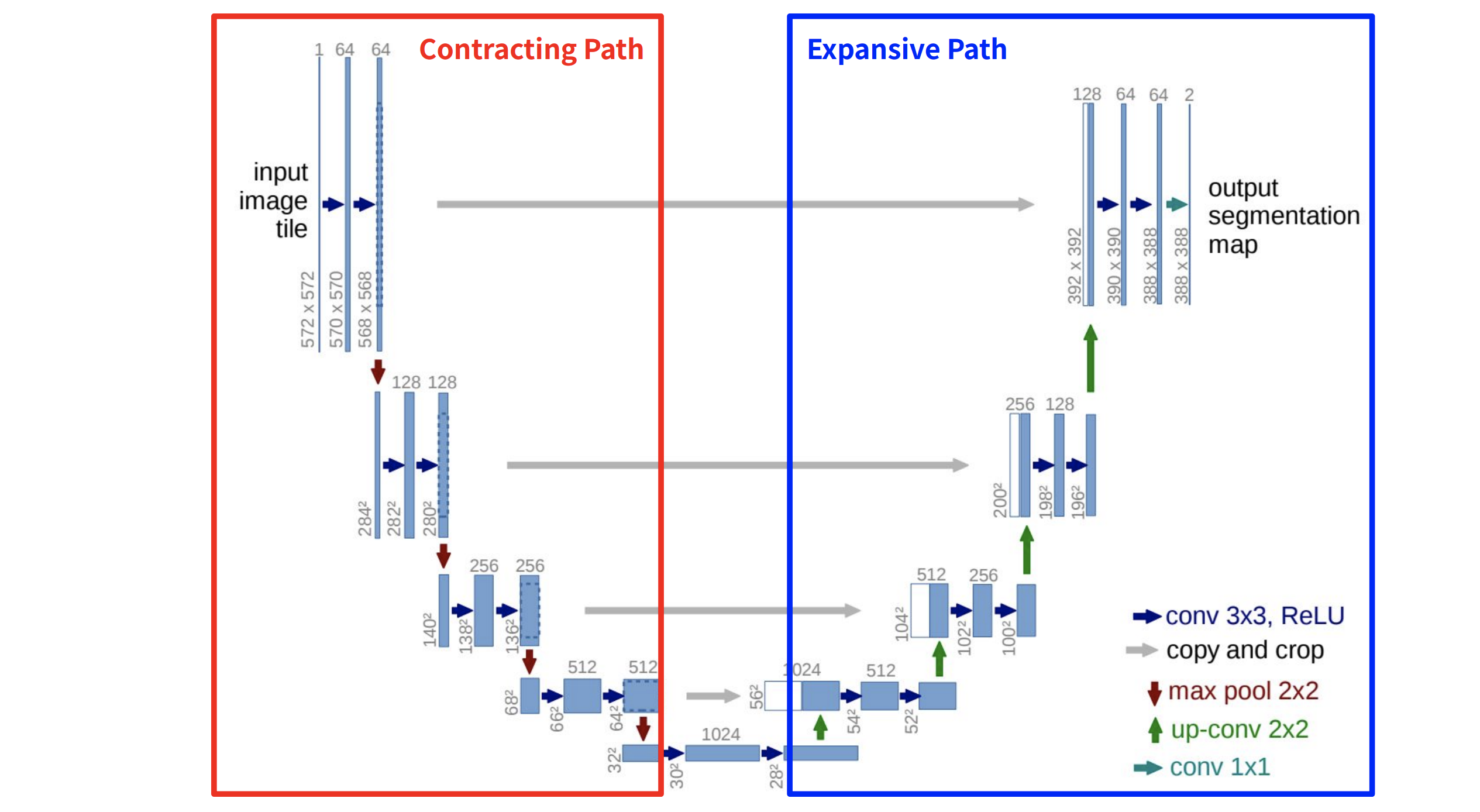
2. Network Architecture
Architecture
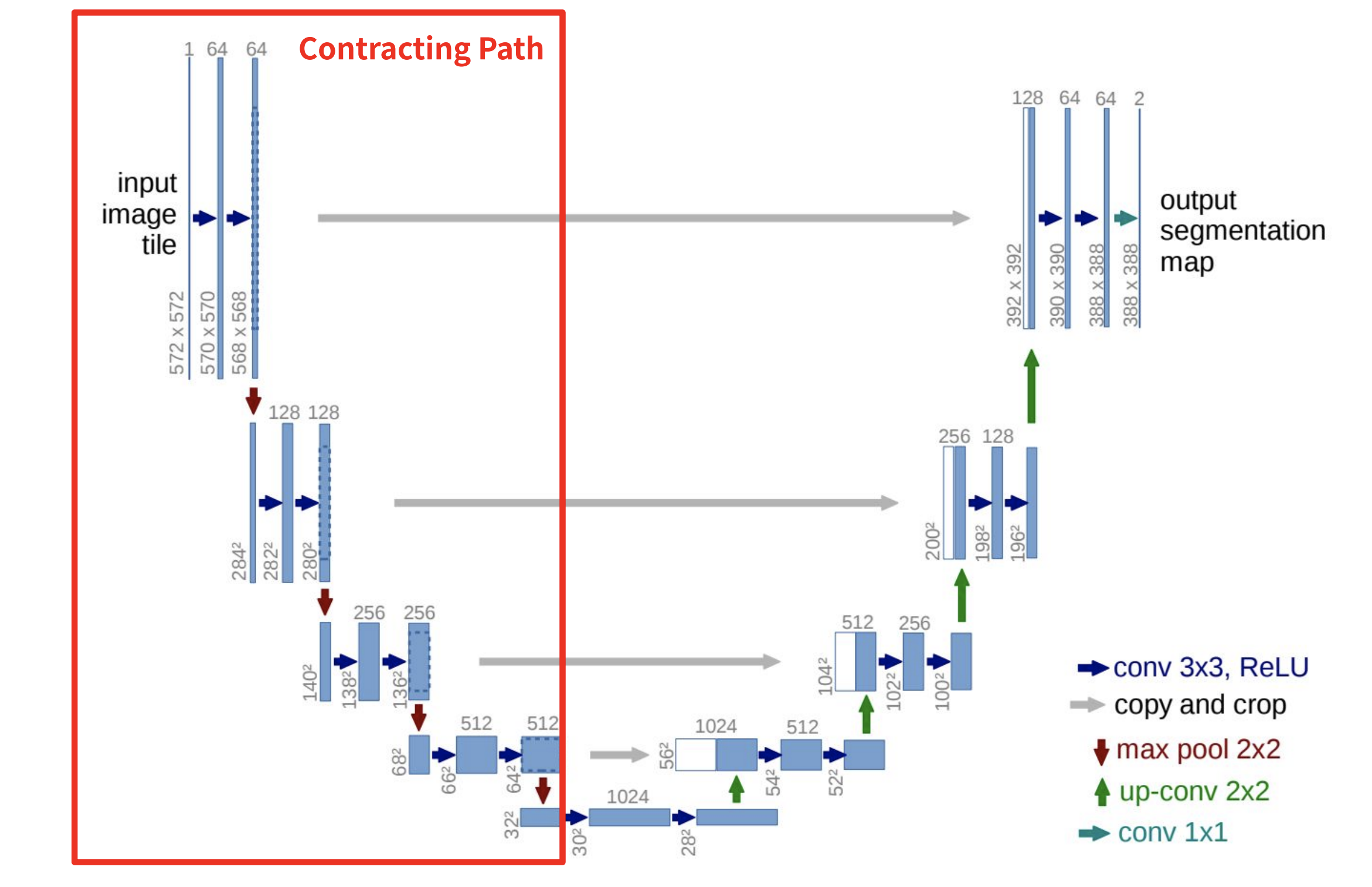
- Contracting Path
- 각 step 마다 3x3 conv를 두 차례식 반복 (패딩x)
- conv layer에는 ReLU 연산이 포함
- conv 이후엔 2x2 max-pooling (stride=2)
- Down Sampling마다 채널 수를 2배로 늘림
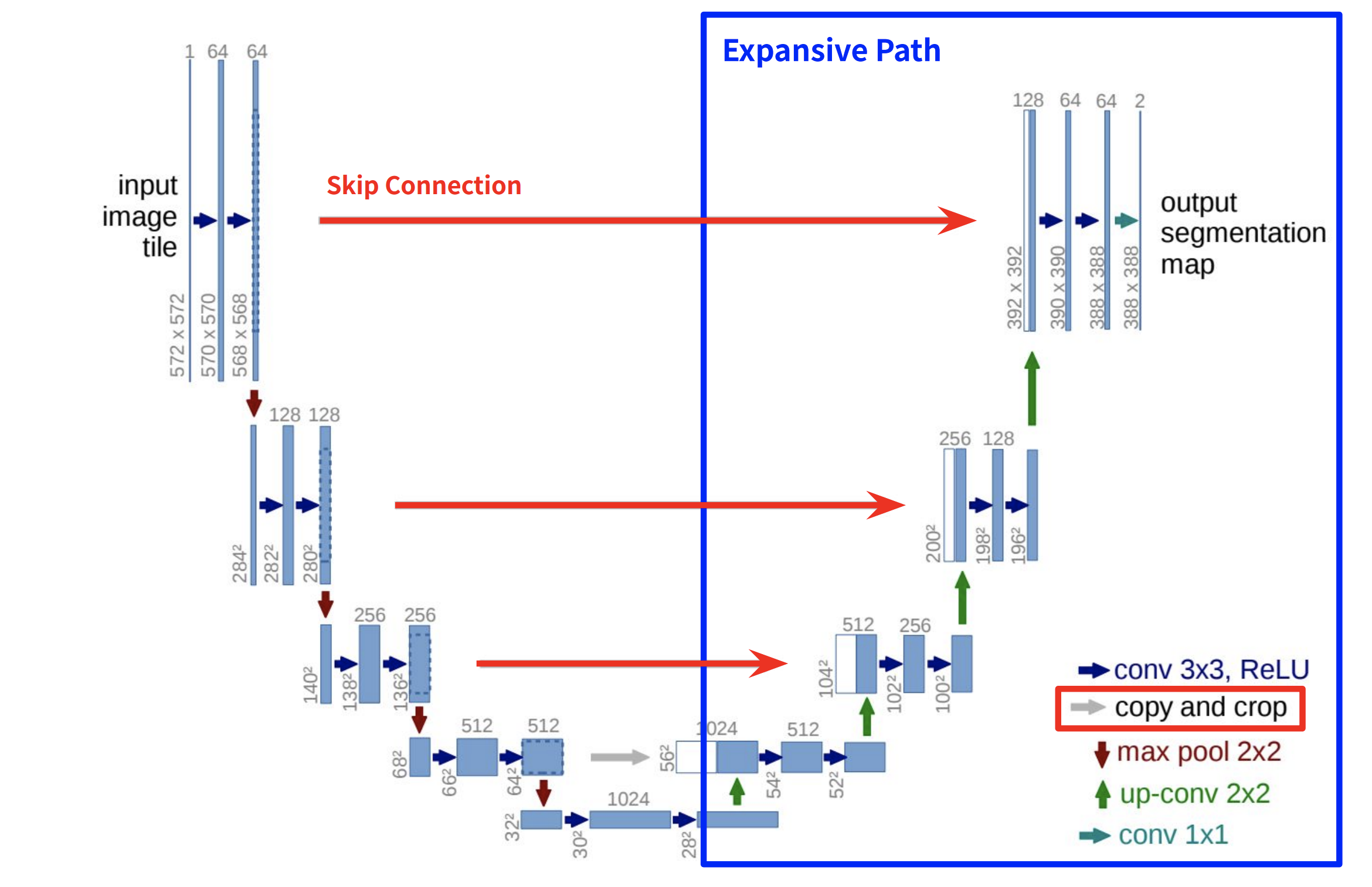
- Expansive Path
- 각 step마다 2x2 Up-conv(stride: 2)을 통해 feature map의 해상도가 2배로 늘어남
- 3x3 conv를 두 차례씩 반복 (패딩 x)
- conv layer에는 ReLU 연산이 포함
- Up-conv된 feature map은 Contracting path에서 cropped된 feature map과 concatenate 시킴 (Skip Connection)
- 마지막 레이어로 1x1 conv를 통해 Segmentation map 출력 (필터 개수 = 총 클래스 수)
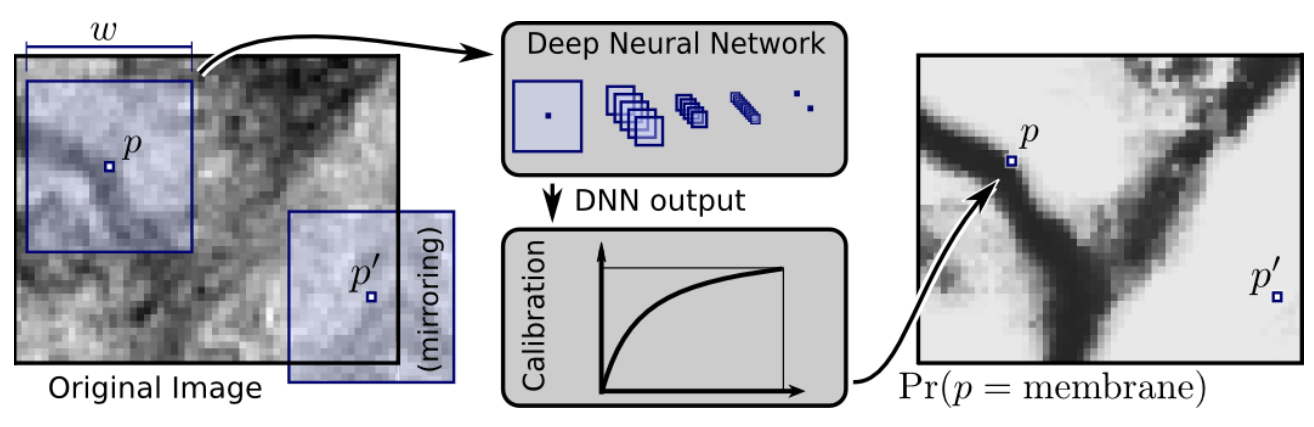
Overlap-Tile
- 크기가 큰 이미지의 경우엔 GPU Memory가 부족한 문제가 있으므로, 이미지 전체를 사용하는 대신 이미지를 각각의 Tile로 나눠 segmentation
- 하단의 사진과 같이 이미지를 타일로 나눠서 입력으로 사용
- segmentation을 위해 각 tile의 주변 픽셀을 같이 입력받기 때문에, 다음 tile에 대한 segmentation을 얻기 위해서는 이전 입력의 일부분이 포함됨 (overlap)
- 이미지의 경계 부분 픽셀에 대한 segmentation을 위해 미러링을 이용한 Extrapolation 기법 사용
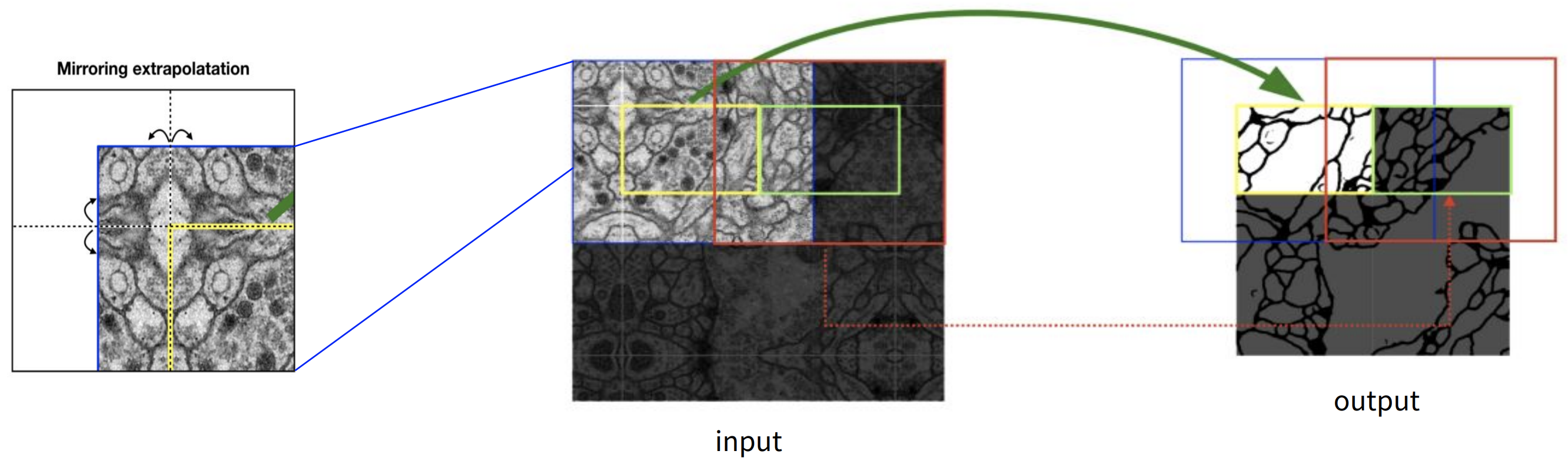
Mirroring Extrapolate
- 이미지의 경계 부분 픽셀에 대한 segmentation을 위해 0이나 임의의 패딩값을 사용하는 대신 이미지 경계 부분의 미러링을 이용한 Extrapolation 기법 사용
- 주요 데이터셋이 세포이므로, 비슷하게 퍼지는 세포의 특성에 따라 높은 성능을 낼 수 있음
3. Training
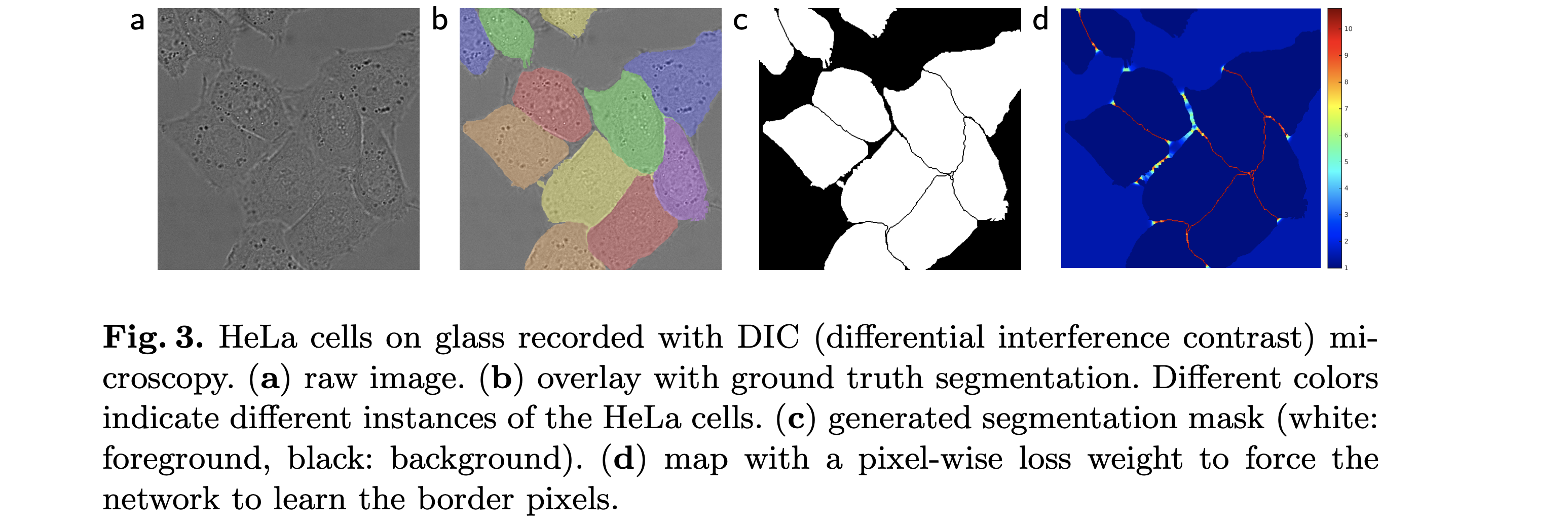
1) SoftMax를 사용하여 예측값을 확률로 변환
픽셀단위 예측값에 SoftMax 함수 P를 적용하여, 각 클래스에 속할 확률을 계산
2) 로그 확률 계산 (Cross Entropy Loss 적용)
정확한 예측일수록 손실이 작고 잘못된 예측일수록 손실이 커짐
3) 픽셀별 Weight 적용하여 Loss 계산
경계선 픽셀을 더 강하게 학습하기 위해 Cross Entropy Loss에
픽셀 고유의 Weight w(x)를 곱함
4) Weight Map 설계
가우시안 분포를 기반으로 경계선 픽셀에 더 높은 가중치를 부여하여 학습을 집중
→ 경계를 더 정교하게 예측할 수 있도록 유도
3.1 Data Augmentation
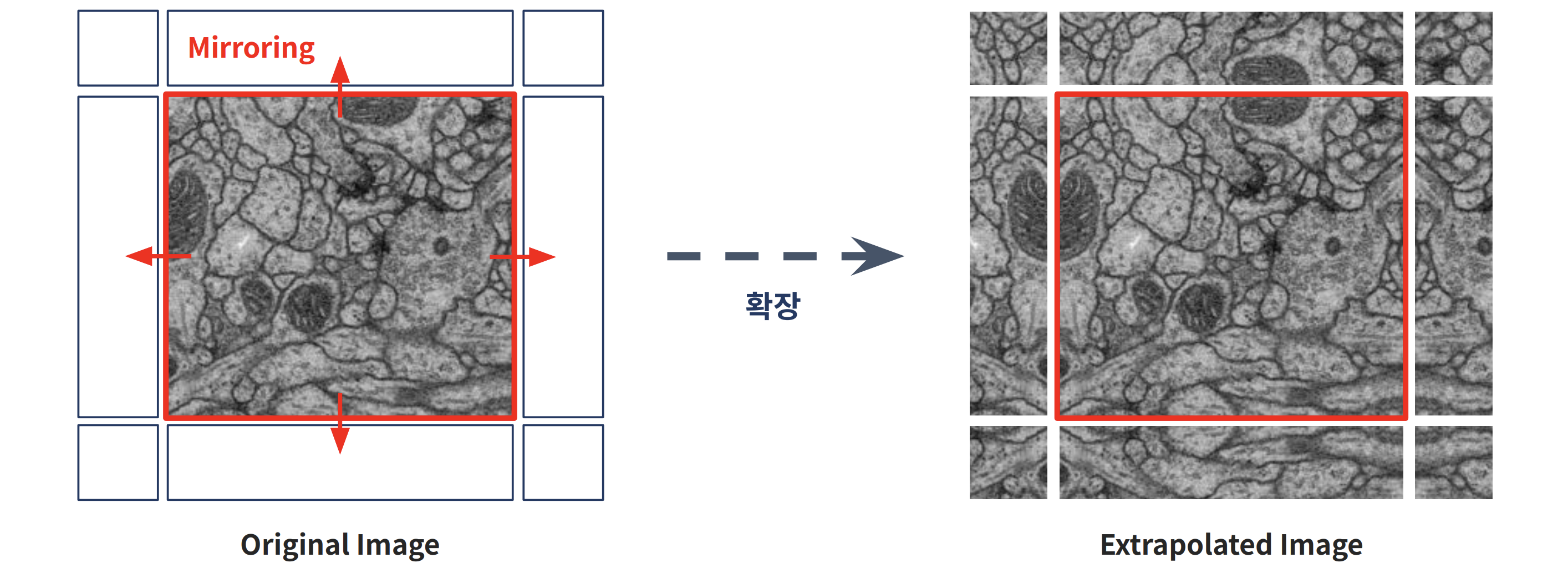
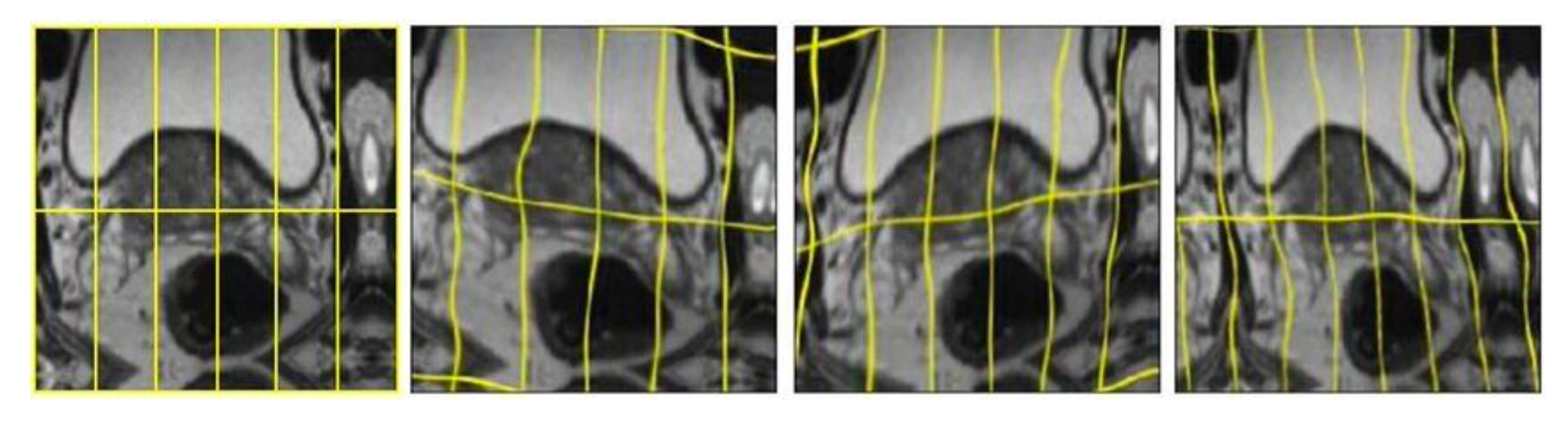
- Elastic Deformation
- 각 이미지 그리드에 확률적 노이즈를 더해 픽셀별로 서로 다른 방향으로 뒤틀림을 주기 때문에, 획일적이지 않고 자연스러운 형태 변형이 가능
- 실제 세포는 관측 시점마다 모양이 달라지므로, 이런 탄성 변형이 순간적인 형태 변화를 효과적으로 모사 가능
- 예시로, 아래의 전립선 MRI에 랜덤 탄성 변형을 적용하면 다양한 형태의 학습 데이터를 얻어, 데이터가 적어도 모델 성능을 크게 끌어올릴 수 있음
4. Experiments
EM Segmentation Challenge
전자 현미경 이미지에서 신경 구조 또는 세포 구조를 분할하는 문제를 다루는 대회
U-Net이 1위(0.000353 Warping Error) 기록
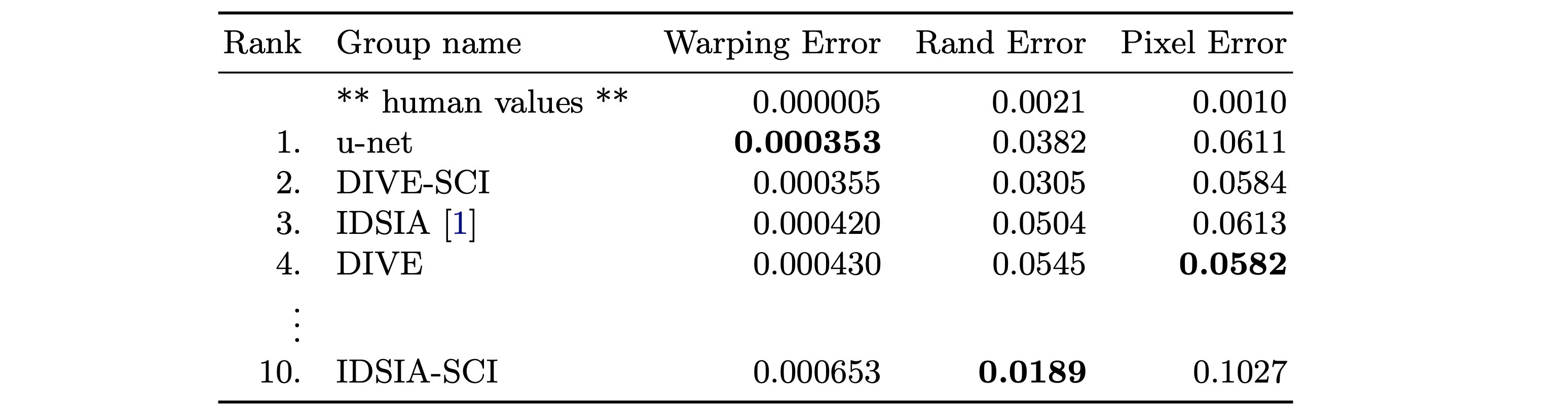
Warping Error : 신경 조직의 구조적 왜곡 정도를 측정하는 지표
Rand Error : 클러스터링 기반으로 객체 간 차이를 측정하는 지표
Pixel Error : 픽셀 단위에서 예측과 정답의 차이를 측정하는 지표ISBI Cell Tracking Chanllenge
세포의 성장, 분열 및 이동을 자동으로 추적하는 문제를 해결하는 대회
ISBI 2015 Cell Tracking Challenge에서 1위
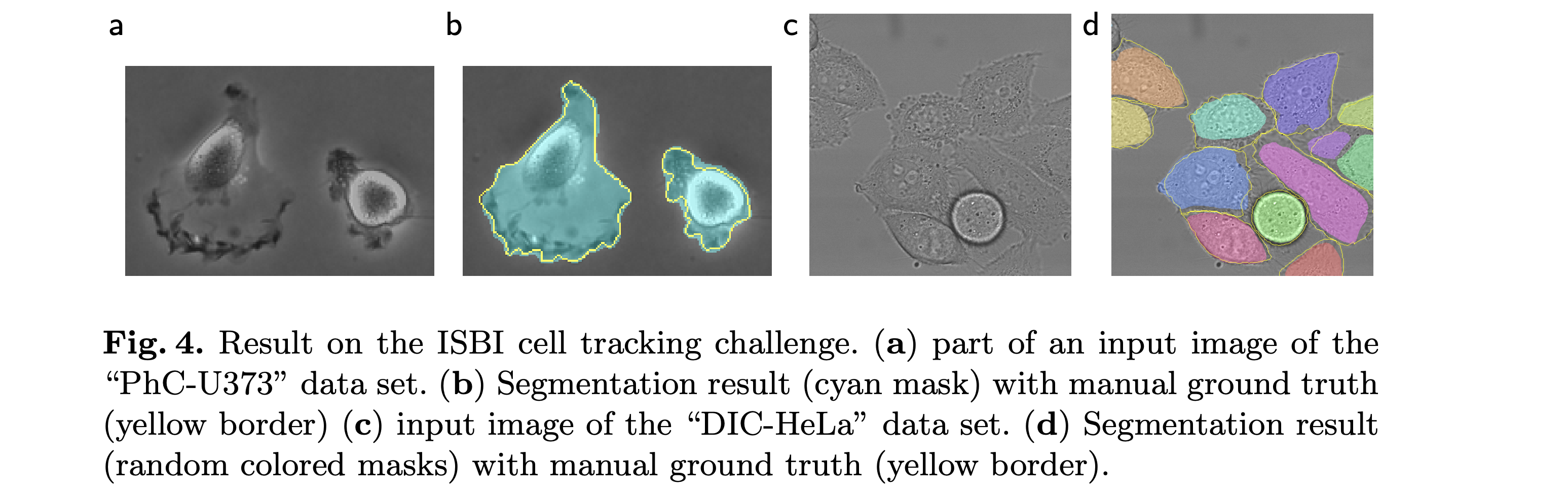
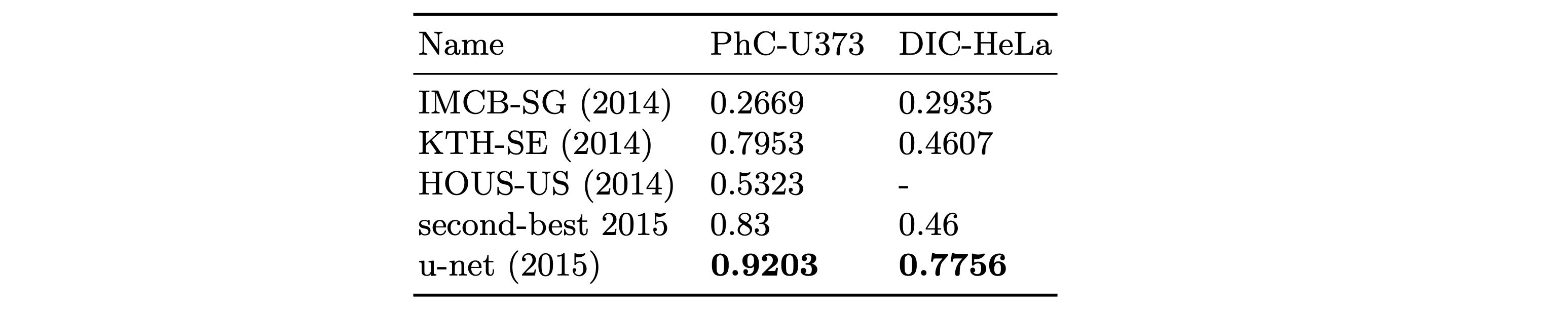
PhC-U373 : 위상차 현미경 이미지
DIC-HeLa : DIC 방식으로 촬영된 HeLa 세포 이미지
모두 IoU(Intersection over Union)로 평가5. Conclusion
U-Net은 다양한 생물의학 이미지 분할 문제에서 매우 우수한 성능을 보임
Elastic Deformation을 활용한 데이터 증강 덕분에 적은 데이터로도 학습 가능
NVidia Titan GPU (6GB)에서 10시간 학습→빠르고 효율적인 모델
∴ U-Net은 의료 영상 분석 및 다양한 이미지 분할 문제에서 표준 모델로 자리 잡음
