EF(Enrichment Factor)
- 정의 : EF는 실제로 발견된 활성 활성 화합물의 비율을 랜덤 선택으로 기대되는 활성 화합물의 비율과 비교한 값
- 공식 :
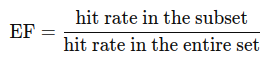 - hit rate는 활성 화합물의 비율
- hit rate는 활성 화합물의 비율
- ex ] 전체 데이터셋에 100개의 화합물이 있고, 그 중 10개가 활성 화합물(hits)이라면, 전체 데이터셋의 hit rate는 10%. 만약 특정 조건에서 20개의 화합물을 선택했을 때 그 중 5개가 활성 화합물이라면, 이 조건에서의 hit rate는 25%. 이 경우 EF는 2.5(25% / 10%) - 결론 : 스크리닝 실험에서 특정 특성의 농축 정도를 나타내며, 기대값에 비해 실제로 발견된 비율을 측정
데이터 설명 및 사용 목적
1. Metadata:
- kmap의 sample id: KMAP 데이터의 샘플 ID.
- cmap의 sample id: CMAP 데이터의 샘플 ID.
- KID: KMAP에서 사용된 샘플의 고유 식별자.
- C dose: CMAP 실험에서 사용된 화합물의 농도.
- K dose: KMAP 실험에서 사용된 화합물의 농도.
- Rdata 파일:
- CMAP - MCF7와 CMAP - 전체: CMAP 프로젝트에서 나온 유전자 발현 데이터.
- KMAP: KMAP 프로젝트에서 나온 유전자 발현 데이터.
전반적인 분석 흐름
- 데이터 불러오기:
R을 사용하여 각 Rdata 파일을 불러옴
Metadata 파일도 불러와서 KMAP와 CMAP의 샘플 ID를 매칭시키기
- 유전자 발현 비교:
같은 샘플에 대해 KMAP와 CMAP에서 각각의 유전자 발현 데이터를 추출
이를 통해 동일 샘플에서 유전자 발현의 up-regulation과 down-regulation을 비교
- Enrichment Factor(EF) 계산:
EF는 특정 유전자의 발현 변화가 얼마나 기대되는지와 실제 관찰된 변화 간의 비율을 나타냄
이는 통계적으로 유의미한 유전자 발현 변화를 식별하는 데 사용
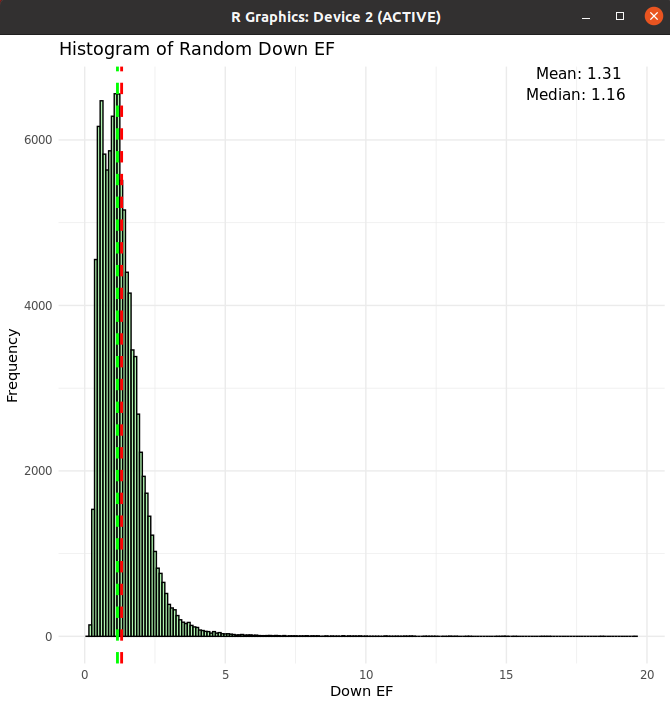
Random cmap으로 EF
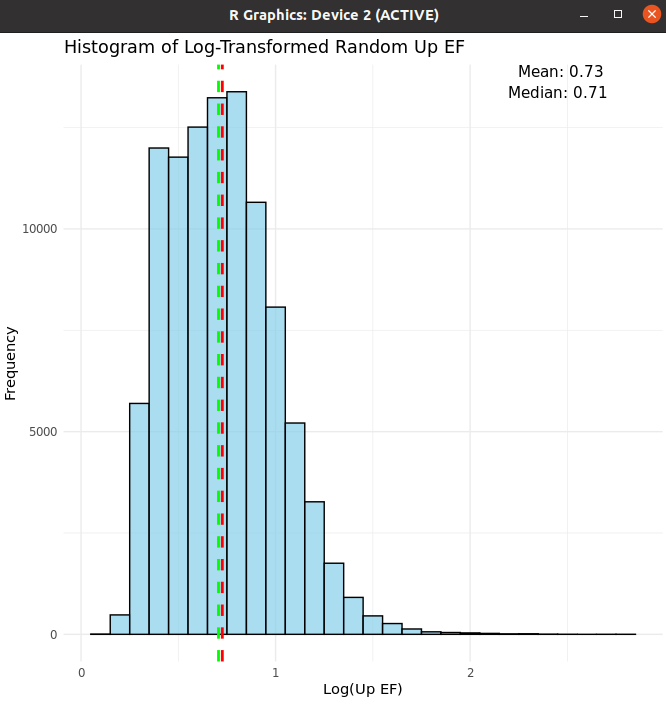
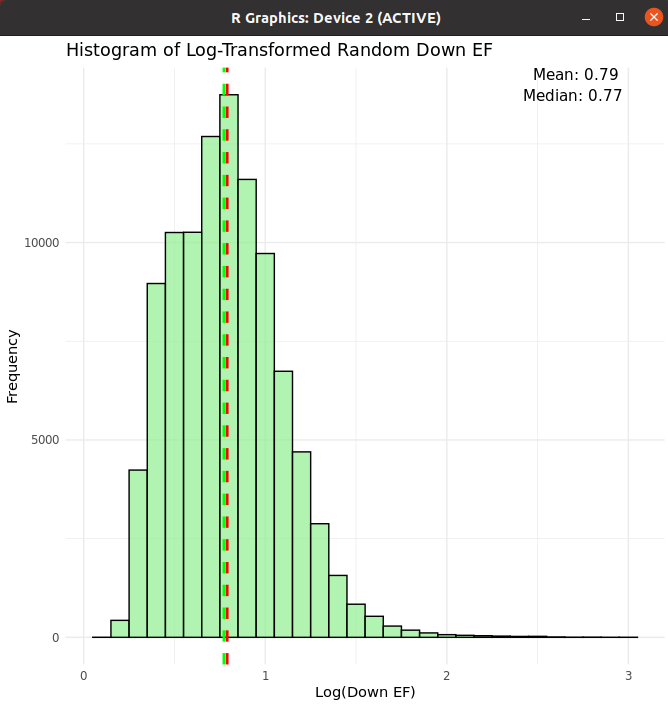
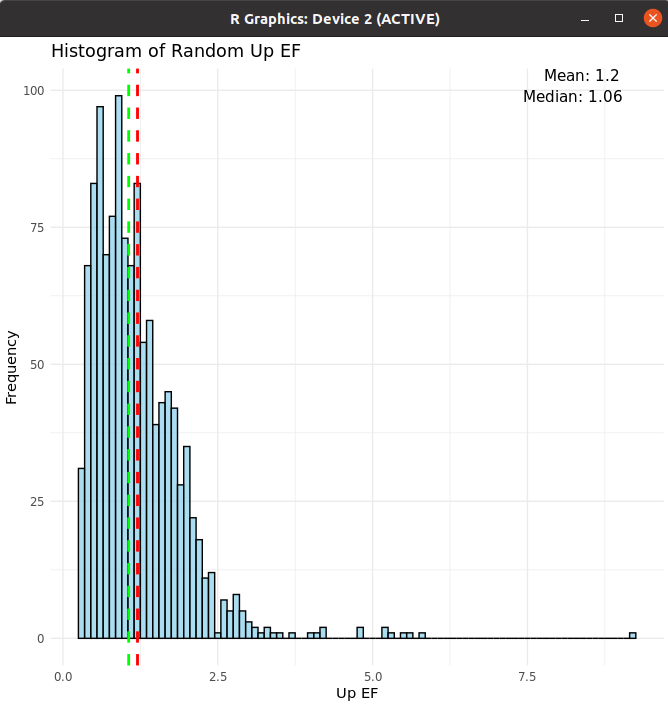
Histogram
- 전체적인 분포 파악을 위함
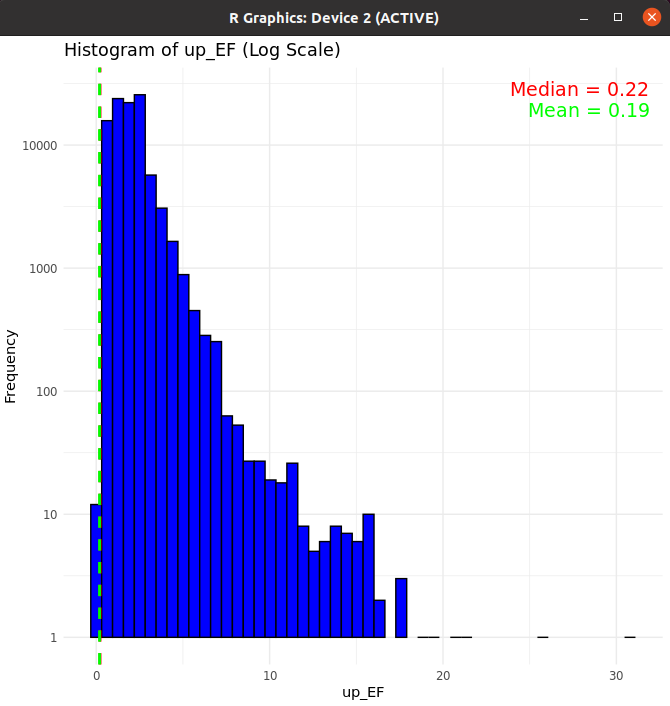
up_EF
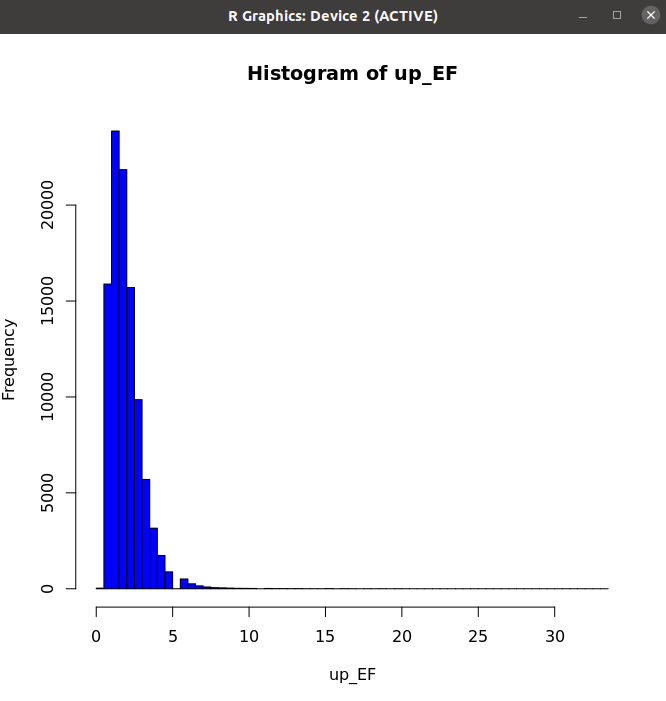
Original Data - Median: 1.66
Original Data - Mean: 1.85
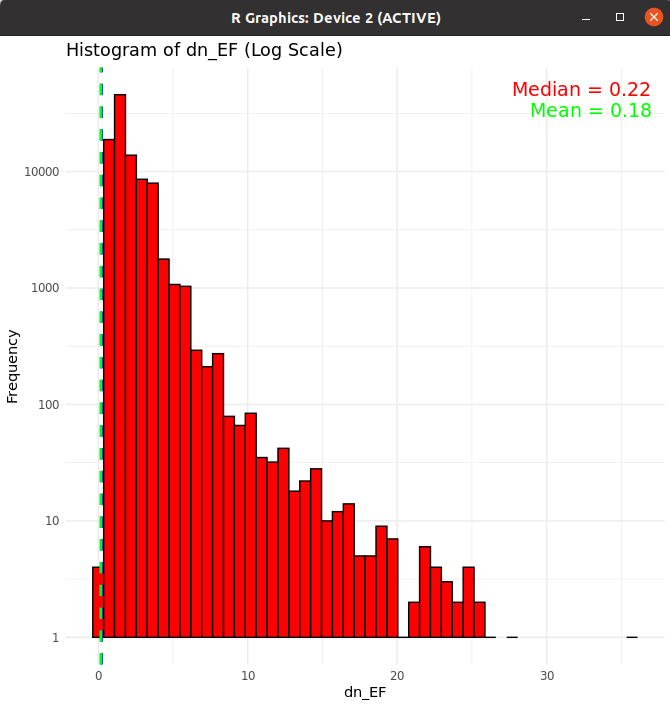
down_EF
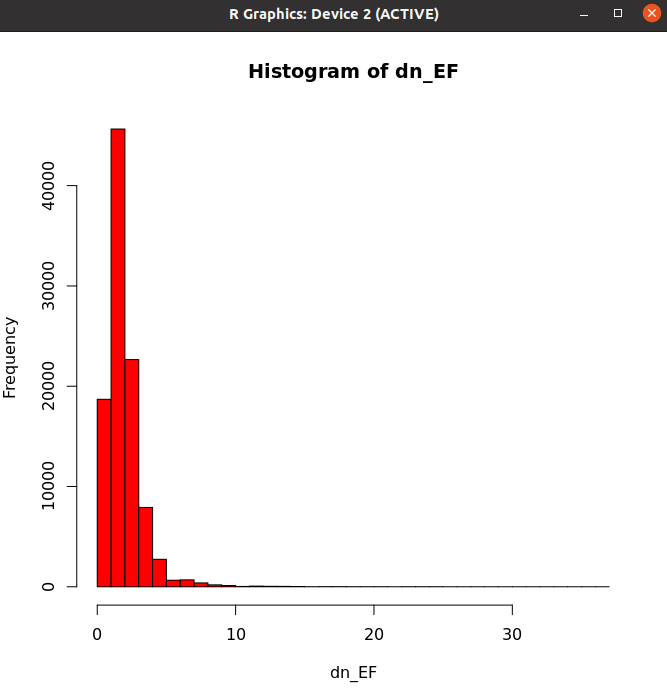
Original Data - Median: 1.66
Original Data - Mean: 1.85
- 로그 적용

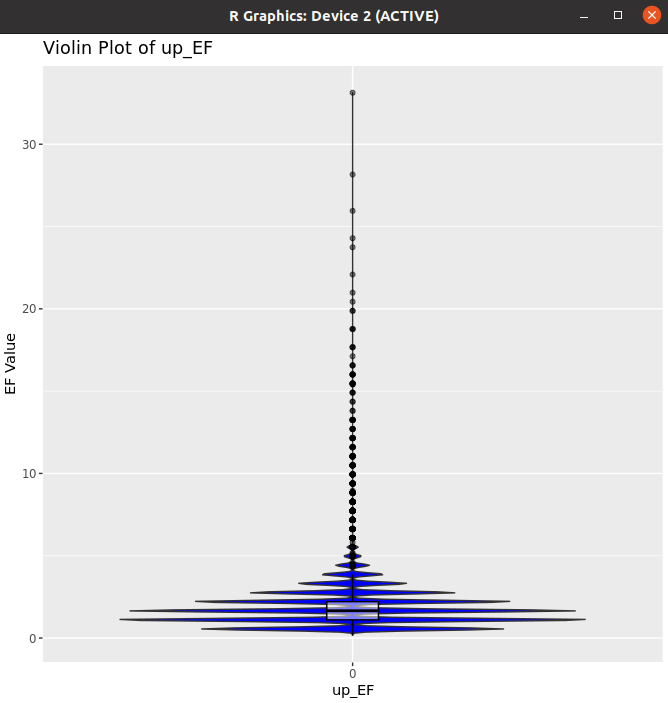
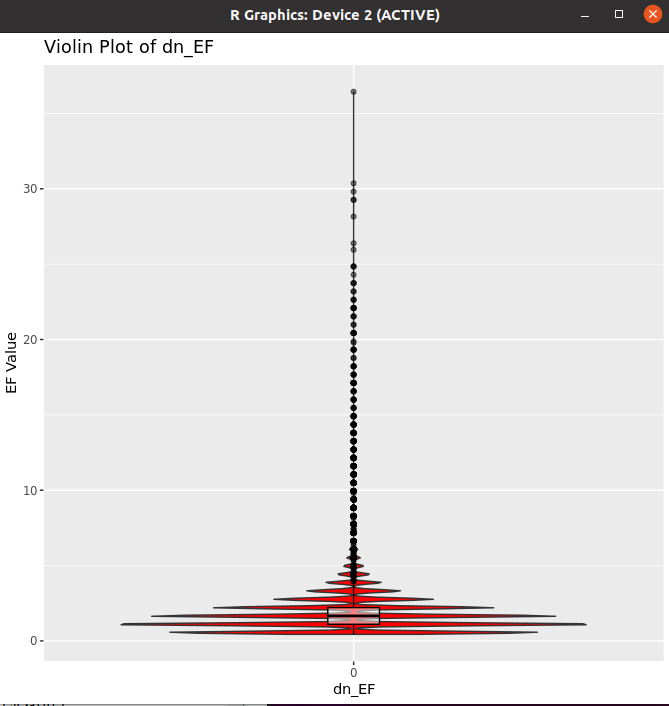
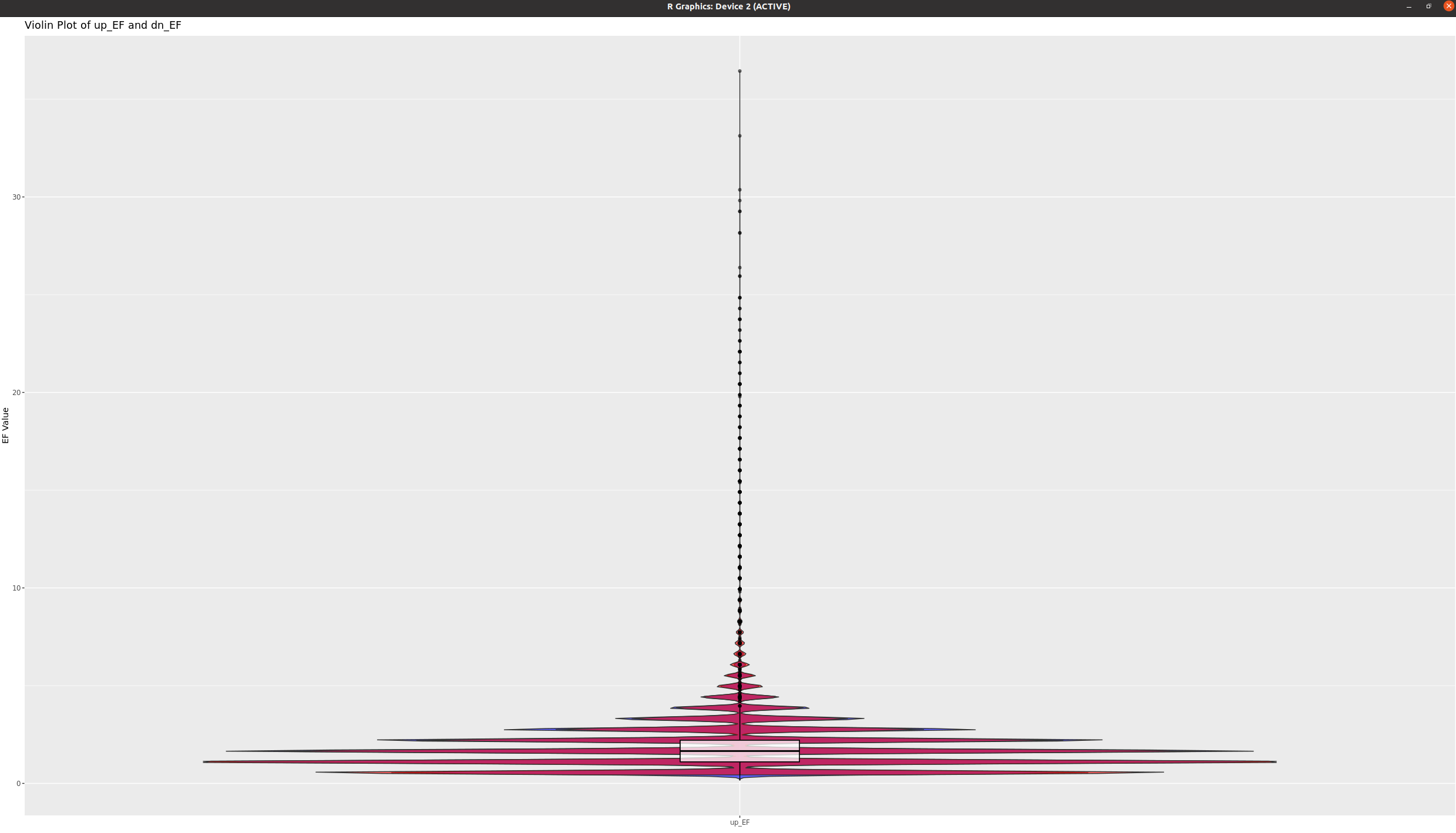
Violin Plot
- 데이터의 밀도와 분포 시각화
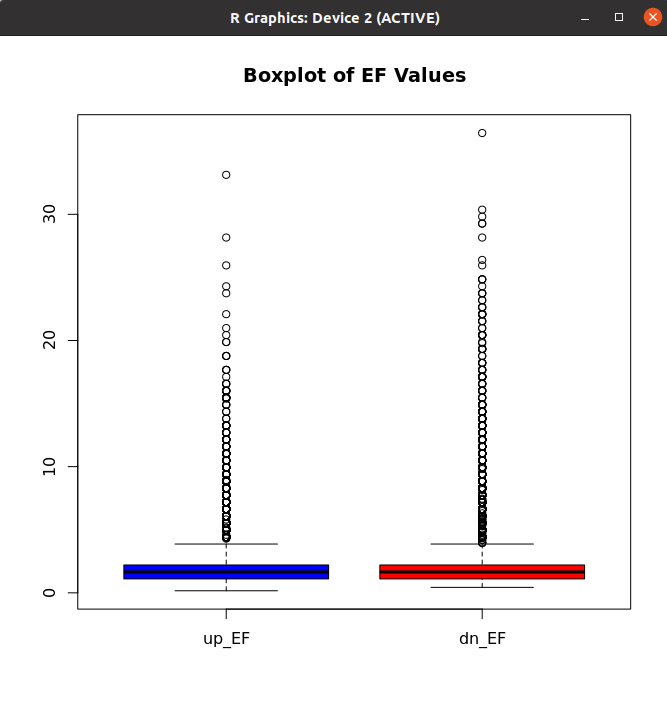
Box Plot
- 두 데이터 간의 비교
core와 반복 수를 늘려봤는데 분포가 달라지지 않았음
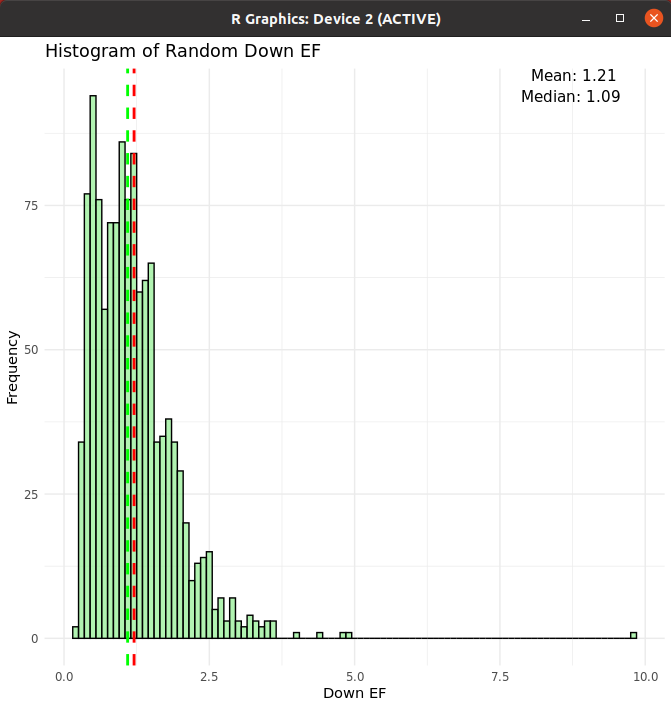
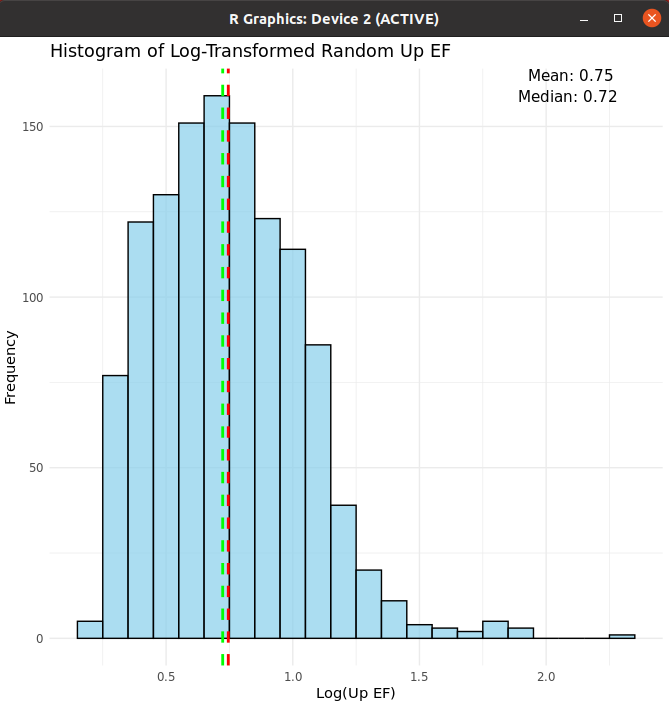
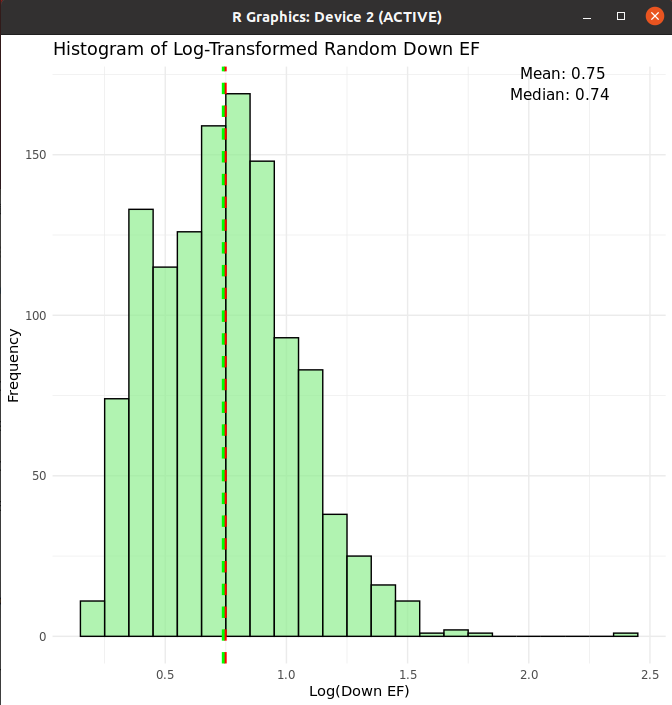
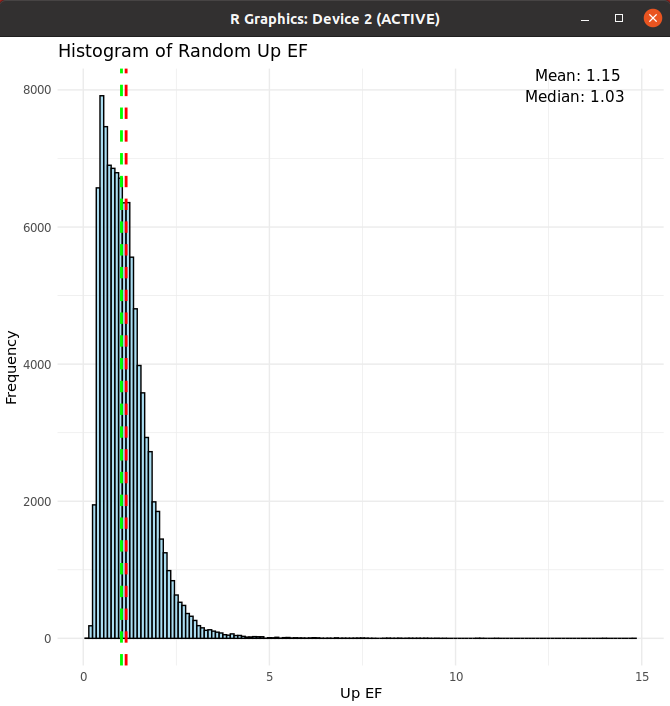
Random sample로 EF
총 데이터 길이만큼 random
- Log 취한 경우
100000번 random 돌린 경우
- log 취한 경우