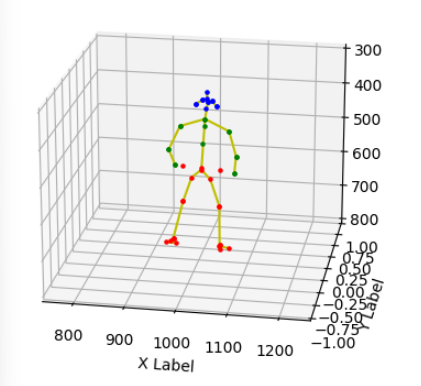
frankmocap output으로부터 3d skeleton 결과를 추출해서 visualize하는 것이 목표이다
0. 탐색
먼저 frankmocap git 페이지를 다시 읽어보자
frankmocap/docs/run_bodymocap.md를 보면 생각보다 다양한 옵션이 있다.
- screen / screenless mode
- renderer_type (default는 opengl, 옵션으로 pytorch3D, openDR)
- webcam input
- openGL GUI mode key
- precomuted Bboxes
내가 봐야할 옵션은 여기다
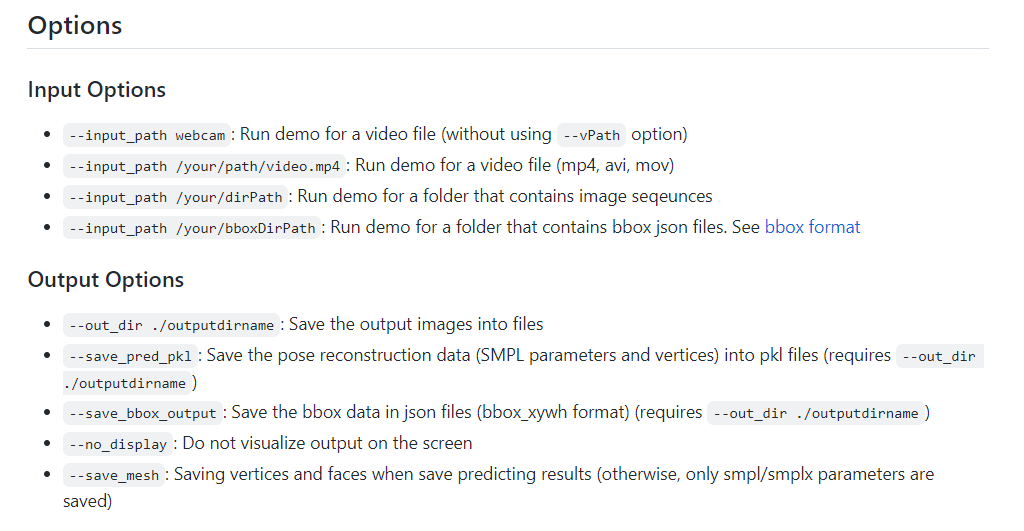
pose reconstruction data(SMPL 파라미터와 정점)은 pkl에 저장되어있다고 써있다
즉 --save_pred_pkl 옵션을 사용하면 될 것 같다
pkl에 대한 정보를 더 찾아보자

맨 마지막 pred_joints_img에 3d skeleton에 대한 정보가 담겨있다고 한다
1. PKL 찾아보기
save pkl 옵션을 킨 채로 코드를 다시 실행해보자
output 디렉토리 경로에 들어가보자
input 영상이 매 frame마다 jpg로 output이 나왔던 것처럼 pkl 역시 매 frame마다 저장되어있다
pkl을 조금 더 자세히 보자
먼저 첫번째 pkl 파일을 읽자
orderedDict 형식으로 순서대로 7개 항목이 있는 것을 볼 수 있다. 우리가 찾는건 pred_output_list에 있을 것 같다

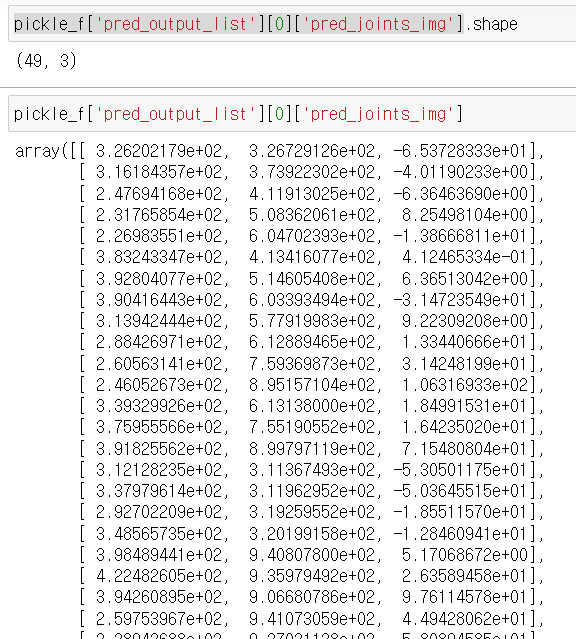
pred_joints_img가 있는 것을 확인할 수 있다

(49,3) 형태로 즉 joint point가 49개, 각각의 joint point는 x,y,z 3차원으로 구성되어있는 것을 알 수 있다.
2. Joint Connection
각 joint point를 찾았다면 연결할 순서를 찾아봐야한다. 먼저 joint point 순서에 대한 정보를 찾아보자.
frankmocap/docs/joint_order.md에 나와있다
'OP Nose', # 0
'OP Neck', # 1
'OP RShoulder', # 2
'OP RElbow', # 3
'OP RWrist', # 4
'OP LShoulder', # 5
'OP LElbow', # 6
'OP LWrist', # 7
'OP MidHip', # 8
'OP RHip', # 9
'OP RKnee', # 10
'OP RAnkle', # 11
'OP LHip', # 12
'OP LKnee', # 13
'OP LAnkle', # 14
'OP REye', # 15
'OP LEye', # 16
'OP REar', # 17
'OP LEar', # 18
'OP LBigToe', # 19
'OP LSmallToe', # 20
'OP LHeel', # 21
'OP RBigToe', # 22
'OP RSmallToe', # 23
'OP RHeel', # 24
'rankle', # 25
'rknee', # 26
'rhip', # 27
'lhip', # 28
'lknee', # 29
'lankle', # 30
'rwrist', # 31
'relbow', # 32
'rshoulder', # 33
'lshoulder', # 34
'lelbow', # 35
'lwrist', # 36
'neck', # 37
'headtop', # 38
'hip', # 39 'Pelvis (MPII)', # 39
'thorax', # 40 'Thorax (MPII)', # 40
'Spine (H36M)', # 41
'Jaw (H36M)', # 42
'Head (H36M)', # 43
'nose', # 44
'leye', # 45 'Left Eye', # 45
'reye', # 46 'Right Eye', # 46
'lear', # 47 'Left Ear', # 47
'rear', # 48 'Right Ear', # 48해당 깃 페이지에서 joint connection 정보는 찾지 못해서
https://github.com/mkocabas/VIBE/blob/master/lib/data_utils/kp_utils.py#L212
여기에서 참고했다
[
[0 , 1],
[1 , 2],
[2 , 3],
[3 , 4],
[1 , 5],
[5 , 6],
[6 , 7],
[1 , 8],
[8 , 9],
[9 ,10],
[10,11],
[8 ,12],
[12,13],
[13,14],
[0 ,15],
[0 ,16],
[15,17],
[16,18],
[21,19],
[19,20],
[14,21],
[11,24],
[24,22],
[22,23],
[0 ,38],
]대충 첫 줄부터 보자면
0번 1번 joint point를 연결 = Nose와 neck을 연결
1번 2번 joint point를 연결 = Neck와 RShoulder를 연결
2번 3번 joint point를 연결 = RShoulder와 RElbow를 연결
3. Visualization
3D 시각화는 아래를 참고했다
https://matplotlib.org/stable/gallery/mplot3d/2dcollections3d.html#sphx-glr-gallery-mplot3d-2dcollections3d-py
한 frame에 대한 시각화부터 해보자
첫번째 pkl 파일을 읽는다
관련 library를 import하고 3d plot figure를 생성한다
import numpy as np
import matplotlib.pyplot as plt
from mpl_toolkits.mplot3d import Axes3D
pickle_f = pd.read_pickle('00000_prediction_result.pkl')
fig = plt.figure()
ax=fig.add_subplot(projection='3d')시각화의 편의를 위해 머리, 상체, 하체의 point 색을 다르게 주었다(clist)
- 머리 : Blue
- 상체 : Green
- 하체 : Red
아까 읽은 pred_joints_img를 반복문으로 돌면서 scatter로 점을 찍는다
clist = ['b','g','g','g','g','g','g','g','r','r','r','r','r','r','r','b','b','b','b','r','r','r','r','r','r','r','r','r','r','r','r','g','g','g','g','g','g','g','b','r','g','g','b','b','b','b','b','b','b']
for i,data in enumerate(pickle_f['pred_output_list'][0]['pred_joints_img']):
ax.scatter(xs=data[0],ys=data[1],zs=0,zdir='y',s=5, c=clist[i])joint points를 찍었으면 joint connection으로 점을 이어준다
이때 R_array는 위에서 찾은 joint connection index를 array로 만든 것
R_array를 반복문으로 돌면서 2개의 joint point의 인덱스를 읽고 그걸로 pred_joints_img에서 점의 좌표를 찾아 plot한다
R_array = np.array([[0 , 1],[1 , 2],[2 , 3],[3 , 4],[1 , 5],[5 , 6],[6 , 7],[1 , 8],[8 , 9],[9 ,10],[10,11],[8 ,12],[12,13],[13,14],[0 ,15],[0 ,16],[15,17],[16,18],[21,19],[19,20],[14,21],[11,24],[24,22],[22,23],[0 ,38],])
print(R_array.shape)
for r_list in R_array:
a = r_list[0]
b = r_list[1]
aplk = pickle_f['pred_output_list'][0]['pred_joints_img'][a]
bplk = pickle_f['pred_output_list'][0]['pred_joints_img'][b]
ax.plot([aplk[0],bplk[0]],[aplk[1],bplk[1]],zs=0,zdir='y',c = 'y')축과 plot 시점을 설정해주고 plotting한다
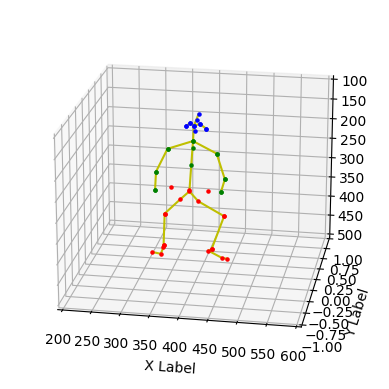
시각화 전체 코드는 git에 올려두었다
https://github.com/dangdang2222/frankmocap_project/blob/main/3D_skeleton_joint_conection_visualize(2D_to_3D).ipynb
