Background
윌름스 종양(WT)의 방사선적 체적 평가는 치료 결정을 안내하는 중요한 지표이다. 그러나 종양의 이질성으로 인해 방사선과 의사는 진단에 있어 주요 보호자 차이가 있으며, 이는 잘못된 진단과 열악한 치료로 이어질 수 있다. 이 연구의 목적은 딥러닝을 사용하여 CT 기반 WT 병소 윤곽을 자동화할 수 있는지 알아보는 것이다.
Introduction
이 연구의 목적은 WT 병변의 분할을 위한 딥러닝의 적용을 탐구하고 영상 분석을 위한 자동 분할의 타당성을 평가하는 것이다.
Data
WT 환자 106명의 데이터에 대해 수행되었다. 또한, 아래의 조건에 맞는 데이터를 사용했다.
- 병리학적으로 검증된 WT
- 치료 전 복부 강화 CT
- 문맥 단계를 포함한 CT 강화 영상
저해상도 영상이나 모션 아티팩트가 있는 환자를 제외한 후 총 105명의 환자의 데이터를 사용하였다.
Image processing
3D Slicer 플랫폼을 사용하여 ROI를 추출했다.
종양 크기는 종양 분할의 복셀 수에 복셀 크기()를 곱하여 계산했다.
Deep learning
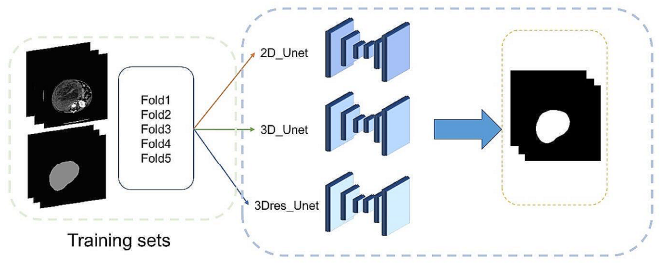
U-net 기반 적응형 분할 프레임워크인 nnU-Net을 자동 종양 분할에 사용했다. 2D Unet, 3D Unet, 3Dres Unet(U-Net Cascade)의 세 가지 모델이 훈련 세트 데이터를 사용하여 훈련되었다. 환자 데이터를 완전히 활용하기 위해 5-fold cross-validation이 훈련 과정에 적용되었다. training 후, 세 모델의 출력이 쌍으로 결합되었고 가장 좋은 모델이 테스트 이미지 세트에서 검증되었다.
그런 다음 결과를 수동 분할 결과와 비교하고 분석했다.
training 과정에서 모든 신경망을 1000번 반복하여 훈련했고, 훈련하는 동안 2D 및 3D 네트워크는 각각 12와 2의 batch size로 512×512 및 80×192×160으로 샘플링되었다.
- overview of the deep learning workflow
Distribution of tumors
전체 데이터 세트와 훈련 세트의 종양 크기 분포는 서로 비교적 가깝고, 중앙값과 평균값은 크게 다르지 않고 일관된 범위를 유지한다. 전체 데이터 세트와 훈련 세트 모두 중간값과 평균값이 테스트 세트에 비해 약간 더 높습니다.
Deep learning-based segmentation
세 개의 네트워크를 기반으로, 훈련 세트에서 5-fold cross-validation을 수행했고
각 네트워크에 대해 가장 높은 DSC를 가진 모델을 선택했다.
이러한 모델을 결합한 결과 6개의 분할 모델이 나왔는데, 6개의 모델 중에서 결합 모델인 3D-3Dres가 최고의 성능을 나타냈다. 따라서 테스트에 3D-3Dres 모델이 사용되었다.
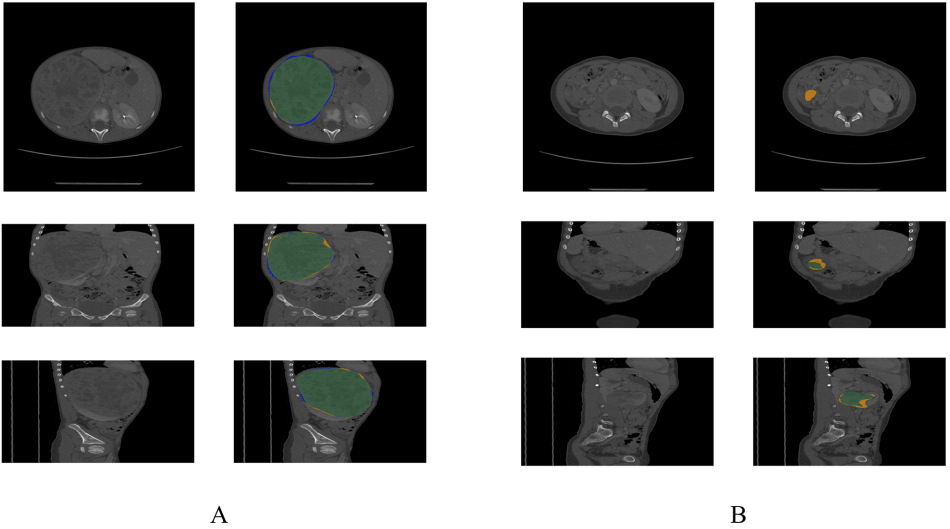
A: CT image of a patient with DSC of 0.9715
B: CT image of a patient with DSC of 0.2927
green area: manual segmentation & automatic segmentation 겹치는 부분
blue area: manual segmentation
yellow area: automatic segmentation
Tumor size
3D 직경 비율에 기반한 predicted volume은 자동 분할 후 volume을 직접 계산한 것보다 수동 분할과 더 일치했다.
Discussion
3차원의 최대 직경을 수동으로 측정한 것과 결합하면, 딥러닝은 종양 크기를 더 정확하게 예측할 수 있다.
하지만 주의해야 할 것은 테스트 세트의 환자가 나이가 더 많았으며, 종양 크기의 경우 테스트 세트의 종양 병변 크기가 약간 더 작았다는 것이다. 따라서 테스트 세트의 더 작은 병변 크기는 모델의 검증에 영향을 미쳤다.
수동으로 분할된 병변 크기의 분포를 보면 크기가 비교적 분리되어 있는 것처럼 보이지만 자동으로 분할된 병변 크기는 중앙 집중화된 분포를 보인다.
비교적 작은 종양을 분할하는 데 상당히 더 나은 성능을 보였으며, 평균 크기의 종양을 분할하는 데 성능은 가장 안 좋았다.