오늘은 graphsage라는 프레임워크를 제안한 논문에 대해 다루어 보겠습니다. Node2vec이나 DeepWalk와 같은 shallow node embedding learning을 통해 고정된 node들에 대한 representation을 학습할 수 있습니다.
하지만, 실제 graph로 표현되는 데이터는 새로운 node들이 실시간으로 추가되는 경우(Evolving Graph)가 매우 많습니다. 예를 들어 인스타그램 사용자를 node, 이들 간의 연결 관계를 edge라고 해 보겠습니다. 어떤 시점에서 명의 인스타그램 사용자들에 대한 node representation을 학습했다고 하죠. 다음 날 10000명의 신규 가입자가 추가되었다면, 명에 대한 node representation을 처음부터 다시 학습해야 합니다.
본 논문에서는 fixed graph에 대한 node embedding을 학습하는 transductive learning 방식의 한계점을 지적하고, evolving graph에서 새롭게 추가된 node에 대해서도 inductive node embedding을 산출할 수 있는 프레임워크인 GraphSage를 제안합니다.
1. Embedding Generation
그래프의 노드 각각에 대한 embedding을 직접 학습하게 되면, 새로운 노드가 추가되었을 때 그 새로운 노드에 대한 embedding을 추론할 수 없습니다. 따라서 GraphSage는, 노드 임베딩이 아닌 aggregation function을 학습하는 방법을 제안합니다.
1.1. Algorithm
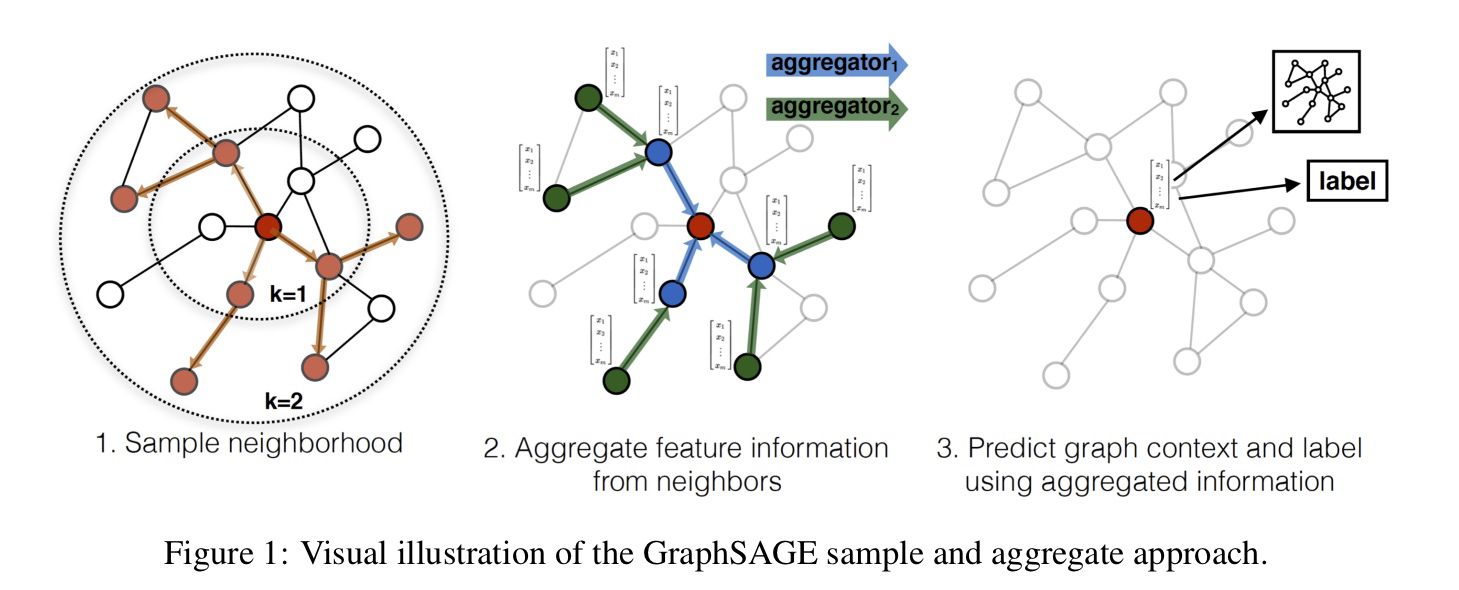
모든 노드들은 feature를 가집니다. node feature는 node embedding과는 다른 개념입니다. 만약 소셜 네트워크 그래프에서 각각의 노드가 개별 사용자를 의미한다면, node feature는 사용자의 성별, 국적, 나이, 혹은 node degree, node centrality 등이 될 수 있겠죠.
위 그림에서 가운데에 있는 빨간색 노드가 새롭게 추가된 노드라고 해 보겠습니다. 우리는 이 빨간색 노드의 embedding을 구해야 합니다.
-
우선 거리(k)를 기준으로 일정 개수의 neighborhood node를 샘플링합니다.
-
그리고 graphsage를 통해 학습된 aggregation function을 통해, 주변 노드의 feature로부터 빨간 노드의 임베딩을 계산합니다.
-
이렇게 추론된 새로운 노드에 대한 임베딩을 downstream task에 활용합니다.
위 과정을 pseudo code로 아래와 같이 나타낼 수 있습니다. 특정 노드의 임베딩을 계산할 때, 거리가 K 만큼 떨어져 있는 노드에서부터 순차적으로 feature aggregation을 적용하는 것이죠. 하지만, 이것을 실제로 구현하기 위해서는 batch를 샘플링하는 방법과 node neighborhood에 대한 정의가 필요합니다.
특정 노드의 임베딩을 계산할 때, 거리가 K 만큼 떨어져 있는 노드에서부터 순차적으로 feature aggregation을 적용하는 것이죠. 하지만, 이것을 실제로 구현하기 위해서는 batch를 샘플링하는 방법과 node neighborhood에 대한 정의가 필요합니다.
1.2. Batch Sampling
위 Algorithm 1 의 line 2를 보면, 그래프에 포함된 모든 노드에 대하여 임베딩을 구합니다. 하지만, graphsage를 학습하는 과정에서는 batch단위로 연산이 이루어져야 합니다. 따라서 본 논문에서는 아래와 같은 batch sampling 알고리즘(line1 ~ line7)을 사용합니다. 
1.3. How to Define Neighborhood?
다음으로 볼 것은 어떻게 특정 node 의 neighborhood 를 정의하는가에 대한 것입니다. 실제 학습 과정에서 를 모든 근접 노드로 적용한다면, 계산 복잡도를 제어할 수 없습니다. 따라서, 각 iteration 마다
uniform random draw 방식으로 정해진 개수의 최 근접 노드를 샘플링합니다.
2. Aggregator function & Learning Graphsage
2.1. Aggregator function
aggregator function은 이웃 노드들로부터의 정보를 aggregate하는 역할을 합니다. 하지만 그래프 데이터의 특성 상, 노드의 neighborhood들 간에는 어떤 순서가 없습니다. 따라서, aggregator function은 symmetric하고 높은 수용력(high representational capacity)을 지님을 동시에 학습 가능해야 합니다. 본 논문에서는 세 가지 variant를 제안합니다.
- Mean Aggregator
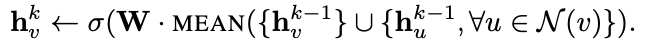 mean aggregator function은 주변 노드의 임베딩과 자기 자신(ego node)의 임베딩을 단순 평균한 후, 선형 변화와 relu를 적용해 줌으로써, 임베딩을 업데이트 합니다.
mean aggregator function은 주변 노드의 임베딩과 자기 자신(ego node)의 임베딩을 단순 평균한 후, 선형 변화와 relu를 적용해 줌으로써, 임베딩을 업데이트 합니다.
-
LSTM Aggregator
LSTM aggregator는 높은 수용력을 가진다는 장점을 갖고 있습니다. 하지만 LSTM 자체는 symmetric한 함수가 아니라는 문제가 있습니다. 따라서 본 연구에서는, 인풋 노드들의 순서를 랜덤하게 조합하는 방식을 취합니다. -
Pooling Aggregator
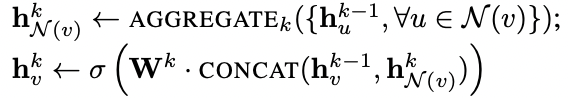 Algorithm 1을 보면, 위와 같은 표현이 있습니다. 여기서 함수는 아래와 같습니다.
Algorithm 1을 보면, 위와 같은 표현이 있습니다. 여기서 함수는 아래와 같습니다. 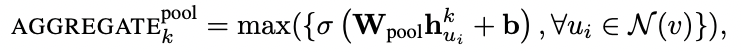 각 노드의 임베딩에 대해 선형 변환(linear transformation)을 수행한 뒤, element-wise max pooling을 통해 이웃 노드들의 정보를 aggregate하는 방식입니다.
각 노드의 임베딩에 대해 선형 변환(linear transformation)을 수행한 뒤, element-wise max pooling을 통해 이웃 노드들의 정보를 aggregate하는 방식입니다.
2.2. Learning Graphsage
본 연구에서 제안하는 것은, 각 노드들의 feature를 aggregate함으로써 각 노드의 임베딩을 추론할 수 있는 aggregator function의 파라미터를 학습하는 것입니다. 하지만 optimization objective는 기존의 node2vec과 같은 shallow embedding network와 크게 다르지 않습니다.
iteration 후의 node representation인 에 대해 손실함수가 계산되며, aggregator function의 파라미터인 가 gradient descent를 통해 학습됩니다.
손실함수는 아래와 같습니다. 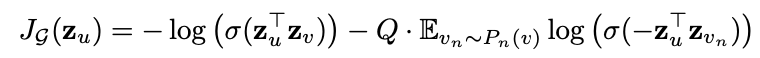 부가 설명을 하자면, 와 는 random walk를 기반으로 이웃으로 설정된 노드 쌍이고, 은 에 대한 negative node(이웃이 아닌 노드)입니다. 즉, 이웃 노드끼리는 유사도가 높은 임베딩을 갖도록, 이웃이 아닌 노드끼리는 유사도가 낮은 임베딩을 갖도록 학습이 이루어지게 됩니다.
부가 설명을 하자면, 와 는 random walk를 기반으로 이웃으로 설정된 노드 쌍이고, 은 에 대한 negative node(이웃이 아닌 노드)입니다. 즉, 이웃 노드끼리는 유사도가 높은 임베딩을 갖도록, 이웃이 아닌 노드끼리는 유사도가 낮은 임베딩을 갖도록 학습이 이루어지게 됩니다.
3. Experiments
3.1. Graph Datasets
Citation Dataset
- undirected graph data
- node : papers / edge : citation between papers
- task : classifying category of the paper(node classification)
- node features : node degree, paper abstract(GloVe embedding)
이 데이터는 시간이 지남에 따라 새로운 노드들이 추가되는 evolving graph입니다. 실험에서는 2000~2004년 동안의 그래프로 graphsage를 학습한 후, 2005년의 그래프를 evaluation에 사용합니다.
Reddit Dataset
- undirected graph data
- node : reddit post / edge : if same user comments on both
- task : classifying community of the post(node classification)
- node features : embedding of post title, post's comment, score of post, number of comments on the post
이 데이터셋은 reddit post들로 이루어진 데이터셋으로, 어떤 두 개의 post가 있을 때 같은 사용자가 두 개의 post에 동시에 댓글을 남기면 그 두개의 post(node)는 연결됩니다. 이 그래프는 2014년 9월 동안의 232,965개의 포스트(Node)들로 이루어져 있으며, 이 중 처음 20일치의 그래프를 graphsage 학습에 사용했고 나머지 10일치의 그래프를 evaluation에 사용했습니다.
Protein-Protein Dataset(PPI)
이 데이터셋은 앞의 두 개(citation, reddit)와는 다르게 evolving graph는 아닙니다. evolving graph가 아닌 일반적인 그래프에서도 graphsage가 뛰어남을 보이기 위해, 이 데이터셋을 실험에 포함시킨 것으로 보입니다.
이 데이터셋은 22개의 graph로 이루어져 있고, 각 graph는 하나의 세포에 상응합니다. 그리고 graph의 각 Node는 단백질에 상응합니다. task는, 각 node에 상응하는 단백질의 역할을 분류하는 것입니다. 20개의 그래프를 학습에 사용하였고, 나머지 2개의 그래프를 evaluation에 사용합니다.
3.2. Experimental Result
Node classification evaluation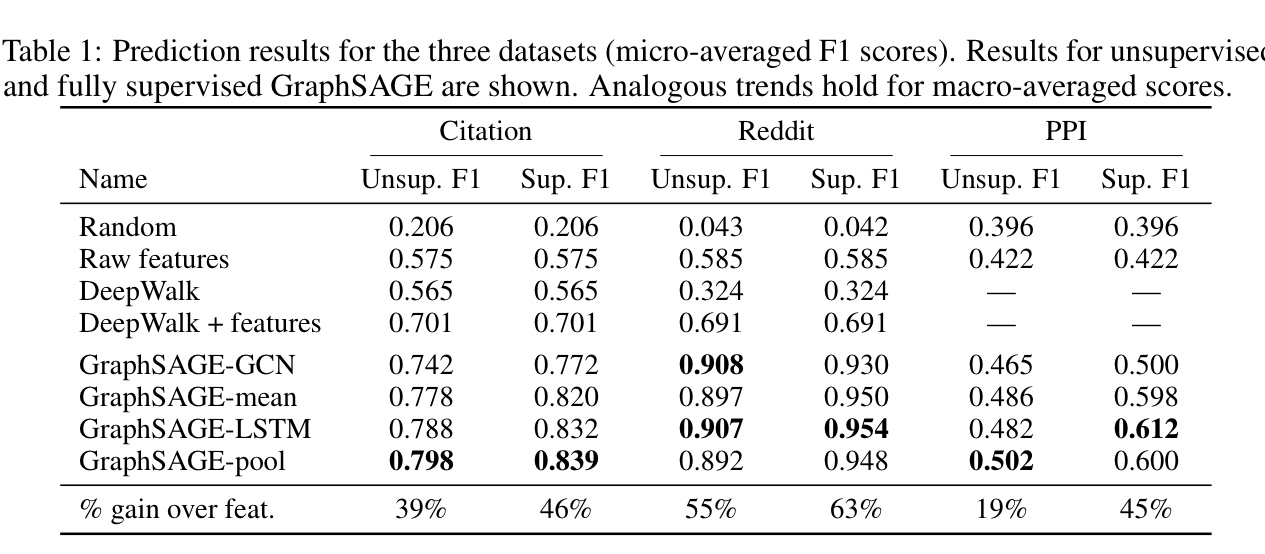
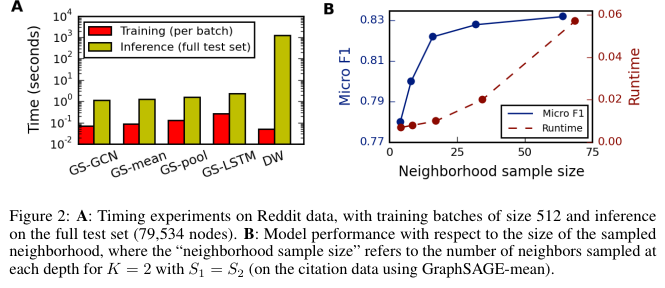
Runtime & parameter sensitivity

쉽게 잘 정리된 글 같아요 도움이 되었습니다 감사합니다 ㅎㅎ