1. 단백질 3D 구조와 시각화란?
단백질 3D 구조(Protein 3D Structure) 란 아미노산 서열이 3차원 공간상에서 접혀서(folding) 만들어내는 고유한 입체구조를 의미한다. 단백질은 이. 입체 구조를 형성해야 비로소 생명 활동에 필요한 기능을 수행할 수 있으므로, 그 구조를 이해하는 것은 매우 중요하다.
단백질 구조 분석 도구
- AlphaFold : 구글 딥마인드가 개발한 인공지능으로, 아미노산 서열만으로 단백질의 3차원 구조를 매우 높은 정확도로 예측한다. AlphaFold Protein Structure Database를 통해 누구나 예측된 구조 데이터를 쉽게 얻을 수 있다.
- py3Dmol : 파이썬 환경, 특히 Jupyter Notebook에서 단백질과 같은 분자의 3D 구조를 시각화할 수 있도록 도와주는 라이브러리이다.
구조 분석의 중요성
- 기능 이해 : 단백질의 구조는 그 기능을 결정한다. 특정 분자와 결합하는 부위(활성 부위, active site) 등을 파악할 수 있다.
- 질병 연구 : 많은 질병이 단백질의 구조 이상과 관련이 있으며, 단백질 구조 분석은 질병의 원인을 밝히는데 결정적인 단서를 제공한다.
- 신약 개발 : 질병 관련 단백질의 3D 구조를 타겟으로 하여 약물 분자를 설계하는 데 활용된다.
2. py3Dmol을 활용한 시각화
본 라이브러리는 Jupyter Notebook과 같은 ipynb 환경에서 작동하므로 미리 준비해두자.
데이터 준비하기
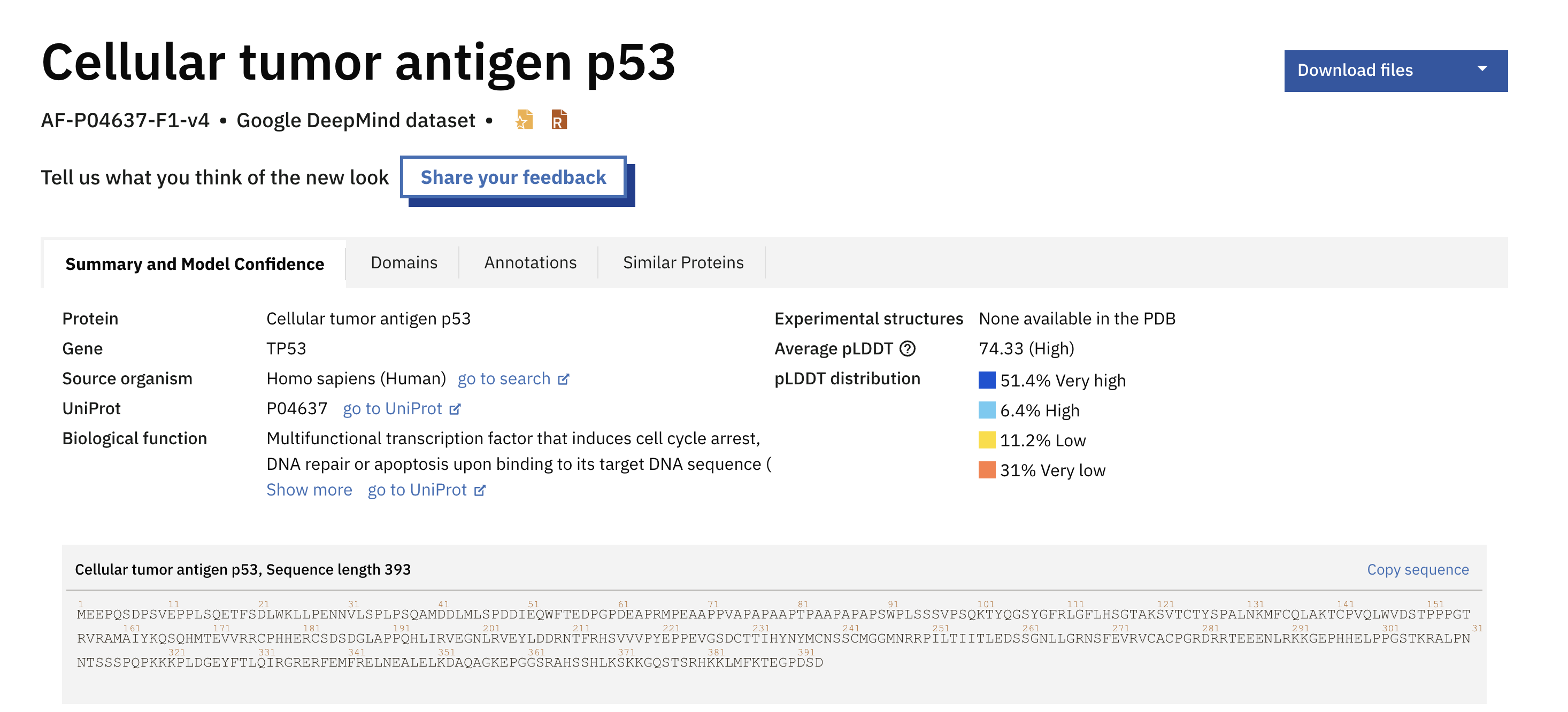
https://alphafold.ebi.ac.uk/
우선 AlphaFold Protein Structure DB 사이트에 접근하여 원하는 데이터를 가져오자.
나는 인간의 p53 단백질을 가져왓고, 단백질 id(Uniprot)는 P04637이다.
저 화면에서 눈여겨 보아야할 것은 Average pLDDT인데, 예측한 구조의 평균 신뢰도를 의미한다.
시각화하기
import py3Dmol
# 뷰어 객체 생성
view = py3Dmol.view(width=800, height=600)
# 파일 열기
with open('AF-P04637-F1-model_v4.pdb','r') as f:
pdb_data = f.read()
view.addModel(pdb_data,'pdb')
view.setStyle({'cartoon':{'color':'spectrum'}})
view.zoomTo()
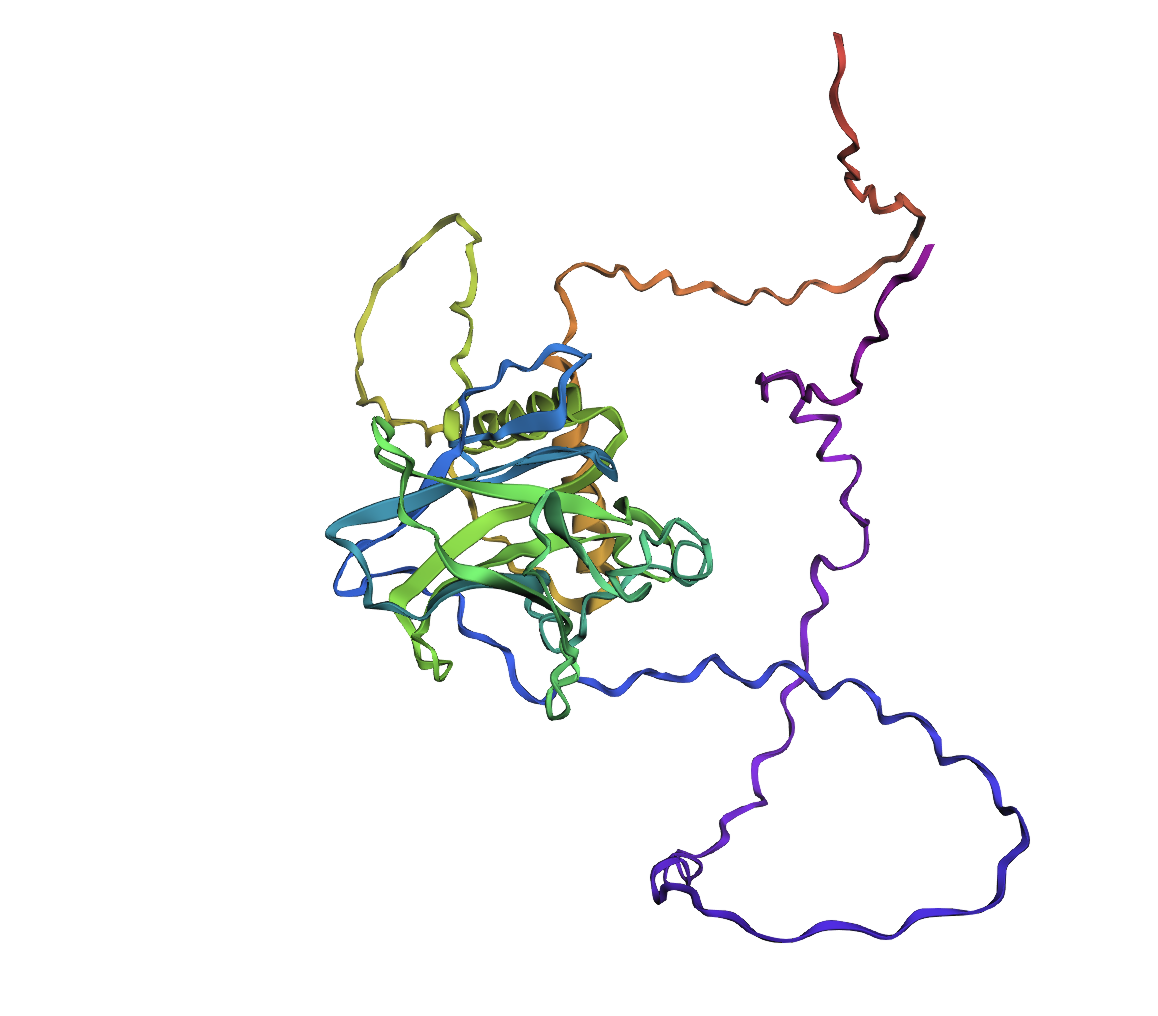
view.show()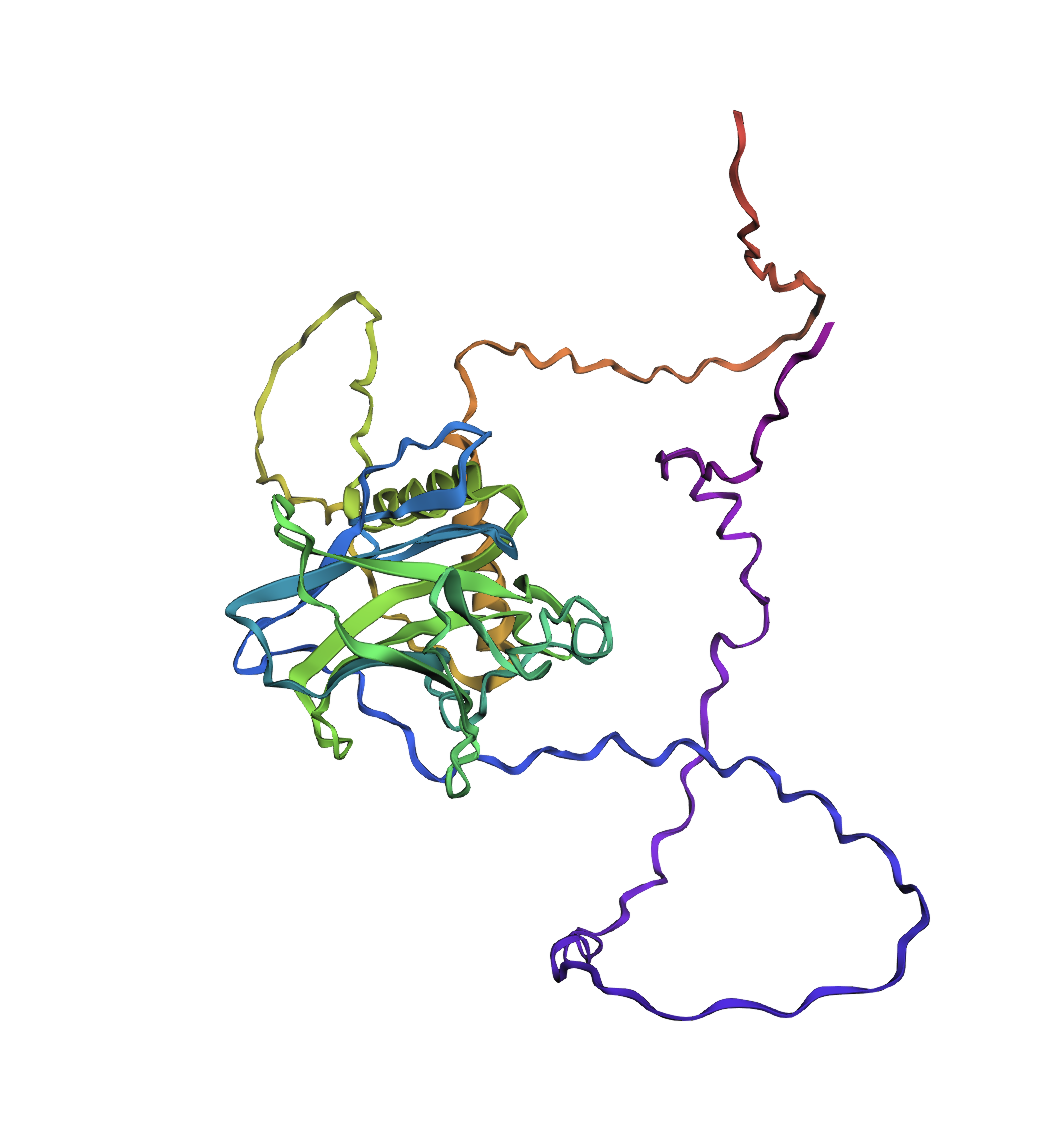
추가적으로 view.setStyle()의 속성을 변경해가며 다른 기준으로 3D 모델링을 할 수 있다.
AlphaFold 신뢰도 기준
# 1. AlphaFold 신뢰도(pLDDT)에 따라 색칠 (bFactor 값 활용)
# 파란색(신뢰도 높음) ~ 빨간색(신뢰도 낮음)
view.setStyle({'cartoon': {'color': 'bFactor'}})
view.zoomTo()
view.show()- 파란색 부분(안정적 코어) : 파란 부분은 예측 신뢰도가 매우 높은 DNA 결합 도메인(DNA-binding domain, DBD) 이다. DNA에 직접 결합하여 유전자 발현을 조절하는 p53의 핵심 기능 부위이다. 대부분의 암 관련 돌연변이가 이 영역에 집중된다.
- 주황색/빨간색 부분(유연한 말단) : 신뢰도가 낮다고 예측 실패가 아닌, 다른 단백질과 상호작용하기 위해 의도적으로 구조가 정해져있지않은 유연한 영역(Flexible Region) 임을 의미한다.
아미노산 종류(Residue Type) 기준
단백질을 구성하는 20가지 아미노산은 각기 다른 화학적 특성을 가지기때문에 , 단백질 표면의 화학적 성질 분포를 시각적으로 이해하는데 도움이 된다.
view.setStyle({'cartoon': {'color': 'spectrum'}})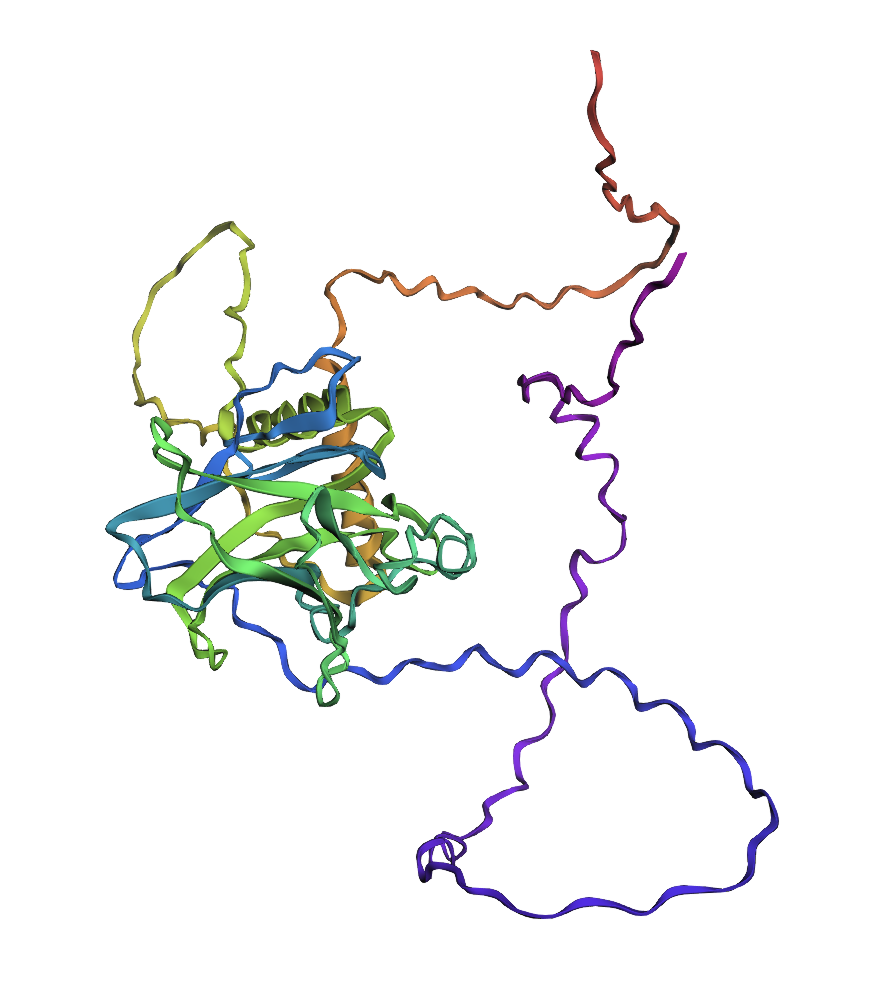
2차 구조(Secondary Structure) 기준
단백질은 Alpha-helix, Beta-sheet, loop 등과 같은 기본적인 2차 구조를 가지고있다.
view.setStyle({'cartoon': {'color': 'ss'}})특정 기능 부위 강조
이번에는 전체를 단색으로 칠하고, 암과 관련된 돌연변이가 자주 발생하는 '핫스팟'부위만 다른 색과 스타일로 지정해보겠다.
# 1. 전체를 연한 회색 카툰으로 초기화
view.setStyle({'cartoon': {'color': 'lightgrey'}})
# 2. 암에서 자주 발견되는 돌연변이 '핫스팟' 잔기(175, 248, 273번)를 공 모양(sphere)으로 강조
hotspot_residues = [175, 248, 273]
view.addStyle({'resi': hotspot_residues},
{'sphere': {'color': 'red'}})
view.show()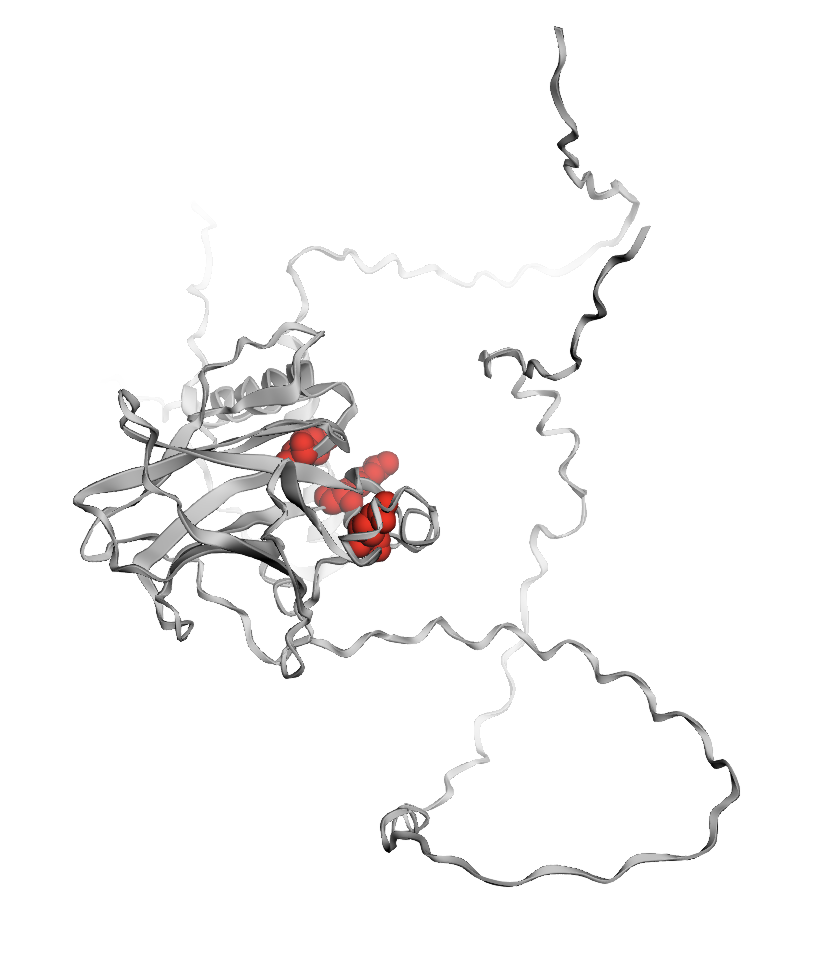
여러 데이터를 가지고 직접 시각화 하면 좋을거같다 :)
