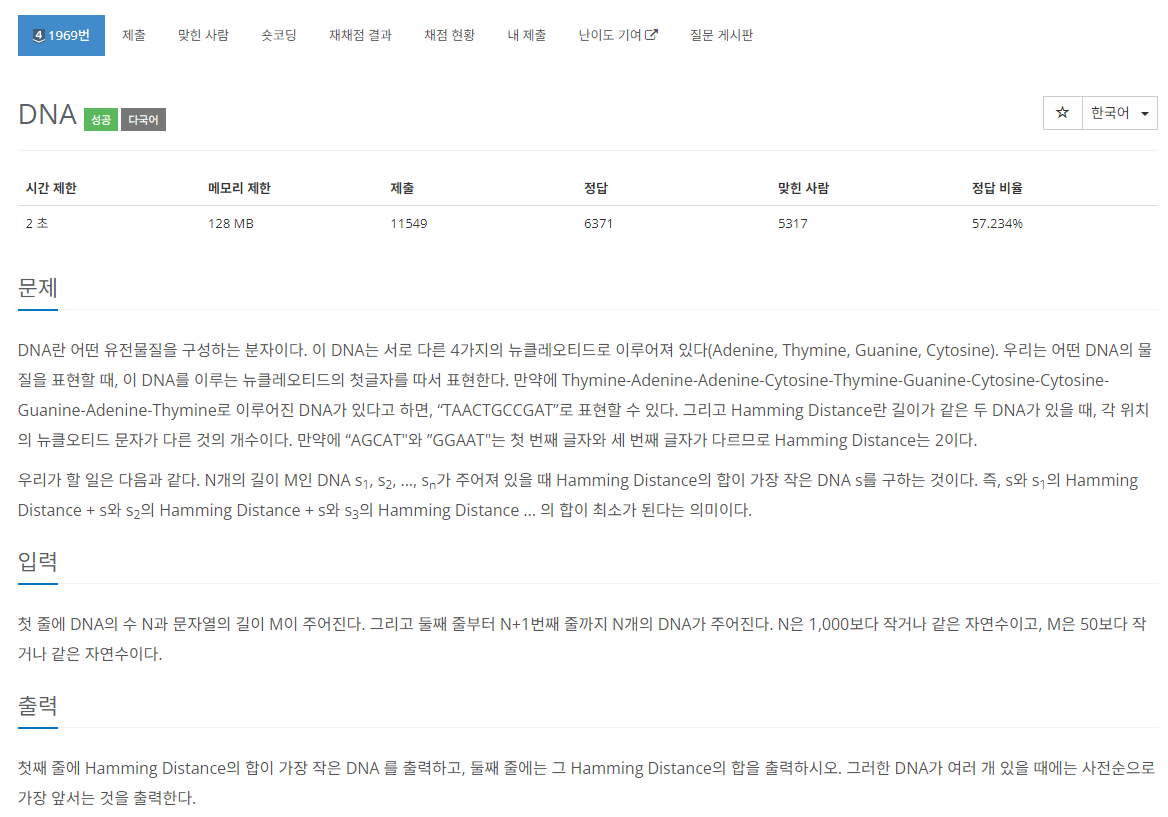
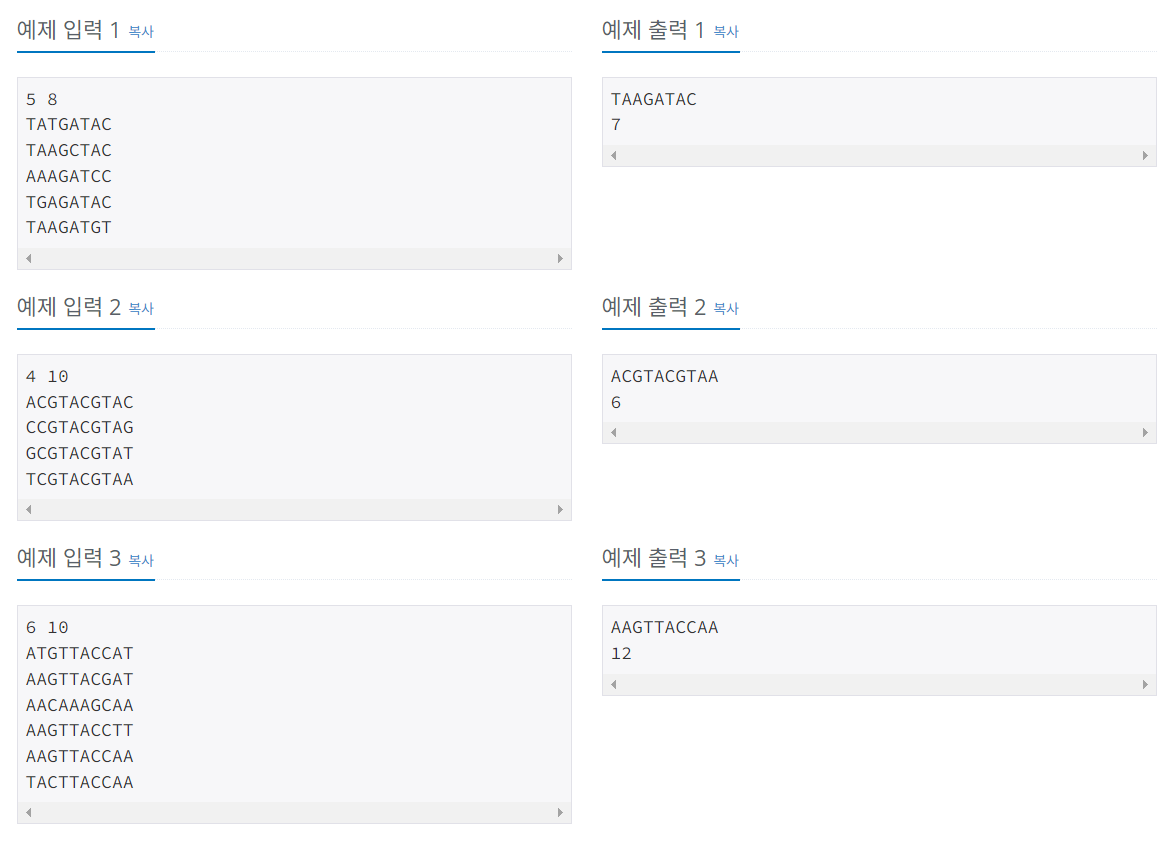
문제
 https://www.acmicpc.net/problem/1969
https://www.acmicpc.net/problem/1969
코드
import java.util.*;
import java.io.*;
class Main {
public static void main(String[] args) throws Exception {
BufferedReader br = new BufferedReader(new InputStreamReader(System.in));
StringTokenizer st = new StringTokenizer(br.readLine());
int N = Integer.parseInt(st.nextToken()); // DNA의 수
int M = Integer.parseInt(st.nextToken()); // 문자열의 길이
//2차원 배열에 입력을 초기화
char[][] arr = new char[N][M];
for (int i = 0; i < N; i++) {
char[] input = br.readLine().toCharArray();
for (int j = 0; j < M; j++) {
arr[i][j] = input[j];
}
}
//4개의 문자의 개수를 Map에 저장
StringBuilder sb = new StringBuilder();
Map<Character, Integer> map = new HashMap<>();
initializeMap(map);
for (int i = 0; i < M; i++) {
for (int j = 0; j < N; j++) {
char cur = arr[j][i];
map.put(cur, map.get(cur) + 1);
}
//등장횟수가 가장 많으면서 사전순으로 앞인 알파벳 구하기
char maxKey = 'Z';
int maxValue = 0;
for (char key : map.keySet()) {
int val = map.get(key);
if (val > maxValue) {
maxKey = key;
maxValue = val;
} else if (val == maxValue) {
if (key < maxKey) {
maxKey = key;
maxValue = val;
}
}
}
sb.append(maxKey);
initializeMap(map);
}
//Hamming Distance 합 구하기
char[] DNA = sb.toString().toCharArray();
int ret = 0;
for (int i = 0; i < N; i++) {
for (int j = 0; j < M; j++) {
if (DNA[j] != arr[i][j]) {
ret++;
}
}
}
sb.append(System.lineSeparator());
sb.append(ret);
System.out.println(sb);
}
private static void initializeMap(Map<Character, Integer> map) {
map.put('A', 0);
map.put('C', 0);
map.put('T', 0);
map.put('G', 0);
}
}문제 이해
- 문제가 딱 보기엔 영어도 많고, 좀 복잡해 보일수도 있는데 두번정도 읽어보면 정확히 문제를 구할 수 있다.
- 입력으로 들어온 N개의 DNA들에 대해서
Hamming Distance의 합을 가장 작게 만드는 dna와 그 합을 구하는 문제이다. - 문제를 이해하기 쉽게 변경해서 생각할 필요가 있다.
Hamming Distance란 두 DNA를 비교하여 각 자리마다 다른 알파벳의 개수를 의미한다.- 따라서 이 합이 최소가 되려면, N개의 DNA들에 대해서
1) 각 자리별로 가장 많이 등장하는 알파벳을 각각 구한 후에
2) 1)에서 구해진 DNA와 입력 DNA들간의 Hamming Distance의 합을 구하면 된다.
풀이 방법
- 위에서 언급한 내용을 그대로 코드로 구현하면 된다.
- 우선
N X M크기의 배열에 모든 입력을 저장한 뒤, 세로 방향으로 각 알파벳의 개수를 구한다. - 이때 각 알파벳의 개수는
Map을 사용하였다. - 이렇게 한줄에 대해서 탐색을 완료한 후에 개수가 가장 크면서 사전순으로 제일 앞에 있는 알파벳을 StringBuilder에 집어넣는다.
- 모든 탐색을 완료했다면, StringBuilder에 있는 값과 N개만큼의 DNA들을 비교해서
Hamming Distance의 합을 구하면 된다.
핵심 포인트
- 이 문제의 핵심포인트는 우선 Hamming Distance의 합을 최소로 하는 DNA가 어떤 것인지 이해하는 것이다. 풀어서 설명하면 N개의 DNA에서 1~M번째의 문자들 중 가장 많이 등장하는 알파벳들의 배열이다.
- 따로 그림은 안그렸지만, 문제를 읽어보면 쉽게 파악할 수 있다.
보완할 점 / 느낀 점
- 문제가 반례가 많은 종류의 문제는 아니고, 문제를 어떻게 이해하고 어떻게 구현하냐가 중요한 문제인 것 같다.
- 어렵진 않았지만, 구현하다보니 좀 길고 복잡해진 감이 있다.
- 다음에 풀떈 Map보다 아스키코드에 맵핑되는 숫자배열을 통해서 개수와 알파벳을 구하도록 구현해봐야겠다.
참고자료
- 좀 더 간략한 구현 : https://lovelyunsh.tistory.com/248
