Unlabeled Data 너는 대체 누구길래 🤔
이게 비운이면 비운이고 행운이면 행운 같은 복불복 게임 테마인 데이터 수집 수난시대를 겪고 있었다. 라벨링 데이터는 없지만 이상하게도 라벨링 되어있지 않은 데이터는 수두룩 깔려있었다.
우선 팀에서 찾은 unlabeled data 리스트는 다음과 같았다.
- Ubenwa Cry Celeb 2023
a. HuggingFace 에서 공개한 아기 울음소리 데이터셋
b. 데이터 수를 체크해보니 자그마치 아니 18000개의 오디오 노다지 밭이었다. - AudioSet(Infant Cry)
a. Google에서 공개한 YouTube 영상에 기반한 오디오 데이터셋- 2,084,320 개의 영상을 오디오 셋으로 묶은 데이터셋
- 이 중 아기 울음소리 영상은 무려 2390개나 포함이 되어 있었다.
Cry Celeb 2023 활용 방안에 대하여
💡 우선 프로젝트 초반이어서 뭔가 무리해 보여도 라벨링 되지 않은 데이터를 뭔가 라벨링 데이터에 편입할 수 있는 방법을 시도해 보았다.(뭐 결국 철퇴됐지만…)프로젝트 초반에 팀에서 MFCC 라는 오디오 특징 벡터를 활용해 보자는 얘기가 나왔다. 물론 모델링 input 관점에서 나온 얘기였지만 라벨링 데이터와 유사도가 높으면 편입해 볼 수 있지 않을까 하는 괜한 가설을 한 번 시도해보고 싶었다.
그래서 다음과 같은 방법으로 유사도를 측정해 보았다.
- 코사인 유사도
- 벡터 간의 거리(Euclidean Distance)
MFCC 란?
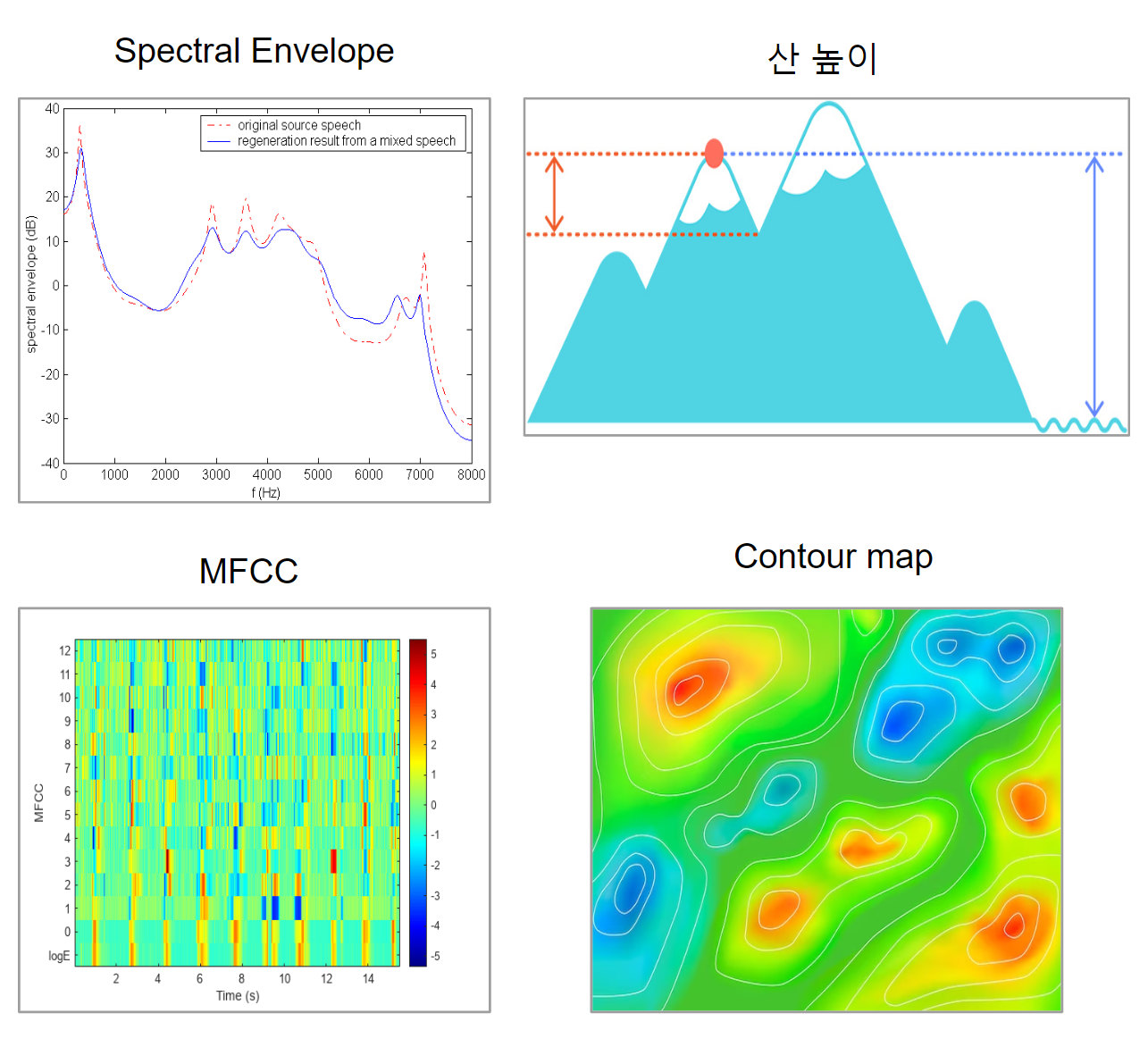
오디오의 Power Spectrum 즉 오디오의 세기를 Time Step마다 특정 계수의 수만큼 나타낸 것이다. 더 쉽게 직관적으로 살펴보면 아래 사진을 볼 수 있다.
위 사진을 보면 Spectral Envelope은 특정 Time Frame에서의 주파수마다 증폭을 나타낸 것이다. 이를 산과 비교해 보면 증폭은 산 높이와 연결 지어 이해해 볼 수 있다. 그리고 MFCC는 산의 전체적인 Contour Map으로 연결 지어 보면 조금 더 직관적으로 이해할 수 있다.
하지만 위 사진은 정확한 묘사는 아니다. 더 자세하고 정확하게 이해하기 위해 Fourier Transform과 Discrete Cosine Transform을 이해해야 하는데 프로젝트 진행을 위해 여기까지 이해하기엔 난이도가 있어서 이 정도로만 이해하고 넘겼다.
코사인 유사도
- 각 클래스마다 MFCC를 구한 후 flatten 시켜 평균 벡터를 구하였다.
- 라벨링 되지 않은 데이터를 1대 1로 라벨링 데이터와 하나씩 비교하는 건 어렵기 때문에 평균 벡터를 구함
-
아래 코드 참조
# hungry audio to MFCCs # MFCC 추출 예 spectro_hungry = [] for path in hungry_path: y, sr = librosa.load(path, sr = 44100) mfccs = librosa.feature.mfcc(y=y, sr=sr) spectro_hungry.append(mfccs) # labeled 와 unlabeled 리사이징 및 백터화 spectro_belly_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_belly] spectro_burp_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_burp] spectro_discomfort_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_discomfort] spectro_hungry_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_hungry] spectro_tired_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_tired] spectro_unlabeled_resized = [np.array(mfcc).flatten()[:160] for mfcc in spectro_unlabeled] # 평균벡터 추출 def mean_vector(vectors): return np.mean(vectors, axis=0) mean_bellypain = mean_vector(spectro_belly_resized) mean_burp = mean_vector(spectro_burp_resized) mean_discomfort = mean_vector(spectro_discomfort_resized) mean_hungry = mean_vector(spectro_hungry_resized) mean_tired = mean_vector(spectro_tired_resized)
-
- 라벨링 되지 않은 데이터를 1대 1로 라벨링 데이터와 하나씩 비교하는 건 어렵기 때문에 평균 벡터를 구함
-
이후 18000 가량의 unlabeled data를 각 클래스의 평균 mfcc 벡터와 코사인 유사도로 비교하였다.
- 아래 코드 참조
# 코사인 유사도 계산 def cosine_similarity(vecA, vecB): return 1 - cosine(vecA, vecB) similarity_result = [] for idx, unlabeled_vector in enumerate(spectro_unlabeled_resized): similarity_bellypain = cosine_similarity(unlabeled_vector, mean_bellypain) similarity_burp = cosine_similarity(unlabeled_vector, mean_burp) similarity_discomfort = cosine_similarity(unlabeled_vector, mean_discomfort) similarity_hungry = cosine_similarity(unlabeled_vector, mean_hungry) similarity_tired = cosine_similarity(unlabeled_vector, mean_tired) # 결과 값을 딕셔너리로 저장 similarity_result.append({ 'Index': f'unlabeled_audio{idx}', 'Bellypain': similarity_bellypain, 'Burp': similarity_burp, 'Discomfort': similarity_discomfort, 'Hungry': similarity_hungry, 'Tired': similarity_tired })
-
코사인 유사도 결과
- 코사인 유사도 0.90 이상 기준
Max_Column Bellypain 647개 Tired 382개 Hungry 145개 Discomfort 96개 Burp 26개- 복통과 피곤함이 가장 유사도가 높게 나온 걸 확인할 수 있었다.
벡터간 거리
-
평균 MFCC 벡터와 unlabeled data의 MFCC를 벡터 간 거리를 계산해 보았다.
- 아래 코드 참조
# 벡터간 거리 측정 def euclidean_distance(vec1, vec2): return np.linalg.norm(np.array(vec1) - np.array(vec2)) distance_result = [] for idx, unlabeled_vector in enumerate(spectro_unlabeled_resized): distance_bellypain = euclidean_distance(unlabeled_vector, mean_bellypain) distance_burp = euclidean_distance(unlabeled_vector, mean_burp) distance_discomfort = euclidean_distance(unlabeled_vector, mean_discomfort) distance_hungry = euclidean_distance(unlabeled_vector, mean_hungry) distance_tired = euclidean_distance(unlabeled_vector, mean_tired) # 결과 값을 딕셔너리로 저장 distance_result.append({ 'Index': f'unlabeled_audio{idx}', 'Bellypain': distance_bellypain, 'Burp': distance_burp, 'Discomfort': distance_discomfort, 'Hungry': distance_hungry, 'Tired': distance_tired }) -
코사인 유사도와 동일한 스케일(0과 1사이)로 보기 위해 Inverse Scaling을 진행하였다.
- Min Max scaling을 조금 변형해서 inverse scaling으로 변환 (코드 출처: ChatGPT)
# 최솟값 최댓값 지정 min_val = 262 max_val = 6253 # Inverse Scaling 함수 정의 def inverse_scaling(x, min_val, max_val): x = np.clip(x, min_val, max_val) return (max_val - x) / (max_val - min_val)
-
코사인 유사도와 벡터 간 거리를 DataFrame으로 합친 후 가중 합을 내어 점수를 내어 보았다.
a. 벡터 간 거리가 코사인 유사도보다 분산이 높은 이유로 가중치를 0.9로 주어 점수에 반영
b. 합산 결과Max_Column Bellypain_score 508 Tired_score 164 Discomfort_score 62 Hungry_score 28 Burp_score 19
결과를 보면 복통과 피곤함이 유사도가 높은 걸 확인할 수 있었다. 합산 후 차이는 있었지만 코사인 유사도와 비슷한 양상을 볼 수 있었다.
회고
-
우선 팀원들과 회의 후 이 방법은 쓰지 않기로 했다. 그 이유는 다음과 같았다.
-
객관적이지 않았다.
a. 육아 관련 전문가가 확인하지 않았고 순전히 우리만의 가설로 진행했다는 점에서 객관적이지 못했다.
b. 하지만 이 가설을 전문가들에게 실험은 해볼 수 있지 않을까 하는 호기심은 들었다. 이러한 방법으로 혹은 보다 더 좋은 방법으로 유사도를 측정해 정말 필요한 데이터만 추려내서 라벨링을 할 수 있다면 데이터 라벨러도 더 확실히 라벨을 할 수 있을뿐더러 라벨링 양이 줄기 때문에 수고가 덜어지지 않을까 하는 생각을 해보게 되었다. -
MFCC 만으로 유사도를 판단하기엔 부족해 보인다.
a. 오디오 데이터는 MFCC 뿐만 아니라 다른 특징들도 있기 때문에 더 다양한 feature 시각에서 유사도를 측정해야 더 robust 한 유사도 값이 나오지 않을까 생각이 된다. -
Unlabeled data와 labeled data의 오디오 길이 차이가 컸다.
a. Unlabeled data는 1초에서 2초가량의 오디오가 대부분이었고 labeled data는 5초 이상의 데이터였다. labeled data를 truncate 해 비교하는 건 정보의 손실이 컸기 때문에 사실 유사도 측정이 이상적이라고 보기는 어려웠다.
-
-
unlabeled data를 루프를 돌려 5초로 맞추고 평균 벡터대신 K-means clustering을 통해 Centroid로 비교해 봤으면 더 좋은 측정이 됐을 것 같다. 하지만 여전히 이건 가설일 뿐이다.
-
AudioSet 활용 방안을 팀과 더 회의를 해봤으면 하는 아쉬움이 있었다.
👉👉 다음 챕터 읽으러 가기