library(party)
library(caret)
# data sampling
data1 <- iris[sample(1:nrow(iris), replace=T),]
data2 <- iris[sample(1:nrow(iris), replace=T),]
data3 <- iris[sample(1:nrow(iris), replace=T),]
data4 <- iris[sample(1:nrow(iris), replace=T),]
data5 <- iris[sample(1:nrow(iris), replace=T),]
# 예측모형 생성
citree1 <- ctree(Species~., data1)
citree2 <- ctree(Species~., data2)
citree3 <- ctree(Species~., data3)
citree4 <- ctree(Species~., data4)
citree5 <- ctree(Species~., data5)
# 예측수행
predicted1 <- predict(citree1, iris)
predicted2 <- predict(citree2, iris)
predicted3 <- predict(citree3, iris)
predicted4 <- predict(citree4, iris)
predicted5 <- predict(citree5, iris)
# 예측모형 결합하여 새로운 예측모형 생성
newmodel <- data.frame(Species=iris$Species,
predicted1,predicted2,predicted3,predicted4,predicted5)
head(newmodel)
newmodel
# 최종모형으로 통합
funcValue <- function(x) {
result <- NULL
for(i in 1:nrow(x)) {
xtab <- table(t(x[i,]))
rvalue <- names(sort(xtab, decreasing = T) [1])
result <- c(result,rvalue)
}
return (result)
}
newmodel
# 최종 모형의 2번째에서 6번째를 통합하여 최종 결과 생성
newmodel$result <- funcValue(newmodel[, 2:6])
newmodel$result
# 최종결과 비교
table(newmodel$result, newmodel$Species)
head(iris)
# 70% training데이터, 30% testing데이터로 구분
idx <- sample(2, nrow(iris), replace=T, prob=c(0.7, 0.3))
trData <- iris[idx == 1, ]
nrow(trData)
teData <- iris[idx == 2, ]
nrow(teData)
library(randomForest)
# 랜덤포레스트 실행 (100개의 tree를 다양한 방법(proximity=T)으로 생성)
RFmodel <- randomForest(Species~., data=trData, ntree=100, proximity=T)
RFmodel
# 시각화
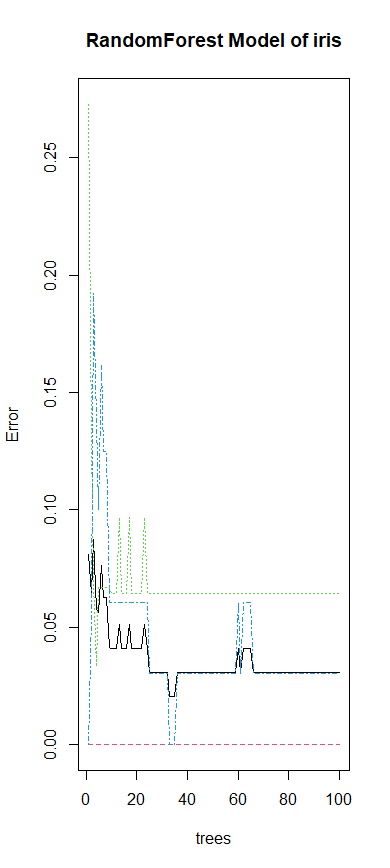
plot(RFmodel, main="RandomForest Model of iris")
# 모델에 사용된 변수 중 중요한 것 확인
importance(RFmodel)
# 중요한 것 시각화
varImpPlot(RFmodel)
# 실제값과 예측값 비교
table(trData$Species, predict(RFmodel))
# 테스트데이터로 예측
pred <- predict(RFmodel, newdata=teData)
# 실제값과 예측값 비교
table(teData$Species, pred)
# 시각화
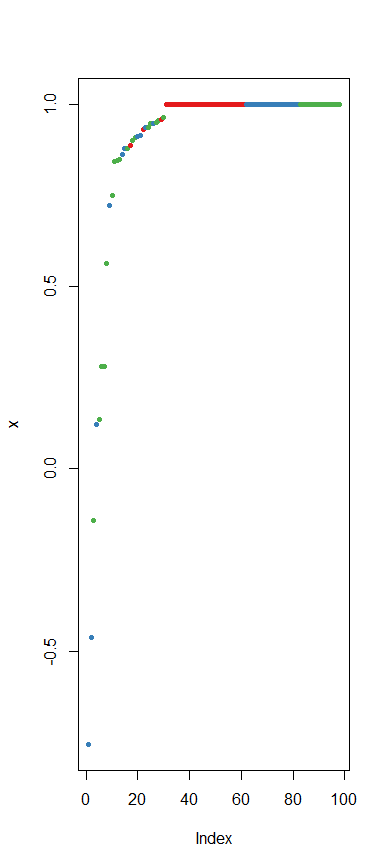
plot(margin(RFmodel, teData$Species))
# 모델에 사용된 변수 중 중요한 것 확인
importance(RFmodel)
# 중요한 것 시각화
varImpPlot(RFmodel)
# 실제값과 예측값 비교
table(trData$Species, predict(RFmodel))
# 테스트데이터로 예측
pred <- predict(RFmodel, newdata=teData)
# 실제값과 예측값 비교
table(teData$Species, pred)
# 시각화
plot(margin(RFmodel, teData$Species))
data(iris)
model <- randomForest(Species ~ ., data = iris)
model
model2 <- randomForest(Species ~ ., data = iris,
ntree = 300, mtry = 4, na.action = na.omit)
model2
model3 <- randomForest(Species ~ ., data = iris,
importance = T, na.action = na.omit)
importance(model3)
varImpPlot(model3)
x1 <- 0.5; x2 <- 0.5
e1 <- -x1 * log2(x1) - x2 * log2(x2)
e1
x1 <- 0.7; x2 <- 0.3
e2 <- -x1 * log2(x1) - x2 * log2(x2)
e2
ntree <- c(400, 500, 600)
mtry <- c(2:4)
param <- data.frame(n = ntree, m = mtry)
param
for(i in param$n) {
cat('ntree =', i, '\n')
for(j in param$m) {
cat('mtry =', j, '\n')
model_iris <- randomForest(Species ~ ., data = iris,
ntree = i, mtry = j, na.action = na.omit)
print(model_iris)
}
}


