INTRO
첫 논문 리뷰 포스트네요, unsupervised learning을 deformable image registration에 적용하는 방법에 대해 연구한 An Unsupervised Learning Model for Deformable Medical Image Registration[1] 논문을 리뷰해보도록 하겠습니다.
해당 포스트에는 Concept과 CNN Architecture 부분만 있습니다. 자세한 내용은 논문을 참고해주세요!
Deformable Image Registration
사람의 뇌를 촬영한 CT영상을 예로 들어 보겠습니다. CT영상은 찰나의 순간을 담고 있기 때문에, 같은 사람의 뇌를 촬영했다 하더라도 촬영할 때마다 다른 형상을 가질 수 밖에 없습니다. 만약 뇌의 종양이 얼마나 진행되었는지를 확인해야 하는 상황이라면, 과거와 현재의 CT영상을 비교하는 일은 생각보다 어려워질 수 있습니다. 이러한 상황에서, 자동으로 두 CT영상의 형상을 비슷하게 맞춰줄 수 있는 기술을 deformable image registration이라고 합니다. 그림을 통해 예를 들어 보겠습니다.
 [2]에서 발췌,
[2]에서 발췌,
왼쪽 -> Moving image, 변환시킬 영상
중앙 -> Fixed image, 고정(목표) 영상
오른쪽 -> 왼쪽 이미지를 변환시킨 영상
Contribution
논문 초반에 저자들이 자신의 연구에 대해 열심히 홍보하고 있지만, 제가 생각하는 이 논문의 기여점은 아래와 같습니다.
- 지도 학습 기반이 아닌, 비지도 학습 기반의 딥러닝 모델을 통해서 별도의 라벨링 없이 학습을 할 수 있다.
- 전통적인 방법이 아닌(어떤 방법인지는 뒤에서 설명하겠습니다) deep neural network(CNN 기반)를 이용해 deformable image registration을 진행했고, 이전 방법과 성능은 비슷하지만 훨씬 더 빠른 실행속도를 보였다.
Problem Definition
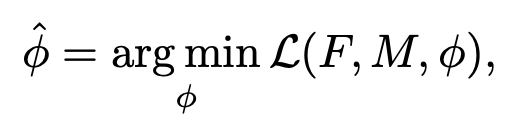
여기서 는 위에서 말한 고정(Fixed) 영상, 은 이동할(Moving) 영상이라고 할 수 있습니다. 그리고 는 registratin field의 value들이라고 할 수 있습니다. 여기서 registration field란, 아래와 같은 vector field를 의미합니다.
 [3]에서 발췌
[3]에서 발췌
그럼 위 식에서, 최소화해야 할 은 무엇일까요?
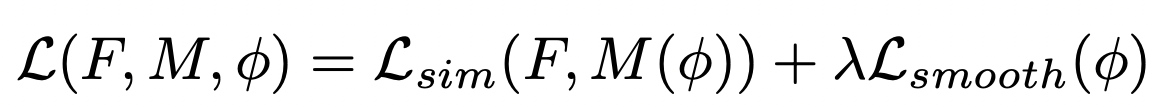
은 위와 같은 Loss Function을 의미하게 되는데요, 여기서 는 (Moving) 이미지를 이동시킨 영상이라고 이해하시면 됩니다. 의 경우에는 F(고정) 영상에 M(움직일) 영상이 얼마나 잘 정합되었는가를 의미하게 됩니다. 그리고 는 registration field의 값을 조금 smooth하게 바꿔주는 역할을 합니다. 이는, 단순히 을 만족시키기 위해 비현실적인 registration field를 만들어내는 현상을 방지해주는 역할을 하는데요. 결과적으로 주어진 데이터들을 정확하게 학습하는 것을 방해하면서 현실적인 registration field를 만들도록 도와주기 때문에 regularization 효과를 가진다, 라고 볼 수 있습니다. 두 식에 대해서는 뒤에서 조금 더 자세하게 말씀드리도록 하겠습니다.
Overview of Method
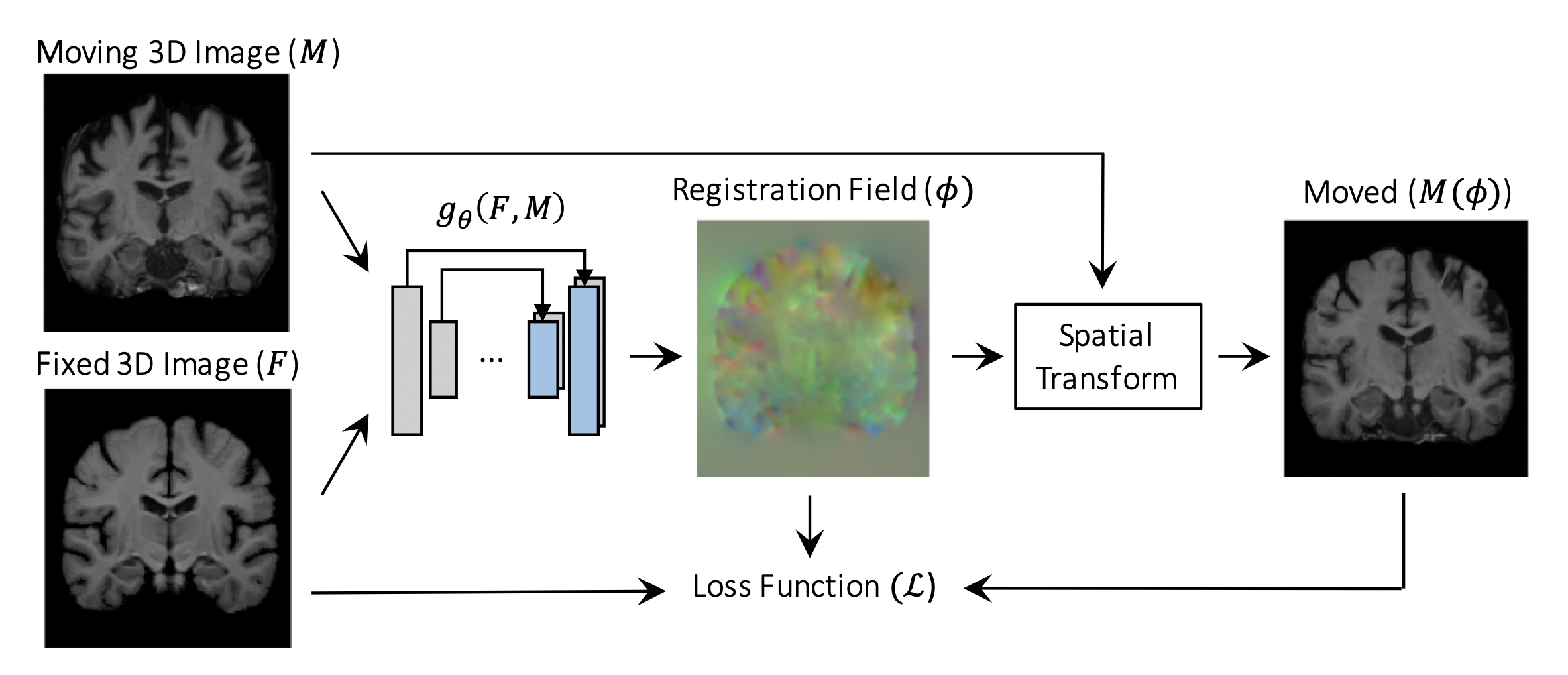
전체적인 모델 구조는 위와 같습니다. 일단, Moving 3D Image와 Fixed 3D Image를 네트워크 에 입력합니다. 여기서 이라는 정의가 생소할 수도 있는데, 그냥 이미지 F와 M을 네트워크에 입력한다, 라는 의미로 받아들일 수 있습니다. 네트워크의 결과로 Registration Field 가 출력됩니다. 그리고, 이 Registration Field를 이용하여 Moving 3D Image를 Transformation하구요, 그 후에 Loss Function을 통해 Transformation 시킨 Moved Image를 Fixed 3D Image와 비교하게 됩니다. 네트워크 앞단부터 차례대로 알아보도록 하겠습니다.
VoxelMorph CNN Architecture
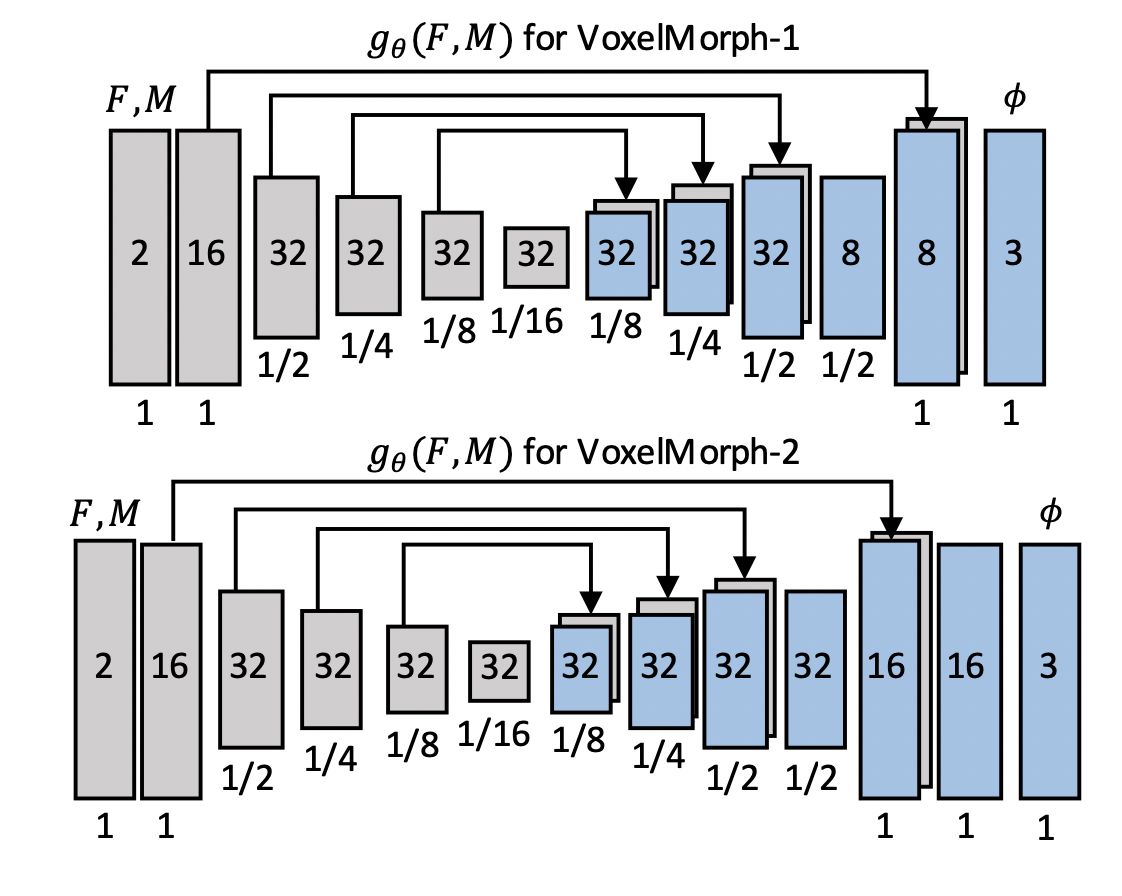
위 그림에서 에 해당하는 부분의 네트워크입니다. 딥러닝을 이용한 의학영상처리에서 유명한 3d-unet[4]과 크게 다르지 않은 네트워크 구조를 가지고 있습니다. 먼저, 입력은 가 됩니다. 아무래도 3d voxel 연산이 메모리를 굉장히 많이 잡아먹다보니, batch size는 1로 고정하게 됩니다.
그 이후에, encoding stage를 지나게 되는데요. kernel로 이루어진 convolutional layer를 지나게 됩니다. 각 레이어 사이에서 이용하는 activation function은 leaky-relu라고 하네요. stride를 적용하기 때문에, 각 layer에서 volume의 size가 반으로 감소하는 것을 확인할 수 있습니다. 여기서 한 가지 유의할 점은, 마지막 layer의 receptive field가 픽셀이 움직일 수 있는 최대 이동거리보다는 같거나 커야 한다는 것입니다. Moving image에서 어떤 위치에 있는 pixel의 ground-truth인 transformation vector의 거리가 receptive field보다 작다면, 대응되는 fixed image의 픽셀을 고려할 수 없기 때문에, 네트워크가 제대로 학습할 수 없게 됩니다.
Decoder stage에서는, encoder stage의 kernel과 동일한 kernel을 이용해서, deconvolutional 연산을 수행하게 됩니다. activation layer로는 동일하게 leaky-relu를 이용하고, skip connection을 통해 부족한 정보량을 보충하게 됩니다. 마지막에는 volume size가 input과 동일한, 3 channel output을 가지게 됩니다. 이는 3차원 공간의 voxel을 transformation vector가 3-dimension이기 때문이겠죠?
논문에서는 CNN architecture로 두 개의 version을 소개하고 있는데요, 두 version의 차이점은 마지막 layer에서 channel 수를 몇 개로 가져가느냐 입니다. input과 동일한 volume size를 가진 output을 내야하기 때문에, output에 가까운 layer에서는 convolutional layer를 통과할 때마다 엄청 많은 연산이 필요하게 됩니다. VoxelMorph-1에서는 그래서 마지막 layer의 channel수를 조금 줄였고, VoxelMorph-2에서는 마지막 layer의 channel수를 조금 늘렸습니다. layer가 깊어지고 channel의 수가 늘어나면, 왠만해서 accuracy가 높아지기 때문에, time complexity와 accuracy간의 trade-off가 존재하게 됩니다. 용도에 맞게 두 개의 model 중 하나를 선택할 수 있게끔 만든 것으로 보입니다.
[1]An Unsupervised Learning Model for Deformable Medical Image Registration
[2]3차원 뇌 자기공명 영상의 비지도 학습 기반 비강체 정합 네트워크
[3]Esmraldi: efficient methods for the fusion of mass spectrometry and magnetic resonance images
[4]3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
