
🔬 U-Net이란?
U-Net은 의학 영상 segmentation을 위해 만들어진 CNN 기반의 네트워크 구조로,
이미지의 각 픽셀을 클래스별로 분류해서 정밀한 분할(Segmentation)을 수행하는 데 특화되어 있어.
이름 그대로 네트워크 모양이 U자형 구조를 하고 있어서 "U-Net"이라고 불려
Abstract
봅 논문에서는, 사용 가능한 주석 데이터를 보다 효율적으로 활용하기 위해 강력한 데이터 증강(data agumetation)을 활용하는 네트워크와 학습 전략을 제안한다.
논문의 아키텍쳐는 문잭을 포착하는 수축경로(contracting path)와, 정확한 위치 정보를 가능하게 해주는 대칭적인 확장 경로 (expanding path)로 구성된다.
이러한 네트워크가 매우 적은 수의 이미지만으로도 end-to-end로 학습될 수 있다.
1.Introduction
기존 CNN의 문제점은
-
classification에 특화되어 있다.
하나의 이미지에 하나의 클래스(label)만 예측
-
의학 영상 처리(biomedical image processing)에서는 이미지 전체에서 각 픽셀별로 클래스를 예측해야한다.
Localization(위치 정보)가 중요함
그리고 CNN과 sliding-window 융합 방식은
- Ciresan은 각 픽셀 주변의 작은 패치(patch)를 보고 그 픽셀의 클래스를 예측하는 네트워크를 만들었다. → 이 방식을 local prediction이 가능하다는 장점이 있고 패치 단위 학습이라 데이터도 많아질 수 있다.
하지만 CNN+sliding-window 방식의 문제점은
-
느리다.
각 픽셀마다 패치를 하나씩 예측해야 하니까 계산량이 많고 중복도 많다.
-
문맥 vs 정확도 트레이드오프
- 큰 패치 : 문맥은 넓게 보지만, pooling이 많아져서 위치 정확도는 감소한다.
- 작은 패치 : 위치는 정확하지만 문맥은 거의 없다.
Fully Convolutional Network (FCN)
여러 계층의 feature를 조합해서 위치 정보와 문맥 정보를 동시에 활용하는 구조이다.
U-net
- 본 논문에서는 FCN 기반 구조를 바탕으로 데이터가 적어도 잘 작동하고 더 정밀한 segmentation이 가능한 구조를 제안했따.
- 구조 특징 :
- 수축 경로(contracting path) + 확장 경로(expanding path)
- 업샘플링을 통해 해상도 복원
- 고해상도 feature를 upsample된 출력과 결합해서 정밀한 예측 가능
기존 방식(CNN, sliding-window)은 위치 정보나 속도 면에서 한계가 있었고, 이를 해결하기 위해 U-Net은 FCN 구조를 확장하여 정밀하고 빠른 segmentation을 수행할 수 있게 만들었다.
2 Network Architecture
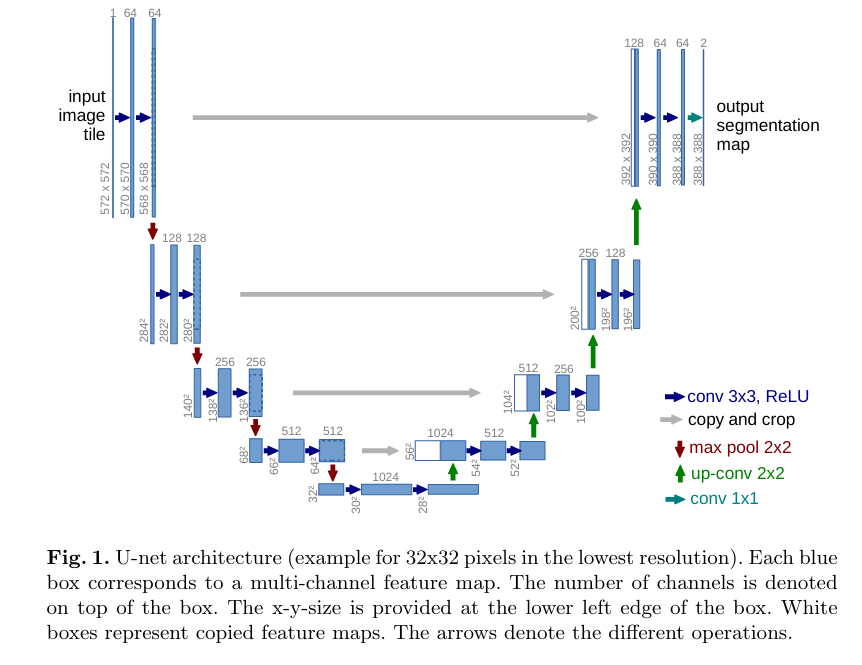
U자형으로 생겨서 U-net이고 중간을 기준으로 왼쪽은 Contracting path(수축 경로)이고 오른쪽은 Expansive path(확장 경로)
1. Contracting Path(수축 경로)
- 기존 CNN 구조와 유사하다.
- 입력 이미지로부터 특징을 추출하는 역할
- 단계 :
- 3x3 컨볼루션 2번 (패딩 없음, valid convolution)
- 각각 ReLU 활성화 함수 적용
- 2x2 Max Pooling (Stride=2)로 다운샘플링
- 각 단계마다 채널 수는 2배씩 증가 ex ) 64 → 128 → 256 → 512 → 1024 (2배씩 채널 수가 증가되게 모델이 설계됨, max pooling을 통해서 공간은 2배씩 줄어들고 channel은 2배씩 늘어나는데 성능이 좋아서 이렇게 쓴다함)
`이 단계에서는 이미지 크기는 줄어들고 대신 더 많은 특징을 출력할 수 있도록 high-level feature을 학습함
2. Expansive path(확장 경로)
- 이미지의 해상도를 점차 복원하는 역할
- 단계 :
- upsampling : feature map 크기를 2배로 확대
- 2x2 up-convolution (transposed convolution)” 적용 → 채널 수 절반으로 줄임
- 같은 위치의 contracting path에서 나온 feature map을 crop해서 concat함
- 왜 crop? ⇒ valid conv라서 공간 크기가 다르기 때문에
- 이어서 3x3 conv x 2 + ReLU
즉, contracting path에서 얻은 고해상도 특징을 upconv를 통해 복원하면서 같이 붙여주는 구조이다.
3. output layer
- 최종 출력에서 1x1 convolution을 사용하여 각 픽셀을 클래스 수만큼의 채널로 매핑함 → 각 픽셀에 대해 softmax 분류 수행
ex) 세포 분할이라면 각 픽셀에 대해 ‘세포냐 아니냐”를 분류(2 class)
4. 전체 구조 요약
| 단계 | 연산 | 해상도 변화 | 채널 수 변화 |
|---|---|---|---|
| 수축 경로 | Conv3×2 + MaxPool | ↓ 작아짐 | ↑ 증가 |
| 최하단 | Conv3×2 | 최저 해상도 | 최고 채널 |
| 확장 경로 | Upsample + UpConv + Concat + Conv3×2 | ↑ 커짐 | ↓ 감소 |
| 출력층 | 1×1 Conv | 입력과 같은 크기 | 클래스 수 |
- 총 23개의 convolution layer로 구성됨
U-net은 수축 경로에서 특징을 추출하고 확장 결로에서 해상도를 복원하면서 segmentation map을 출력하는 구조다. skip connection을 통해 정밀한 픽셀 예측이 가능해진다.
3. training
1) 학습 방법 개요
- 입력 이미지와 그에 대응되는 분할 정답 이미지(segmentation map) 을 가지고 딥러닝 프레임 워크인 (caffe)에서 SGD를 이용해 네트워크를 학습시킨다.
2) 출력 이미지가 작아진다
- U-Net에서는 padding을 하지 않는 valid convolution을 사용함
- Conv 연산을 할 때 입력보다 출력이 작아지는 구조
- 그래서 출력 segmentation map은 입력보다 경계(border)만큼 작음
3) GPU 메모리 효율을 위한 전략
- 연산 효율을 위해 batch size보다 큰 입력 tile을 사용하는게 낫다
- 그래서 batch size는 1 이미지 하나씩 학습하면서 한 번에 더 많은 공간 정보를 활용하도록 설정함
4) 모멘텀(momentum) 설정
- 모멘텀 값은 0.99로 설정
- 이전에 본 학습 샘플들의 경향을 현재의 weight 업데이트에 많이 반영하겠다는 뜻
- 안정적으로 최적화가 진행되도록 도움을 줌
5) Loss Function 구성
- softmax + Cross Entropy
- 픽셀 단위로 softmax 연산을 수행
- cross entropy 손실을 적용해 모델을 훈련시킨다.
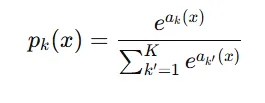
- a_k(x): 픽셀 위치 x의 채널 k에서의 활성화 값
- p_k(x): 그 픽셀이 클래스 k일 확률
- softmax 결과는 확률처럼 해석 가능
cross Entropy 손실 함수
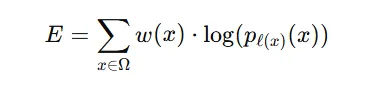
- ℓ(x): 해당 픽셀의 정답 클래스
- w(x): 픽셀별 가중치 (→ 이게 아주 중요!)
6) 픽셀별 가중치 맵 (Weight Map)
U-Net은 그냥 평등하게 학습하지 않음!
특정 픽셀에 더 높은 중요도(가중치)를 부여해서 더 집중적으로 학습하게 만듦
그 이유는?
- 클래스 불균형 문제 해결(ex. 배경이 너무 많고, 세포는 적음)
- 닿아 있는 세포들 사이의 경계를 잘 학습하도록 유도
가중치 계산 공식 :
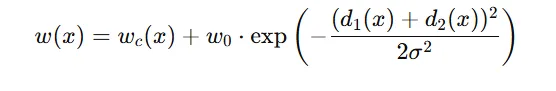
- wc(x): 클래스 간 비율 보정을 위한 기본 가중치
- d1(x),d2(x): 픽셀 x가 가장 가까운 두 셀 경계까지의 거리
- w0=10, 5σ=5: 실험에서 사용된 값
→ 이렇게 하면 경계 근처에 있는 픽셀의 손실 값이 커지도록 설계됨 → 경계 학습 강화
7) 가중치 초기화 (Weight Initialization)
- 딥러닝 네트워크가 깊어질수록, 일부 레이어는 활성화가 너무 크거나 너무 작아져서 죽어버릴 수도 있음.
- 이를 방지하기 위해 He Initialization 방식 사용:
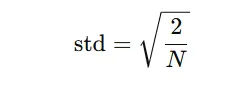
- N : 해당 레이어의 입력 노드 수
- 예 : 3x3 conv에서 이전 채널이 64개라면 → N = 9 x 64 = 576
→ 이렇게 하면 각 레이어의 출력 분산이 비슷하게 유지되며 안정적으로 학습됨
요약 정리
| 항목 | 설명 |
|---|---|
| 학습 도구 | Caffe + SGD |
| 입력/출력 크기 | unpadded conv 사용 → 출력이 입력보다 작아짐 |
| 메모리 전략 | batch size = 1, 큰 타일 사용 |
| 모멘텀 | 0.99 → 이전 업데이트 반영 높임 |
| 손실 함수 | 픽셀-wise softmax + weighted cross entropy |
| 가중치 맵 | 경계 강조, 클래스 불균형 보정 |
| weight init | He initialization (표준편차 √(2/N)) 사용 |
3.1 Data Augmentation
U-Net 주요 목표 중 하나 :
“아주 적은 수의 주석 이미지로도 네트워크가 잘 학습되게 하는 것”
그걸 가능하게 해주는 핵심 전략이 바로 이 data-augmentation이야
- Shift (이동)
- 이미지 위치를 조금씩 움직임 → 위치 변화에 대한 불변성(robustness) 학습
- 이미지 위치를 조금씩 움직임 → 위치 변화에 대한 불변성(robustness) 학습
- Rotation (회전)
- 세포는 다양한 각도로 놓일 수 있으니까 → 회전해도 잘 인식하도록 만듦
- 세포는 다양한 각도로 놓일 수 있으니까 → 회전해도 잘 인식하도록 만듦
- Elastic Deformation (탄성 변형) ← 핵심!
- 이미지를 부드럽게 휘게 하는 변형
- 세포 조직처럼 말랑말랑하고 유동적인 구조에 매우 적합한 증강 방식
✔️ 생의학 영상에서는 이런 비정형적인 변형이 실제로 자주 나타남 → 이걸 미리 학습시켜야 실제 상황에서도 잘 작동함
탄성 변형(elastic deformation) 방식
- 3x3 격자(grid) 위에 무작위로 이동 벡터(displacement vecotr)를 생성
- 이 벡터는 표준편차 10 픽셀의 정규분포(Gaussian)에서 샘플링됨
- 이후 bicubic interpolation을 통해 모든 픽셀의 변형을 부드럽게 계산
즉, 이미지를 자엽스럽게 “우글우글” 휘게 만들어서 네트워크가 그런 상황에도 견디게 학습시키는 거야
Dropout도 추가로 사용함
- contracting path의 마지막에 dropout layer를 삽입
- 이는 간접적인 데이터 증강 역할도 하며 과적합 방지에도 효과적
4. Experiments
| 항목 | 설명 |
|---|---|
| 실험 데이터 | 신경세포 EM, U373 위상차, HeLa DIC 영상 |
| 데이터 수 | 매우 적음 (20~30장 수준) |
| 방법 | U-Net + 강력한 데이터 증강 |
| 결과 | 기존 최고 모델들보다 압도적 성능 |
| 성능 지표 | Warping error, IoU (Intersection over Union) 등에서 우위 |
“U-net은 매우 적은 데이터만으로도 다양한 생의학 영상에서 정밀한 분할(segmentation)을 수행하며, 기존 최고 방법들을 압도적으로 능가했다!
5. Conclusion
U-net 아키텍처는 매우 다양한 생의학 영상 분할(biomedical segmentation) 응용 분야에서 아주 뛰어난 성능을 보였다.
elastic deformation을 활용한 데이터 증강 덕분에 주석된 이미지가 아주 적어도 충분하며 학습 시간도 Nvidia Titan GPU (6GB)에서 단 10시간으로 매우 합리적인 수준이다.
우리 caffe 기반의 전체 구현과 학습된 네트워크를 함께 제공한다.
U-net 아키텍처가 다른 많은 분야의 작업에도 손쉽게 적용될 수 있다.