[Paper Review] Divide-and-Rule: Self-Supervised Learning for Survival Analysis in Colorectal Cancer
Paper Review

Divide-and-Rule: Self-Supervised Learning for Survival Analysis in Colorectal Cancer
Introduction
Colorectal cancer (CRC) 환자 slide를 self-supervised representation learning 방식으로 feature space에 embedding 하는 과정에서, 독특한 loss를 설계하여 clustering 하였고, 환자의 정보를 patch cluster 간의 interaction과 clustering 정보로 표현하여 예후예측에 활용함.
Method
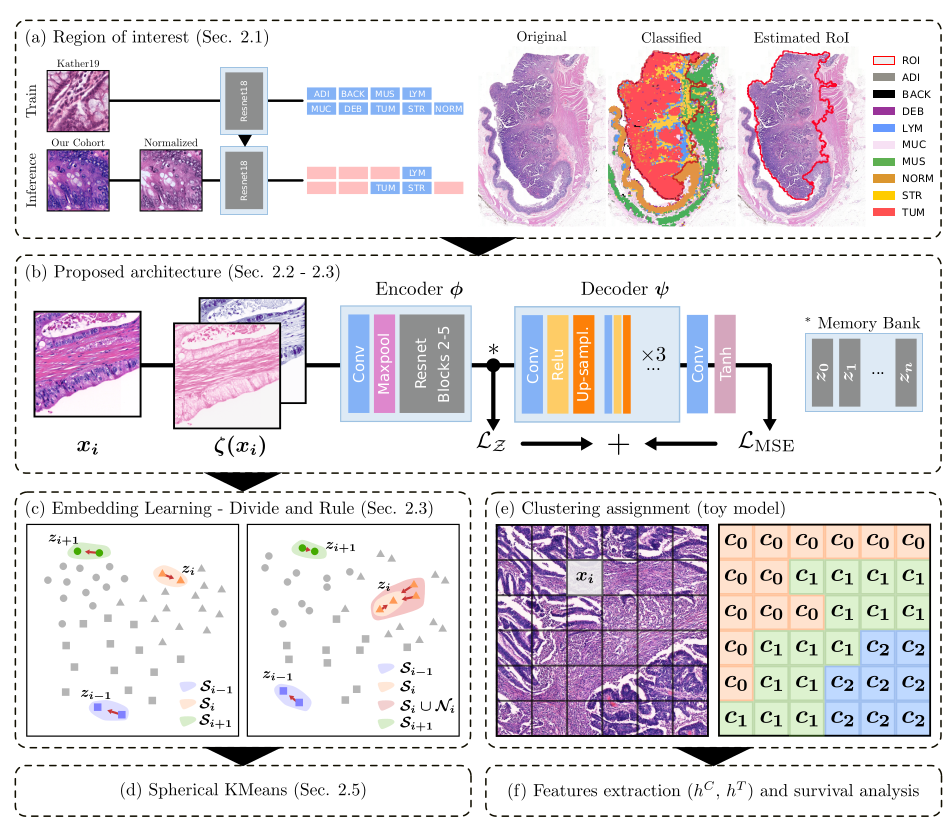
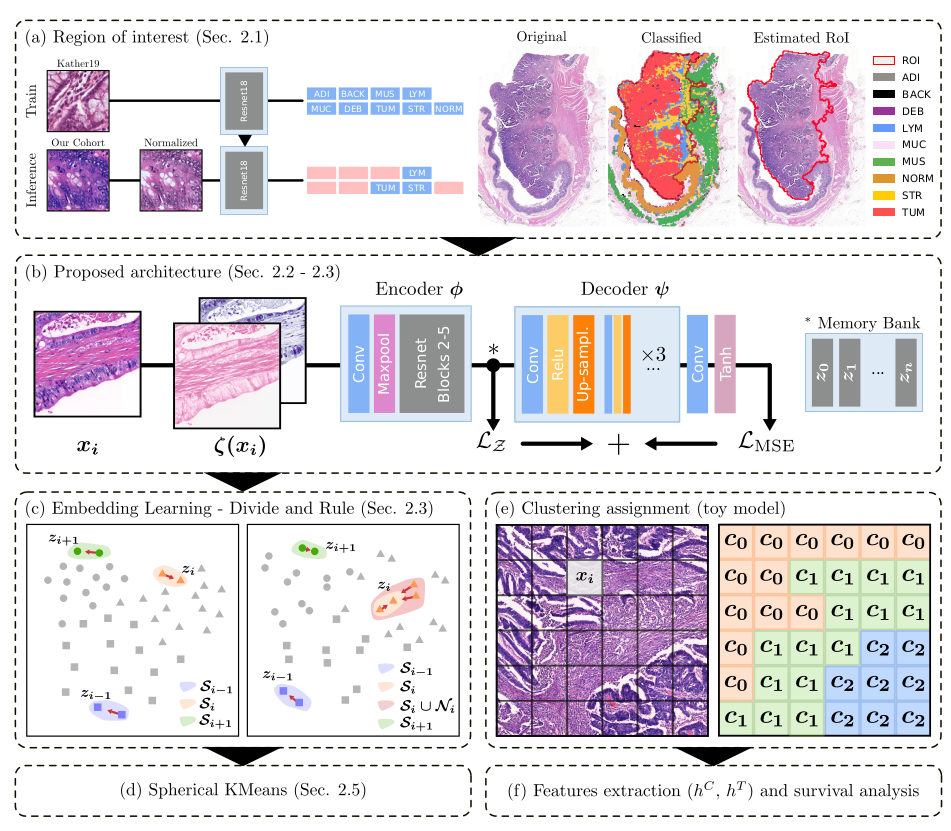
본 논문의 pipeline의 간략한 소개이다. 크게 세 단계로 구성되어 있다.
ROI detection
다른 데이터셋에서 이미 훈련된, tissue 종류를 내놓는 multi-class classification 모델을 데려와 원하는 tissue 종류만을 잡아낸다. 이 논문에서 사용하는 데이터셋과 다른 데이터셋을 맞추기 위해 color normalization을 거친다. 원하는 tissue 종류에서 patch를 뽑아낸다.
Feature extraction & self-supervised learning
H&E staining image를 H channel, E channel 두 채널로 보내고, feature space 상에 embedding 시킨다. 이 때, self-supervised learning 방식으로 encoding을 거치는데, image 상의 실제 위치, feature space 상의 위치 등을 고려한 clustering이 이루어지도록 설계된 loss를 사용한다.
Survival model
환자의 정보를 표상하기 위해, 각 patch가 특정 cluster에 속할 확률과 특정 cluster에서 특정 cluster로 바뀔 확률인 transition probability로 환자의 정보를 표현한다. (총 K+K^2개의 column) survival model에 넣기 전에 negative log likelihood loss을 통해 각 feature의 significance를 계산하여 forward selection 을 거쳐 column을 골라 최종적으로 모델을 구성하도록 한다.
ROI detection

Kather2018에서 소개된, 100000장의 CRC image로 훈련된 multi-class classification 모델을 통해 데이터를 학습한다. 여러 종류의 tissue 중 본 논문에서는 lymphocytes (LYM), cancer-associated stroma (STR), colorectal adenocarcinoma epithelium (TUM) 을 class-of-interest 삼아 골라내었다. 또한 stain normalization에 거쳐 target domain의 color space와 match 되도록 설계하였다.
Self-supervised representation learning
Colorization learning
Autoencoder는 image의 structure보다 color를 보는 경향이 있음.
이를 피하고자 colorization learning을 먼저 진행하고 encoding을 하도록 설계함.
Image를 H, E channels로 embedding함.
Convolutional autoencoder (CAE) and MSE
이후 CAE를 이용해 latent space 로 보내고 decoder로 upsampling 과정을 거쳐 input image를 재생성한 후 per-pixel difference를 계산하고 loss 항에 포함시킴.
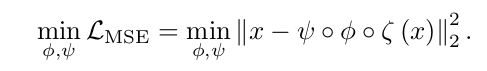
Proposed Divide-and-Rule approach
어렵다.
본 논문의 representation learning의 기본 theme 자체는, 실제 image의 spatial proximity를 feature space 상에도 표현하자 이다. 이를 위해, 실제 image 상에서 가까운 patch는 positive pair로 간주하고, 이를 제외한 batch 내의 patch는 negative pair로 간주하여 distant하게 표상되도록 하였다.
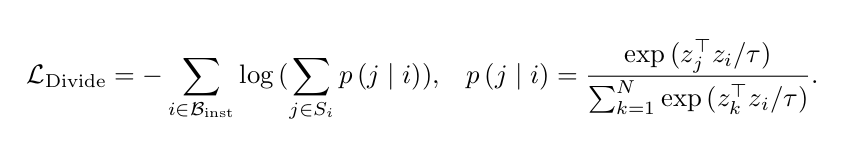
구체적으로, variant of the cross-entropy 인 nonparametric softmax classifier를 이용하여 instance loss를 계산한다. (원래 cross entropy는 transpose되는 vector가 w로 따로 존재하는데, 이렇게 설계되면 class phenotype으로 작동하여 instance들 사이의 explicit comparison를 방해한다.)
이를 통해, 내부에 있는 instance에 대해서는 positive pair로, 그 외의 안에 있는 instance에 대해서는 negative pair로 작용한다. log 안에 있는 집합은 실제 image에서 특정 patch와 닿고 있는, overlap 되고 있는 patch들의 집합이라고 생각하면 된다.
이후 끄트머리 epoch에서 하나의 loss가 더 추가된다.
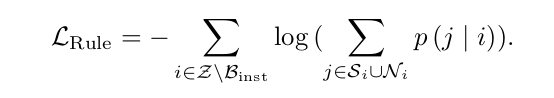
이전 divide loss에서는 멀찍이 떨어뜨리는 데에 초점을 두었다면, 이번 rule loss는 feature space 상의 different patch들 간의 similarity를 표현하고자 하였다. 구체적으로, (neighbor)는 실제 image가 아닌 feature space 상의 similarity가 가장 높은 top K개의 patches의 집합이다. 이들을 positive pair로 간주함으로써 그들간의 거리는 가깝게 표상되도록 설계하였다.
또한 저자는 점진적으로 neighbor set이라 여겨지는 vicinity의 범위를 늘려가며 학습하였다. 상대적으로 high entropy를 가지는 sample들은 서로 다른 class로 표현되어야 하므로 에 놓고, 상대적으로 low entropy를 가지는 sample들은 적은 수의 class로 표현되어야 하므로 set에 놓았다. 이 entropy의 기준을 점점 높여감으로써 처음에는 조금만 달라도 즉 조금만 entropy가 높아도 서로 멀게 encoding했다가 후반 epoch에서는 어느정도 entropy가 높아도 가까이에 encoding 되도록 하였다.
마지막으로 weighting term와 함께 소개되었던 각 training loss 들이 join 되도록 하였다.

Dictionary Learning - similarity를 계산할 때 dataset 내에 모든 sample과 계산하면 cost가 너무 크기에, queue 형태의 memory bank 를 사용하여 complexity를 줄이고자 하였다.
Survival Analysis
Clustering Assignment
위 과정은 모두 어떻게 각 patch들을 feature space상에 표상할지를 학습하는 과정이었다. 위 모델과 함께, spherical K-Means clustering 알고리즘을 이용해 환자(slide)마다 각 patch들이 feature space 상에 K 개의 clusters로 patch들이 표상되도록 하였다.

또한 tumor-related image region 들 간의 interaction을 표현하기 위해, patient descriptor로써 두 가지 probability를 정의한다.
- Probability that a patch belongs to cluster k
- Probability transition bewtween a patch and its neighbors N(s)
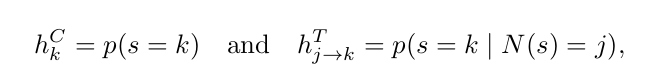
하지만, 이는 각 patch에 대해 존재하는 probability 정보인데 이를 어떻게 patient-level 정보로 사용하는지는 나와있지 않음.
Survival
데이터도 적고 환자마다 총 개의 feature가 존재하기에, overfitting 문제를 해결하고자 forward variable selection using log partial likelihood function 방식을 사용하였다.
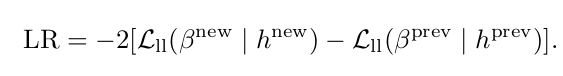
Training 과정에서, 먼저 아무 feature도 포함시키지 않은 것과 하나의 feature 만 포함시킨 것의 log partial likelihood function loss 차이를 구하고, 이 차이가 가장 significant한 feature 부터 포함시킨다. 보통 chi distribution에서 p-value를 계산한다고 한다.

마지막으로 개별 환자 데이터에 overfit 하지 않음을 validate하기 위해 LOOCV를 통해 linear estimator를 predict하고, C-index를 계산하여 최종 성능을 측정하였다. 구체적으로 beta^(-i)는 i 번째 patient를 제외한 모든 patient에서 계산되었다. (LOOCV)
Experimental Results
Dataset
660 unlabeled WSIs of CRC linked to 374 patients.
Mucinous adenocarcinoma 환자는 표준 adenocarcinoma 환자와 좀 다른 feature를 띄기에 제외.
는 slide만 존재, 는 slide와 histopathological feature 함께 존재.
Settings
ResNet-18 for the encoder, latent space dimensions d = 512.
Succession of CV layers for the decoder.
- 초반 20 epochs - only MSE loss with early stopping
- 이후 20 epochs - MSE loss + divide loss
- 마지막 3 epochs - MSE loss + divide loss + rule loss
Clustering Embedding Space : K = 8, K = 16
Ablataion Study and Survival Analysis Results

DCS, DCA, DEC는 본 논문에서 사용한 baseline algorithm들이다.
5번째 실험인 w/o Rule loss는 MSE loss와 divide loss만 사용한 실험이다. 이 때 성능저하가 있었는데, 이는 divide loss가 data 를 scatter하여 self instance representation에 집중하였기 때문이라 해석하였다.
또한 마지막 실험에서 최고 성능을 보였고, forward selection에서 가장 많은 feature (=13)을 선택한 것을, subtle patches interaction을 더 잘 잡아내었다고 해석하였다.
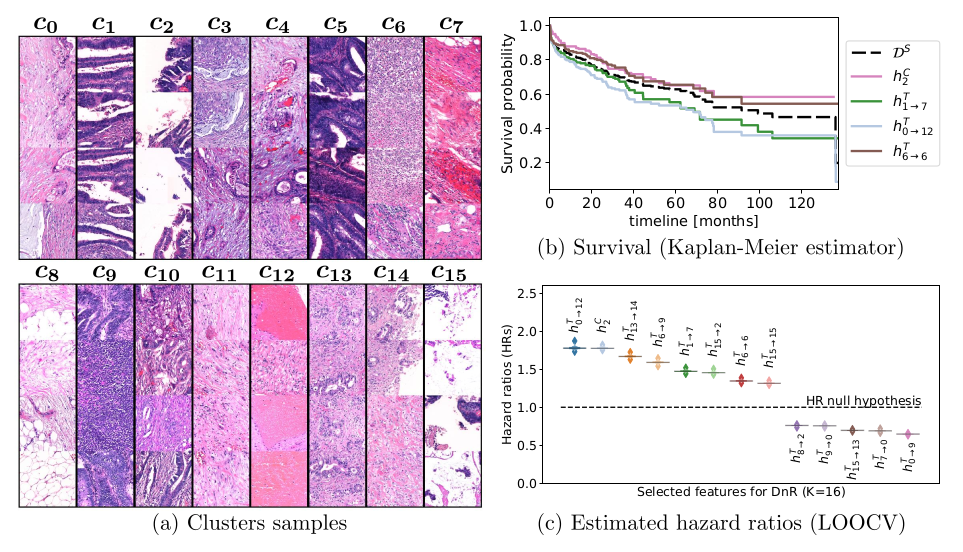
Clusters demonstrate different tumor and stroma interactions (c 0 , c 1 , c 5 , c 9 ), inflammatory tissues (c 6 ), muscles and large vessels (c 7 ), collagen and small vessels (c 8 ), blood and veins (c 11 ) or connective tissues (c 12 ). Some clusters do not directly represent the type of tissue but rather the positioning information such as c 2 , which describe the edge of the WSI.
KL estimation 결과, blood vessel과 tumor stroma 사이의 interaction은 lower survival outcome과 관련 있었고, tumor stroma와 connective tissue 사이의 interaction도 비슷한 추세를 보였다.
(해당 feature coefficient가 높은 group에 대해 KM estimation을 했을 것으로 추측.)
Conclusion
- selecting image patches: image patch를 self supervised representation learning 방식으로 feature space 상에 embedding하고, cluster probability와 cluster transition probability를 통해 patient를 나타내 linear estimator를 학습하여 성능을 측정하고 tumor related region간의 interaction을 확인함.
독특한 loss를 통해 patch들이 실제 image 상의 정보를 함유한채 feature space 상에 표상되도록 하였다. 읽는 동안 물음표가 많았고,, 구체적으로 설명해주지 않은 부분이 꽤 많았던 것 같다.
