시본 (seaborn) 통계 그래프
- 판다스(그래프) + 맷플랍립(시각화) = 시본(통계 그래프)
- 시본 공식 사이트 https://seaborn.pydata.org/
- 시본 개발 사이트 : https://github.com/mwaskom/seaborn
- 시본 개발 사이트 샘플링 : https://github.com/mwaskom/seaborn-data
작업은 Google의 colab에서 하였다.
필요한 모듈을 import 해주자.
import matplotlib as mpl
import matplotlib.pyplot as plt # 서브 패키지 모듈
import numpy as np
import seaborn as sns
#데이터 파일의 경로를 지정한다.
from pathlib import Path
data_path = Path('경로)붓꽃 데이터(iris)를 가져와 보자
iris = sns.load_dataset('iris')
iris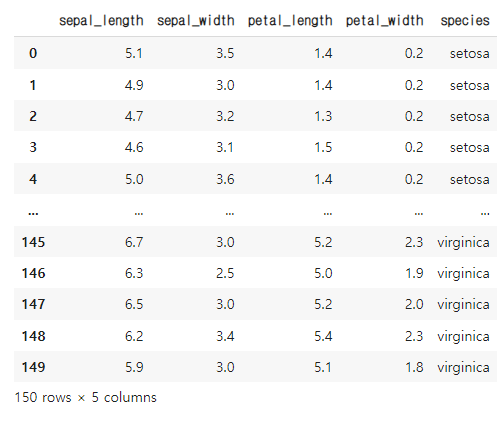
이변량 분포의 산포도를 그려보자. joinplot()
# 데이터셋에 있는 열 이름들을 이용해서 x축,y축 데이터를 지정한다.
sns_plot = sns.jointplot(x = 'sepal_length', y= 'sepal_width',data=iris, kind ='scatter')
sns_plot.set_axis_labels(xlabel = '꽃받침_길이',ylabel = 'sepal_length', size =14)
plt.suptitle('scatter plot',y = 1.04, size =24) # y값으로 제목을 띄워보자.
sns_plot.savefig('/content/gdrive/My Drive/test/시본_산포도.png',dpi=600)
plt.show()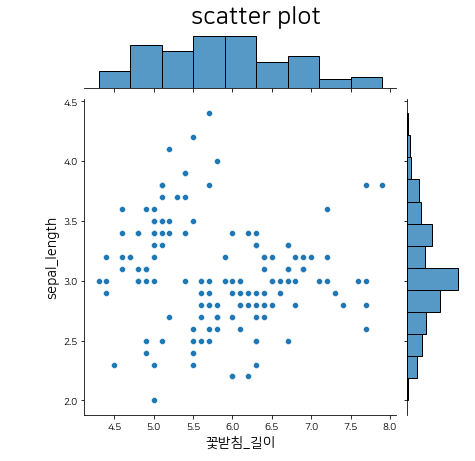
jointplot(x값,y값,data,그래프 형식(종류))
set_axis_labels(xlabel,ylabel,size)로 x,y의 이름 사이즈를 설정
plt(matplotlib.pyplot)의suptitle()메소드로 제목을 지정할 수 있다.
savefig()로 저장 dpi = 해상도관련
핵밀도 추정 분포도 (kernel density estimation)
# kind = kde
# cmap = sns.cubehelix_palatte()
# 데이터셋에 있는 열 이름들을 이용해서 x축,y축 데이터를 지정한다.
sns_plot = sns.jointplot(x = 'sepal_length', y= 'sepal_width',data=iris,
kind ='kde',
camp = 'plasma') # 핵밀도 추정 분포도
sns_plot.set_axis_labels(xlabel = '꽃받침_너비',ylabel = '꽃받침_길이', size =14)
plt.suptitle('붓꽃의 꽃받침 길이/넓이 산포도',y = 1.04, size =24) # y값으로 제목을 띄워보자.
sns_plot.savefig('시본_산포도03.png',dpi=600)
plt.show()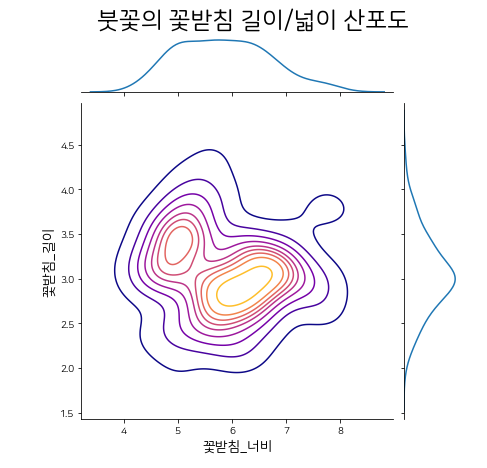
camp= 'plasma' 색상
다변량 분포의 산포도 그리기
# 꽃받침 길이, 넓이, 꽃잎의 길이, 넓이
sns_plot = sns.pairplot(iris,hue= 'species')
plt.suptitle('붓꽃의 다변량 비교 분포',y=1.04, size=24)
# sns_plot.savefig(data_path/'시본_다변량04.png', dpi=600)
plt.show()
#해당 데이터 간의 열 (column)에 담긴 데이터들의 분포를 알 수 있고 , 각 열간의 상관 관계를 볼 수 있다.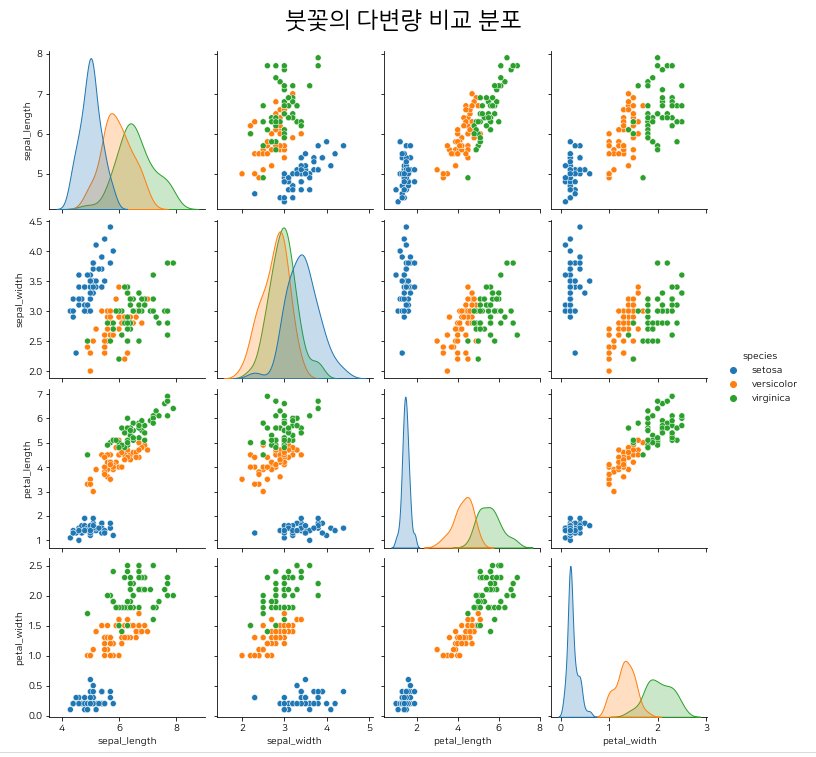
가시화
plot= 얼개(사물의 구조) = 그림
plotting= 얼개 짜기 = 그리기 = 그림의 얼개를 미리 짜둔다 = 눈에 보이지 않지만 밑그림을 다 그려 둔다.
데이터 시각화 (Data visualization , 데이터 가시화) : 데이터를 직관적으로 알아 볼수 있게 그림이나 표로 나타내는 일
선 차트를 사용하자. (값 배열이 하나인 경우)
plt.plot([1,3,2,7,4]) # y값
plt.show() # x = 위치 값 0,1,2,3,4 (y값 5개 일 경우)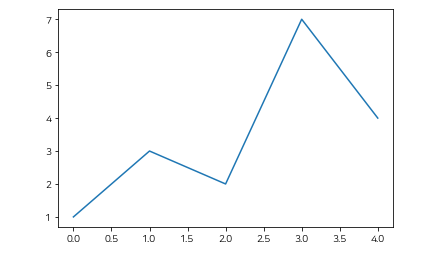
위와 같은 예
plt.plot([1,2,3,4,5,6,7,8,9,10])
plt.show() # x = 0,1,2,3,4,5,6,7,8,9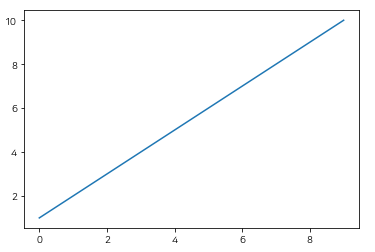
축(axis) : x축과 y축을 이야기 한다.
값 배열이 하나인 경우
x축 : 리스트 원소 개수에 자동으로 맞춰진다.
y축 : 리스트 원소 값 중 최댓값에 자동으로 맞춰진다.
선 차트를 사용하자. (값 배열이 두개인 경우)
plt.plot([1,3,2,7,4,8,11,7] , [2,4,6,8,10,12,14,16]) # ( [x값] [y값] )
plt.show()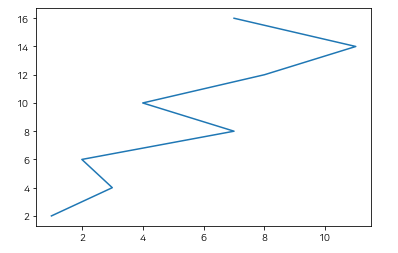
plt.plot([0,1,2,3,4,5,6,7,8,9] , [0,6,2,7,3,8,4,9,5,10]) # ( [x값] [y값] )
plt.show()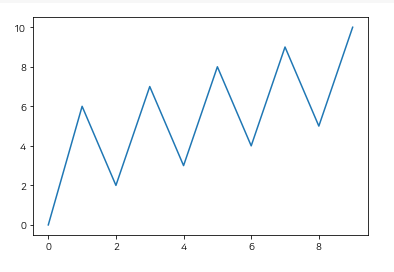
값 배열이 두 개인 경우
x축 : 첫번째 배열의 원소가 x값
y축 : 두번째 배열의 원소가 y값
그래프 풍부화 : 탄젠트를 사용한 그래프를 이용해서 편향치에 대한 판별을 유도한다.
x = np.linspace(-2*np.pi, 2*np.pi, 20)
#스타일 지정
plt.plot(x, np.tanh(x), color='red',linestyle=':',marker = 's')
plt.plot(x, np.tanh(x -2), color='blue',linestyle='--',marker = 'x')
# 그래프 제목 추가
plt.title('쌍곡 탄젠트그래프', fontsize = 20)
#축 제목 추가
plt.xlabel('파이')
plt.ylabel('쌍곡 탄젠트')
plt.grid() # 격자
#범례 표시
plt.legend(['편향치 없다.'],['편향치 있다.'], loc=4) #loc로 범례 위치 지정
plt.show()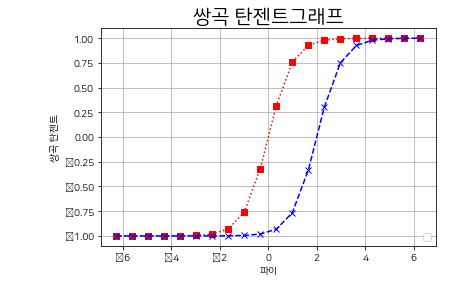
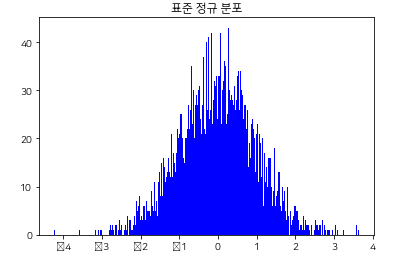
히스토그램 연속 막대 그래프
# ex) 표준 정규 분포(기대값 = 0, 표준편차 = 1)
x = np.random.randn(10000)
plt.hist(x,bins= 1000, color='blue')
plt.title('표준 정규 분포') #bins = 1000으로 세분화
plt.show()
막대 차트 : 불연속 적인 데이터를 그려내는 막대형 차트(신호 차트)
x = ['신호1','신호2','신호3','신호4','신호5']
y = [0.1100, 0.1213, 0.0612, 0.0901,0.1131]
# 세로 막대 그래프 width 값으로 폭을 조정
# plt.bar(x,y,width= 0.8)
# 가로 막대 그래프
plt.barh(x,y)
plt.title('신호기 오차 비교',fontsize=16)
plt.show()
#가로 일때는 x와 y가 뒤바뀐다.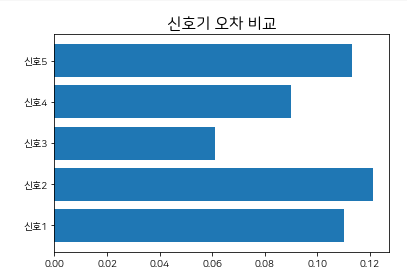
from pathlib import Path
data_path = Path('/content/gdrive/My Drive/test')
#파일 경로
# file_path = Path('경로')
x =['신호1','신호2','신호3','신호4','신호5' ]
y = [0.1100, 0.1213, 0.0612, 0.0901, 0.1131]
int_y = np.multiply(y, 10000)
plt.pie(int_y, labels=x, autopct='%.1f%%', explode = (0, 0.0, 0.2, 0, 0))
plt.title("신호오차비율" , fontsize=20)
# plt.savefig("경로.png", dpi =600)
plt.show()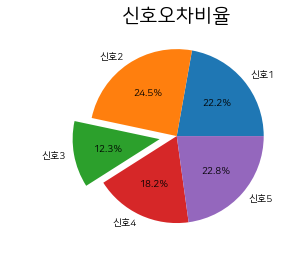
흑백변환 pyplot.imshow()를 사용해 흑백 변환작업
#이미지를 로드할 때 3차원 ndarray로 변환되어 리턴한다.
img_res = plt.imread('경로 ') #이미지 읽기
# plt.imshow(img_res)
#axis로 2개의 축을 지정한다. 지정된 축에 맞게 평균을 구한다.
plt.imshow(img_res.mean(axis=2))
#축 숨기기
plt.xticks([]) #리스트를 비워둔다.
plt.yticks([])
#plt.show()
plt.savefig("경로", dpi =600)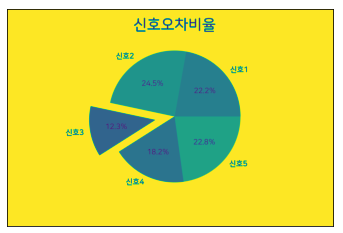
흑백변환 openCV를 사용해 흑백 변환작업
import cv2
cv2_res = cv2.imread('경로',cv2.IMREAD_GRAYSCALE)
plt.imshow(cv2_res)
#축 숨기기
plt.xticks([]) #리스트를 비워둔다.
plt.yticks([])
plt.show()
