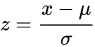총 49개마리의 생선의 길이와 무게에 대한 데이터가 있다.
49마리의 생선 중 35마리는 도미(1), 14마리는 빙어(0)이다.
해당 데이터는 Kaggle에 공개된 데이터셋이다. Kaggle의 생선 데이터셋
fish_length = [25.4, 26.3, 26.5, 29.0, 29.0, 29.7, 29.7, 30.0, 30.0, 30.7, 31.0, 31.0,
31.5, 32.0, 32.0, 32.0, 33.0, 33.0, 33.5, 33.5, 34.0, 34.0, 34.5, 35.0,
35.0, 35.0, 35.0, 36.0, 36.0, 37.0, 38.5, 38.5, 39.5, 41.0, 41.0, 9.8,
10.5, 10.6, 11.0, 11.2, 11.3, 11.8, 11.8, 12.0, 12.2, 12.4, 13.0, 14.3, 15.0]
fish_weight = [242.0, 290.0, 340.0, 363.0, 430.0, 450.0, 500.0, 390.0, 450.0, 500.0, 475.0, 500.0,
500.0, 340.0, 600.0, 600.0, 700.0, 700.0, 610.0, 650.0, 575.0, 685.0, 620.0, 680.0,
700.0, 725.0, 720.0, 714.0, 850.0, 1000.0, 920.0, 955.0, 925.0, 975.0, 950.0, 6.7,
7.5, 7.0, 9.7, 9.8, 8.7, 10.0, 9.9, 9.8, 12.2, 13.4, 12.2, 19.7, 19.9]사이킷런의 KNeighborsClassifier의 을 사용하기 위하여 길이와 무게에 대한 데이터를 하나의 2차원 리스트로 연결해준다.
fish_data = np.column_stack((fish_length, fish_weight))np.column_stack이란?
- 이 함수는 행 벡터가 주어지면 열(axis=1) 방향으로 합치는 함수입니다
a = np.array([1,2,3]) b = np.array([4,5,6]) np.column_stack((a,b)) # shape=(3, 2) #출력 결과 # array([[1, 4], # [2, 5], # [3, 6]])
이제 타깃 데이터에 대해 만든다.
# 1이 35개, 0이 14개가 연결된 일차원 리스트 생성
fish_target = np.concatenate((np.ones(35), np.zeros(14)))이제 이 데이터들을 학습용 데이터와 테스트용 데이터로 나눈다.
from sklearn.model_selection import train_test_split
train_X, test_X, train_y, test_y = train_test_split(fish_data, fish_target, random_state=42)
- train_test_split() 함수는 사이킷런의 model_selection이란 모듈 안에 있다.
- 이 함수는 전달되는 데이터를 비율에 맞게 학습용 데이터와 훈련용 데이터로 알맞게 나누어줌.
- 비율의 기본값은 학습용 데이터: 0.75, 테스트용 데이터: 0.25
넘파이 배열의 shape속성으로 해당 배열들의 크기를 출력해보자.
print(train_X.shape, test_X.shape)
#출력결과: (36, 2), (13, 2)print(train_y.shape, test_y.shape)
#출력결과: (36,) (13,)총 49개의 데이터를 학습용 데이터와 테스트 데이터로 각각 36개, 13개로 나눈 것을 볼 수 있다.
한 번 테스트 데이터셋을 출력해보자.
print(test_y)
#출력결과: [0. 0. 1. 0. 1. 0. 1. 1. 1. 1. 1. 1. 1.]총 13개의 테스트 데이터셋 중 도미(1)은 10개, 빙어(0)는 3개가 있다. 얼핏 보면 테스트 데이터셋이 잘 섞인 것 같지만, 비율을 계산해보면 두 생선의 비율은 3.3 : 1 이다.
하지만 원래의 데이터셋의 두 생선의 비율은 2.5:1이므로, 빙어의 비율이 조금 모자른 것을 볼 수 있다. 이러면 샘플링 편향이 나타날 수 있다.
샘플링 편향이란?
- 특정 종류의 샘플이 과도하게 많아, 샘플링이 한쪽으로 치우친 경우이다.
- 샘플링 편향을 가지고 있다면 제대로 된 지도학습 모델을 만들 수 없다.
이런 샘플링 편향을 방지하기 위하여 train_test_split 함수의 stratify 매개변수에 타깃 데이터를 전달하여 비율에 맞게 데이터를 나눈다.
train_X, test_X, train_y, test_y = train_test_split(fish_data, fish_target, random_state=42, stratify=fish_target)다시 test_y를 출력한다면 타깃 데이터의 비율에 맞게 데이터를 나눈다.
print(test_y)
# 출력결과: [0. 0. 1. 0. 1. 0. 1. 1. 1. 1. 1. 1. 1.] (2.25: 1)생선 데이터를 비율에 맞게 학습용 데이터, 테스트용 데이터로 나누었다.
이제 k-최근접 이웃 알고리즘을 이용하여 모델을 훈련시킨 후 평가한다.
from sklearn.neighbors import KNeighborsClassifier
kn = KNeighborsClassifier()
kn.fit(train_X, train_y) # 학습용 데이터를 이용하여 모델 훈련
kn.score(test_X, test_y) #테스트 데이터를 이용하여 모델 평가
#출력결과: 1.0출력결과는 1.0으로 모든 데이터를 올바르게 분류했다는 것을 볼 수 있다.
현재 25cm, 150g의 피처를 가진 '도미'가 있다.
한번 해당 도미를 새로운 생선 데이터로 만들어, 모델을 이용하여 해당 생선은 도미인지 빙어인지 분류를 해보자.
new_data = [25,150] #25cm, 150g인 도미 데이터
print(kn.predict([new_data]))
#출력결과: [0.]도미 데이터를 넣었지만 결과는 빙어(0)로 나왔다. 위에서 정확도가 1.0이 나와서 당연히 예측 결과가 맞을 줄 알았지만, 예측 결과가 틀렸다. 그 이유는 무엇일까?
이 새로운 데이터를 우리가 훈련시킨 학습용 데이터와 함께 산점도로 그려보자.
plt.scatter(train_X[:, 0], train_X[:, 1])
plt.scatter(new_data[0], new_data[1], marker='^')
plt.xlabel('length')
plt.ylabel('weight')
plt.show()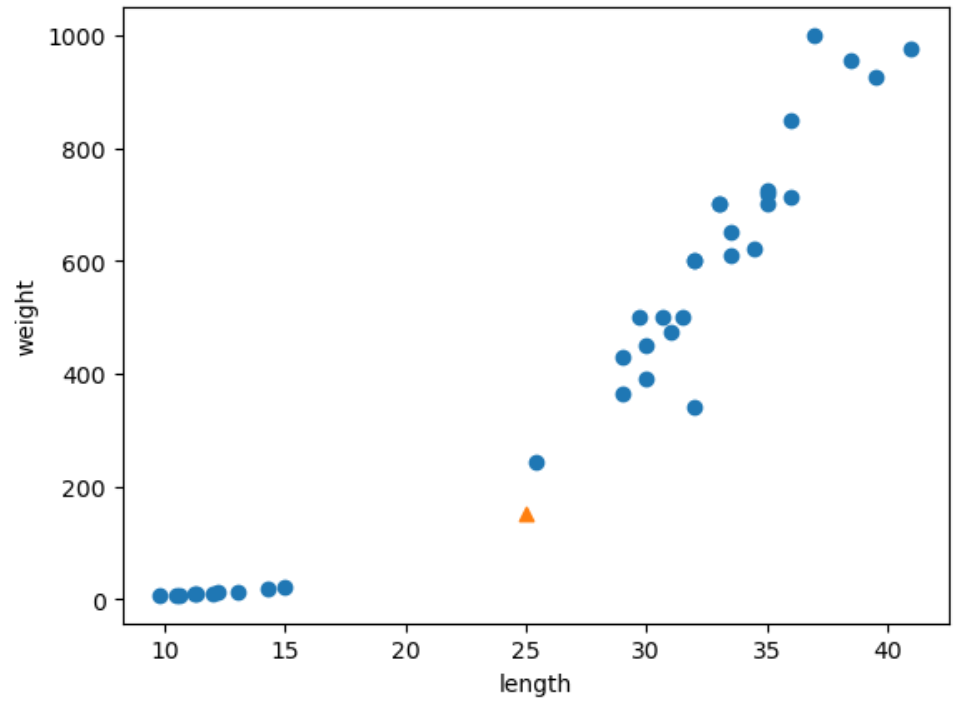
위의 삼각형 데이터가 25cm, 150g에 대한 새로운 데이터다. 여기에서 이상한 점이 있다.
직관적으로 산점도를 봤을 때 새로운 데이터는 왼쪽에 깔린 빙어 데이터보다는 오른쪽으로 뻗은 도미 데이터에 가깝다. 하지만 어째서 해당 데이터를 빙어 데이터에 가깝다고 분류한 것일까.
우선 해당 샘플에 대하여 가장 가까운 점 5개를 구해보자.
distance, indexes = kn.kneighbors([new_data])kneighbors() 메서드는 이웃까지의 거리와 이웃 샘플의 인덱스를 반환한다.
n_neighbors의 기본값은 5이므로, 5개의 이웃에 대한 거리와 인덱스를 반환한다
샘플과 근접한 5개의 점을 다시 산점도로 그려보자.
plt.scatter(train_X[:, 0], train_X[:, 1])
plt.scatter(new_data[0], new_data[1], marker='^')
plt.scatter(train_X[indexes, 0], train_X[indexes, 1], marker='D')
plt.xlabel('length')
plt.ylabel('weight')
plt.show()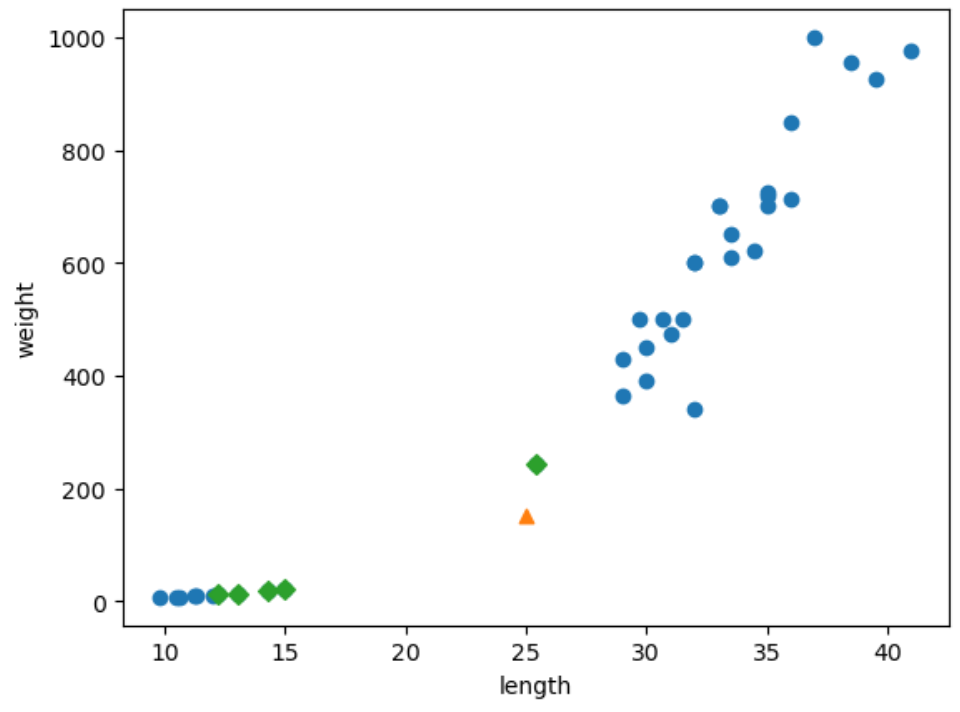
kneighbors() 메서드의 반환값 중 하나인 인덱스를 이용하여 타깃 데이터를 확인하면 더 명확하게 볼 수 있다.
print(train_y[indexes])
#출력결과: [[1. 0. 0. 0. 0.]]가장 가까운 이웃 중 4개는 빙어(0), 1개는 도미(1)이다. 그렇기 때문에 학습 모델은 해당 데이터를 빙어라고 판단한 것이다.
산점도로 보면 직관적으로 도미와 가깝게 보이는데 왜 빙어로 판단을 할까?
그 이유는 가장 가까운 이웃은 거리로 판단하기 때문이다.
산점도를 보면 x축의 범위는 10~40, y축의 범위는 0~1000 이다. 따라서 y축을 기준으로 조금만 멀어져도 거리가 아주 큰 값으로 계산이 된다. 즉, 샘플 기준으로 y축으로 멀리 떨어진 도미가 아닌, x축 기준으로 멀리 떨어진 빙어의 거리가 더 짧게 계산된 것이다.
이 상황을 두 특성의 스케일(scale)이 다르다고 말한다. 특성 간 스케일이 다른 경우는 굉장히 흔하다. 어떤 사람이 방의 넓이를 재는데 가로는 cm, 세로는 inch로 쟀다면, 두 수치가 같아도 직사각형으로 보일 것이다.
k-최근점 이웃 알고리즘을 포함한, 다른 거리 기반 알고리즘일 경우에 샘플 간의 거리에 영향을 많이 받으므로, 특성값을 일정한 기준으로 맞춰 주어야 한다.
이러한 작업을 데이터 전처리라 한다.
데이터 전처리 중 하나는 표준점수(standard score)이다. (혹은 z-score이라고 부름)
표준점수는 각 특성값이 평균에서 얼마나 떨어져 있는지를 나타낸 것이다. 이를 통해 실제 특성값의 크기와 상관없이 동일한 조건으로 비교할 수 있다.
x가 원수치, σ는 표준편차, μ는 평균이라고 합니다.
위 공식을 이용해서 z-score 즉 표준점수를 구할 수 있습니다.
이제 학습용 데이터의 표준편차, 평균을 이용하여 표준편차를 구해보자.
mean = np.mean(train_X, axis=0)
std = np.std(train_X, axis=0)
train_scaled = (train_X - mean) / std
print(mean) #[ 27.29722222 454.09722222]
print(std) #[ 9.98244253 323.29893931]이제 새로운 데이터도 표준화 후, 직관적으로 확인하기 위하여 산점도로 그려보자.
new_data = (new_data - mean) / std
plt.scatter(train_scaled[:, 0], train_scaled[:, 1])
plt.scatter(new_data[0], new_data[1], marker='^')
plt.xlabel('length')
plt.ylabel('weight')
plt.show()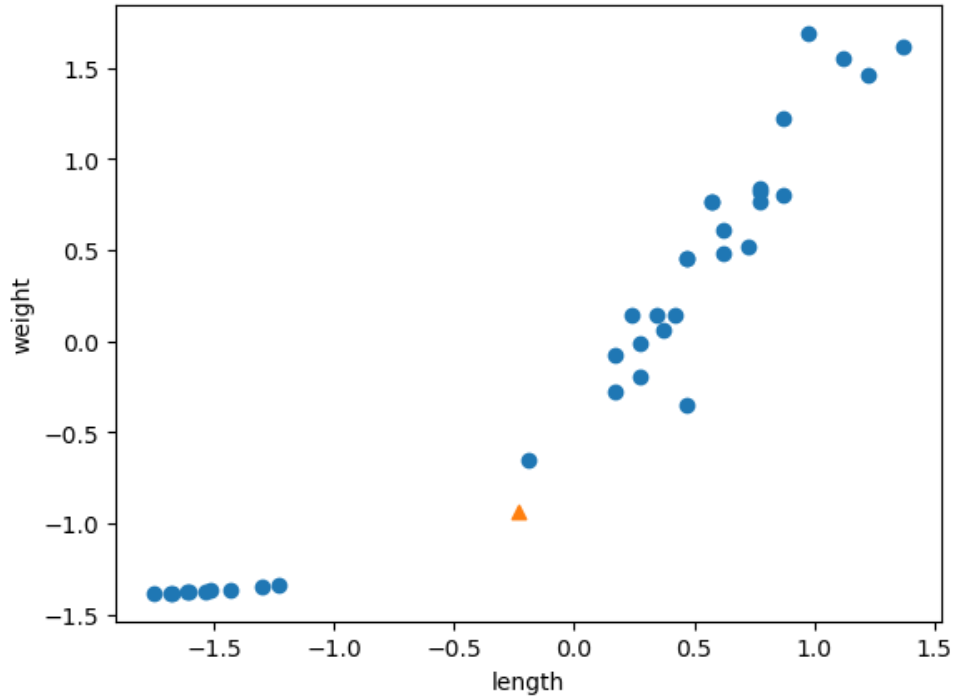
위에서의 산점도와 다른 점은 x축과 y축의 범위가 -1.5~1.5 사이로 바뀌었다. 학습용 데이터의 두 특성이 비슷한 범위로 설정됐다.
이제 표준화가 된 해당 데이터셋으로 k-최근접 이웃 모델을 다시 훈련해보자.
kn.fit(train_scaled, train_y)
kn.score(test_scaled, test_y) #결과: 1.0정확성(Accuracy)는 표준화 전과 마찬가지로 1.0이 나왔다. 이제 문제의 새로운 데이터 25cm, 150g인 도미에 대한 데이터를 넣어보자.
print(kn.predict([new_data]))
#출력결과: [1.]드디어 도미(1)로 판단했다. 마지막으로 아까 했던 kneighbors() 메서드를 이용하여 해당 데이터와 가장 근접한 데이터를 산점도로 그려보자.
plt.scatter(train_scaled[:,0], train_scaled[:,1])
plt.scatter(new_data[0], new_data[1], marker='^')
plt.scatter(train_scaled[indexes,0], train_scaled[indexes,1], marker='D')
plt.xlabel('length')
plt.ylabel('weight')
plt.show()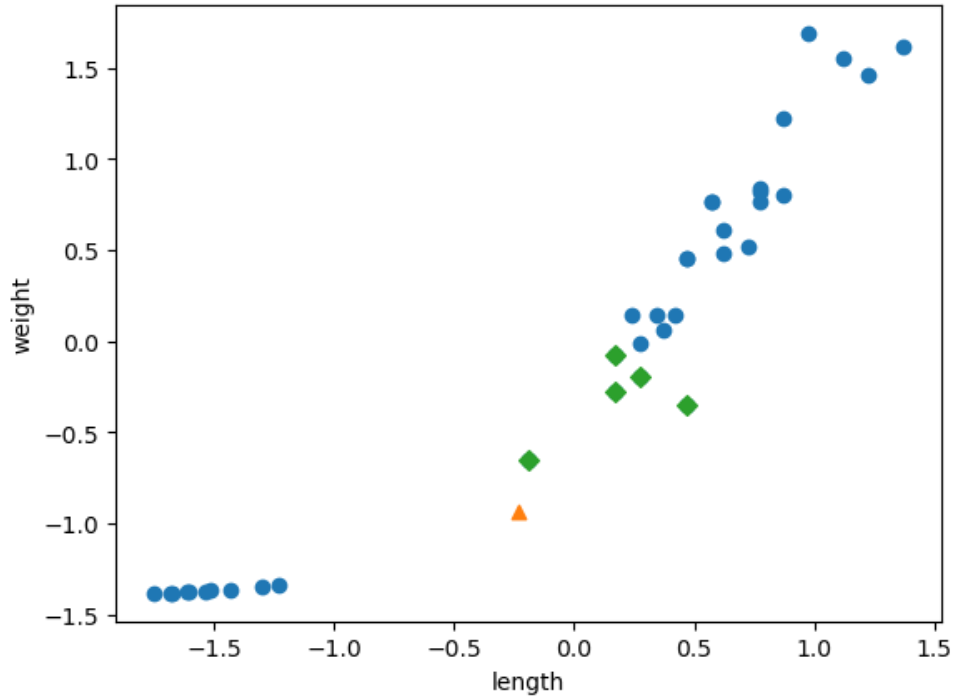
표준화 전과는 다르게 해당 데이터와 가장 가까운 샘플은 모두 도미인 것을 확인할 수 있다.