우리 학과에서는 학과는 커리큘럼 상 주언어로 Java, Python, C를 사용한다.
다른 언어들(C++, Ocaml, Java script,...)들을 배우긴 하지만 한 학기정도만 사용하게 되고 프로젝트는 돌고 돌아 친숙한 Java나 Python을 사용하게 된다.
그럼에도 불구하고 일명 HOT한 언어들은 계속해서 나오고 있는데,
Type Script라든가 Go, Kotlin 등등 여러 언어가 있지만 그 중 하나가 Rust였다.
Rust는 안전성, 속도, 병렬 처리를 중심으로 설계된 언어로, 다음과 같은 특징들이 있다.
- 메모리 안전성: 컴파일 시점에서 메모리 안전성을 검사해서 메모리 누수 및 메모리 관련 오류를 방지한다.
- 소유권 시스템: 각 변수의 데이터에 대한 소유권을 관리하여 데이터 경합과 같은 문제를 효율적으로 관리한다.
- 병렬 및 동시성 처리: 데이터 경합이 없으므로 안전하게 병렬 처리를 수행할 수 있다.
- 성능: 저수준 언어의 성능을 유지하면서도 고수준 언어의 추상화를 제공한다.
작년에 졸업 프로젝트를 진행하면서 지도교수님이 상담을 해주셨는데 그 중 C/C++언어를 주 언어로 사용하는 팀원에게 교수님이 "자네는 Rust 공부해 볼 생각 없나?"라고 말씀하셨다.
이렇게 Rust를 들어보긴 했지만 실제로 사용해 볼 일도 별로 없고
같은 프로그램이더라도 Language에 따라 속도가 확연히 차이가 날 수 있다고는 하지만 실감해 본 적이 없었는데 이번에 인턴을 진행하면서 뜻밖의 부분에서 Rust를 마주치게 되었다.
내가 현재 인턴을 진행하는 회사는 Proteomics 관련 회사로,
단백질 분석을 위해 다양한 프로그램들을 사용한다.
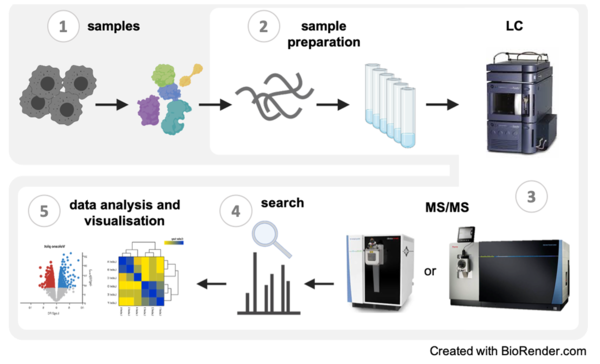 출처: https://www.leibniz-fli.de/research/core-facilities-services/cf-proteomics
출처: https://www.leibniz-fli.de/research/core-facilities-services/cf-proteomics
단백질 분석 과정(위의 사진에서 4번 과정)에서는
FASTA file을 읽고, 효소 소화 과정 시뮬레이션을 통해 단백질 서열을 펩타이드로 분해하고, 질량 분석을 위해 B/Y이온을 만드는 과정이 필요하다.
Proteomics Database Search Engine에서 이러한 단백질 서열 분석을 하고 특정한 단백질이나 펩타이드의 존재를 확인하기 위해 해당 기능을 포함하게 된다.
내가 맡은 업무는 아니지만 함께 인턴을 하던 친구가 Fragment Indexing에 Rust를 도입하는 업무를 맡게 되었다.
동일한 Indexing 프로그램을 하나는 C#으로, 다른 하나는 Rust로 작성하였는데
놀랍게도 C#은 소요 시간 5분, Rust는 50초가 나왔다.
물론 여러 경우를 생각해봐야겠지만 단순히 생각하면 언어를 바꿔서 소요시간이 1/6이 되었다.
Proteomics도 모르고 Rust를 처음 써본 그 친구가 Fragment Indexing 부분에서 속도를 6배로 높였는데,
만약에 Protemics Database Search Engine이 Rust로 작성되어 있다면?
우리의 멘토링을 담당하시던 박사님이 오픈소스 프로그램 중에서 Rust로 개발된 SAGE를 발견하셨다.

출처: https://github.com/lazear/sage
Github 페이지의 Sage 소개글은 다음과 같이 적혀있다.
Sage: proteomics searching so fast it seems like magic
정말 마법처럼 보일정도로 빠르다.
박사님은 회의 시작 전에 Search를 돌려놓고 회의 중에 결과가 다 나와버렸다며 아주 기뻐하셨다.
우리 회사는 빠르게 SAGE를 도입하기로 결정했고,
나는 SAGE를 클라우드에서, 그리고 우리의 Web Platform에서 실행하도록 Migration하는 역할을 맡았다.
내가 Proteomics에 대한 지식이 풍부했거나 이 분야에서 오랜 기간 일했다면
이 과정을 통해 배우는 것이 더 많았겠지만
내가 이런 Hot한 언어가 현업에서 사용되는 것을 보면서 느낀 것은
- 프로그램 Language를 변경함으로써 생산성을 말도 안되게 높일 수 있다
- 적절한 Language를 선택하는 것도 중요하다
- 각 언어의 장/단점을 이해하고 있는 것도 도움이 많이 되겠다.
- 필요한 기능에 대해서 open source 프로그램을 이용하는 것도 좋은 방법이다.
- 편하다고 안주하지말고 신기술, 새로운 언어를 배우는 것을 멈추지 말자.
였다.
잘 모르는 분야에서 일을 하는 것이 물론 힘이 들 때도 있지만(SAGE의 Configuration file을 이해하기 위해서 구글과 ChatGPT와 한참을 싸웠다)
새로운 것을 배운다는 것, 일을 하면서 얻어가는 것이 있다는 것은 정말 즐거운 일이다.
Rust가 Hot한 언어라고 말만 들었을 때의 나와,
실제로 그 Hot한 언어가 어떤 변화를 이끌어내는지 두 눈으로 보고 경험한 나는 분명히 다를 것이다.
