1. library
library(readr)
library(dplyr)
library(tidyr)
library(stringr)
library(tidyverse)2. 누적 데이터 기준
1) 경로 확인 및 데이터 호출
getwd()
setwd("C:/Rwork/csse_covid_19_daily_reports")
sample2022 <- read.csv('07-31-2022.csv')
sample2021 <- read.csv('07-31-2021.csv')2) 대한민국 확진자 및 사망자
- filter(): 특정 컬럼의 특정 값 추출
- select(): 필요한 컬럼만 선택
# 한국 %>% 필요한 컬럼만
korea2022 <- sample2022 %>% filter(Country_Region=='Korea, South') %>% select(Country_Region, Confirmed, Deaths)
korea2021 <- sample2021 %>% filter(Country_Region=='Korea, South') %>% select(Country_Region, Confirmed, Deaths)
# 2022 및 2021 한국 merge
korea_result <- merge(korea2022, korea2021, by='Country_Region', all = T)
# 변수 <- 2022(확진자, 사망자) - 2021(확진자, 사망자)
korea_result$total_con <- korea_result$Confirmed.x - korea_result$Confirmed.y
korea_result$total_dea <- korea_result$Deaths.x - korea_result$Deaths.y
# 최종변수 <- 지역명, 총 확진자, 총 사망자
korea_final <- korea_result %>% select(Country_Region, total_con, total_dea)
View(korea_final)3) 확인하기
| Country_Region | total_con | total_dea | |
|---|---|---|---|
| 1 | Korea, South | 19,620,952 | 22,970 |
3. 일별 데이터 기준
1) 경로 확인 및 데이터 호출
# 경로 확인 및 설정: 2108~2207 기간 내 모든 파일 ---------
getwd()
setwd("C:/Rwork/2108~2207")
# 01-01-2022 ~ 07-31-2022
sample_2022 <- list.files (pattern = '*2022.csv')
# 08-01-2021 ~ 12-31-2021
sample_2021 <- list.files (pattern = '*2021.csv')
# 08-01-2021 ~ 07-31-2022
sample_korea <- c(sample_2021, sample_2022)
# 경로 확인 및 설정: 07-31-2021 -------------------------
getwd()
setwd("C:/Rwork/csse_covid_19_daily_reports")
# 07-31-2021
sample_korea2 <- read.csv('07-31-2021.csv')2) 대한민국 확진자 및 사망자
(1) 대상 기간 시작 날짜 하루 전 날의 데이터 정제
: 일별 데이터를 추출 하고자 하는 날의 전 날 데이터를 정제
# 07-31-2021 파일에서 한국 및 필요컬럼만
dailyBasic_korea <- sample_korea2 %>% group_by(Country_Region) %>% filter(Country_Region=='Korea, South') %>% select(Country_Region, Confirmed, Deaths)(2) 반복문
: 2)-(1)의 정제과정을 365번 반복하기 위함
# 반복문에서 사용될 변수 (지역명 컬럼만 넣기)
dailyBasic_korea1 <- dailyBasic_korea[ ,-c(2,3)]
dailyBasic_korea2 <- dailyBasic_korea[ ,-c(2,3)]
# '기준일의 일일수치 == (기준일 - 전일)' 확인을 위한 반복문
for(i in 1:365){
samKo <- read.csv(sample_korea[i])
tempKo <- samKo %>% group_by(Country_Region) %>% filter(Country_Region=='Korea, South') %>% select(Country_Region, Confirmed, Deaths)
hub <- merge(tempKo, dailyBasic_korea, by="Country_Region", all=T)
hub$daily <- hub$Confirmed.x - hub$Confirmed.y
hub$daily2 <- hub$Deaths.x - hub$Deaths.y
dailyBasic_korea1 <- merge(dailyBasic_korea1, hub[c(1,6)], by='Country_Region', all=T)
dailyBasic_korea2 <- merge(dailyBasic_korea2, hub[c(1,7)], by='Country_Region', all=T)
dailyBasic_korea <- tempKo
}
# 최종변수 <- 지역명 컬럼
dailyKo_final <- dailyBasic_korea[ , -c(2, 3)]
# 변수 <- rowSums(지역명 컬럼 제외한 일일 수치) %>% data.frame 으로 만들기
a <- rowSums(dailyBasic_korea1[ , -1]) %>% as.data.frame()
b <- rowSums(dailyBasic_korea2[ , -1]) %>% as.data.frame()
# 새 컬럼 <- 지역별 총합
dailyKo_final$total_con2 <- a[ ,1]
dailyKo_final$total_dea2 <- b[ ,1](3) 누적 기준 데이터와 일별 기준 데이터 비교
# 새 컬럼 <- 1.1의 값과 1.2의 값 비교(차이)
dailyKo_final$compare_con <- dailyKo_final$total_con2 - korea_final$total_con
dailyKo_final$compare_dea <- dailyKo_final$total_dea2 - korea_final$total_dea(4) 비교 결과
- head() 사용: 상위 5개만, 정렬 기준 국가명 기준 오름차순
| Country_Region | compare_con | compare_dea | |
|---|---|---|---|
| 1 | Korea, South | 0 | 0 |
>> 두 값의 차이가 0으로 다른 방법으로 산출했지만 결과가 같은 것이 확인된다. 즉, 데이터의 이상이 없음을 알 수 있다.
4. 월별 데이터 기준
마지막에 진행될 시각화 과정에서 월별 데이터를 사용할 예정이므로 산출 되어 있는 일별 데이터를 활용하여 월별 데이터를 생성한다.
1) 생성
# 월별 변수 생성 & 일별 데이터 담기 & 행이름 열로 변경 후 이름 수정
monthly <- cbind(month, dailyFinal)
monthly <- aggregate(cbind(Korea_dailyCon, Korea_dailyDea) ~ month, data = monthly, sum) %>% .[c(8, 9, 10, 11, 12, 1, 2, 3, 4, 5, 6, 7), ]
rownames(monthly)=NULL
monthly["month"] = c("2021-08", "2021-09", "2021-10", "2021-11", "2021-12", "2022-01", "2022-02", "2022-03", "2022-04", "2022-05", "2022-06", "2022-07")
colnames(monthly) <- c("Month", "Confirmed", "Deaths")
View(monthly)2) 확인
| Month | Confirmed | Deaths | |
|---|---|---|---|
| 1 | 2021-08 | 53,658 | 194 |
| 2 | 2021-09 | 60,328 | 205 |
| 3 | 2021-10 | 52,613 | 361 |
| 4 | 2021-11 | 85,964 | 801 |
| 5 | 2021-12 | 182,903 | 1,966 |
| 6 | 2022-01 | 228,789 | 1,147 |
| 7 | 2022-02 | 2,409,707 | 1,398 |
| 8 | 2022-03 | 10,102,369 | 8,420 |
| 9 | 2022-04 | 3,899,831 | 6,285 |
| 10 | 2022-05 | 843,766 | 1,322 |
| 11 | 2022-06 | 249,442 | 358 |
| 12 | 2022-07 | 1,451,882 | 513 |
5. 기술 통계량
대상 기간(1년) 내 기술 통계량을 확인하기 위해 산출 되어 있는 일별 데이터를 활용한다. 또한, 2개 컬럼에 365개 값이 있기 때문에 날짜를 추출하여 일별 데이터와 병합한다.
1) 기술 통계 위한 데이터 프레임 생성
(1) 날짜 추출
파일명을 활용하여 날짜를 추출한다.
# 1) 날짜 추출
date <- str_replace(basename(sample_korea[1]), '.csv', '') %>% str_replace_all('-', '.') %>% as.data.frame()
month <- substr(basename(sample_korea[1]), 1, 2) %>% as.data.frame()
colnames(date) <- 'Korea, South_date'
colnames(month) <- 'month'
for(i in 2:365){
tempDate <- str_replace(basename(sample_korea[i]), '.csv', '') %>% str_replace_all('-', '.')
tempMonth <- substr(basename(sample_korea[i]), 1, 2)
date <- rbind(date, tempDate)
month <- rbind(month, tempMonth)
}(2) 일별 데이터와 병합
추출한 날짜를 일별 데이터 프레임과 병합한다.
# 2) 추출한 값 새로운 변수에 넣기
daily_conKo <- t(dailyBasic_korea1[-c(1,1)]) %>% as.data.frame()
daily_deaKo <- t(dailyBasic_korea2[-c(1,1)]) %>% as.data.frame()
# 3) 추출날짜와 병합 & 컬럼 명 변경
daily_conKo <- cbind(daily_conKo, date)
daily_deaKo <- cbind(daily_deaKo, date)
colnames(daily_conKo) <- c('Korea_dailyCon', 'Korea_dailyDate')
colnames(daily_deaKo) <- c('Korea_dailyDea', 'Korea_dailyDate')
# 4) 날짜 열 행이름으로 변경 후, 날짜 열 삭제 & 확진자테이블과 사망자테이블 병합
rownames(daily_conKo) <- daily_conKo[,2]
daily_conKo[,2] <- NULL
rownames(daily_deaKo) <- daily_deaKo[,2]
daily_deaKo[,2] <- NULL
dailyFinal <-cbind(daily_conKo, daily_deaKo)
View(dailyFinal)(3) 데이터 확인
head()를 사용해 일부 데이터만 확인한다.
| Korea_dailyCon | Korea_dailyDea | |
|---|---|---|
| 08.01.2021 | 1,215 | 1 |
| 08.02.2021 | 1,201 | 5 |
| 08.03.2021 | 1,723 | 2 |
| 08.04.2021 | 1,776 | 3 |
| 08.05.2021 | 1,704 | 4 |
>> 날짜도 정상적으로 잘 추출 된 것이 확인 된다.
2) 기술 통계량 확인
(1) library
- library(moments): 왜도(skewness), 첨도(kurtosis) 함수 사용 위함
(2) summary()
: 데이터의 평균(Mean), 중앙값(Median), 최솟값(Min.), 최대값(Max.), 쿼틸(1st Qu., 3rd Qu.)를 확인
summary(dailyFinal)▼ Console ▼
Korea_dailyCon Korea_dailyDea Min. 0 0.00 1st Qu. 2,248 10.00 Median 7,186 23.00 Mean 53,756 62.93 3rd Qu. 47,743 58.00 Max. 621,317 582.00
>> 확진자 및 사망자 모두 중앙값보다 평균이 큰 것을 보았을 때 양의 왜도 일 것으로 짐작됨
(3) sd()
- 표준편차: 주어진 데이터가 평균으로부터 얼마나 떨어져 있는지를 나타내는 지표
sd(dailyFinal$Korea_dailyCon) # =console=> 99,674.25
sd(dailyFinal$Korea_dailyDea) # =console=> 95.70366>> 확진자 및 사망자 모두 평균으로 부터 매우 큰 범위로 분포하고 있음
(4) var()
- 분산: 데이터가 얼마나 넓게 퍼져 있는지를 나타내는 지표
# 일별 확진자 분산
var(dailyFinal$Korea_dailyCon) # =console=> 9,934,955,523
# 일별 사망자 분산
var(dailyFinal$Korea_dailyDea) # =console=> 9,159.19>> 확진자 및 사망자 모두 평균으로 부터 매우 넓게 퍼져 있음 (표준 편차와 비슷한 경향)
(5) kurtosis()
- 첨도: 데이터가 평균 주위에 얼마나 집중 되어 있는지를 나타내는 지표
kurtosis(dailyFinal$Korea_dailyCon) # =console=> 9.314153
kurtosis(dailyFinal$Korea_dailyDea) # =console=> 8.856395>> 확진자 및 사망자 모두 양수로 평균 주위에 값들이 많이 집중되어 있음
(6) skewness()
- 왜도: 데이터의 분포의 대칭을 나타내는 지표
skewness(dailyFinal$Korea_dailyCon) # =console=> 2.520424
skewness(dailyFinal$Korea_dailyDea) # =console=> 2.451759>> 확진자 및 사망자 모두 양수로 오른쪽으로 치우쳐져 있는(오른쪽 꼬리가 더 긴 형태) 양의 왜도임을 알 수 있음
(7) range()
- 범위: 데이터의 최솟값과 최댓값의 차이를 나타내는 지표
range(dailyFinal$Korea_dailyCon) # =console=> 0 621317
range(dailyFinal$Korea_dailyDea) # =console=> 0 582>> 확진자 및 사망자 모두 매우 큰 범위로 분포하고 있음
(8) table()
: 데이터 내 값들의 등장한 횟수를 계산하여 테이블(표)의 형태로 반환
table(dailyFinal$Korea_dailyCon); table(dailyFinal$Korea_dailyDea)>> 확진자 및 사망자 모두 다양한 값들이 존재
(9) sum()
: 총 합
sum(dailyFinal$Korea_dailyCon) # =console=> 19,620,952
sum(dailyFinal$Korea_dailyDea) # =console=> 22,970(10) str()
: 관측수
str(dailyFinal)▼ Console ▼
'data.frame': 365 obs. of 2 variables:
$ Korea_dailyCon: int 1215 1201 1723 1776 1704 1822 1728 1492 1539 2219 ...
$ Korea_dailyDea: int 1 5 2 3 4 3 5 4 9 1 ...
3) 해석
확진자 및 사망자 데이터 모두 값의 분포가 다양하고 범위가 큰 것 등 대체로 동일한 통계량을 나타냄을 알 수 있다. 특히 '양의 왜도'의 경우 이상치가 존재할 가능성이 높지만 대상 기간이 21년 8월 부터 22년 7월임을 감안 했을 때 21년 말에 발생하여 22년 초 대유행을 일으킨 오미크론에 영향임을 미루어 짐작할 수 있다. 그렇다면 범위가 크고, 평균 주위에 분포하면서도 넓게 퍼져있는 값 등의 원인 역시 동일한 상황임을 알 수 있다.
6. 시각화
앞서 살펴본 기술통계량을 통해 알게 된 사실을 염두에 두고 해석 한다.
1) box plot
- 일별 데이터 기준 박스 플롯
- 데이터의 분포와 이상치를 시각화
- 평균(Mean), 중앙값(Median), 최솟값(Min.), 최대값(Max.), 쿼틸(1st Qu., 3rd Qu.) 확인 가능
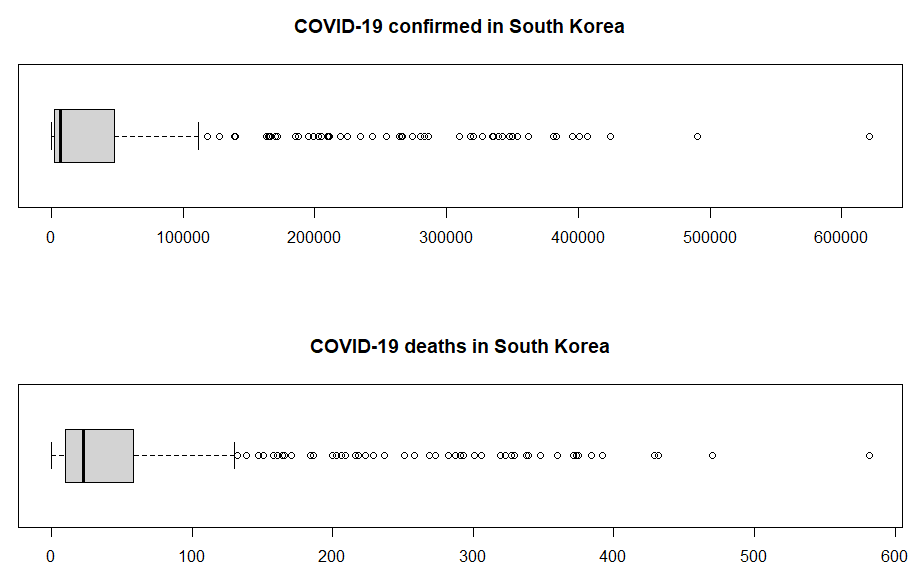
>> 기술통계량에서 봤듯이, 범위가 굉장히 크고 이상치로 보이는 것들이 많다. 하지만 이 값들을 바로 이상치로 판단해서 제외하는 것은 위험하므로 특정 상황이나 원인 등을 고려해야 한다.
2) histogram
- 월별 데이터 기준 히스토그램
- 데이터의 분포를 시각화
- X축은 데이터의 구간을, Y축은 해당 구간에 속하는 데이터의 빈도
- 데이터의 분포 모양과 중심 경향, 분산 등을 파악하는 데에 유용
- 옵션 변경 시 데이터의 밀도 추정에도 활용 가능
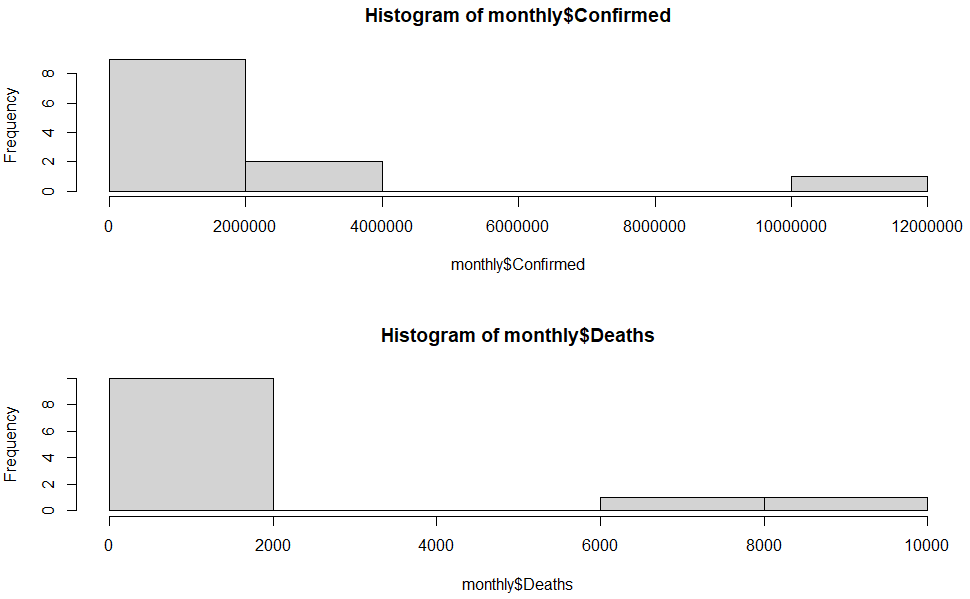
>> 기술통계량에서 봤듯이, 분포가 몰려 있는 구간이 있고 몰려 있는 구간 외 몇몇 값이 존재함을 알 수 있다. box plot의 경우와 마찬가지의 이유로 해당 그래프로 값의 발생 원인을 단순하게 추정해서는 안 된다.
3) Bar chart
- 월별 데이터 기준 막대 그래프
- 범주형 변수의 분포, 상대적 크기, 변화 등을 표현하는 데에 사용
- X축은 범주를, Y축은 해당 범주의 값(빈도, 비율 등)
(1) library
내장 함수가 아닌 ggplot을 사용하여 깔끔하고 가독성 좋은 그래프를 생성한다.
library(ggplot2)(2) 확진자
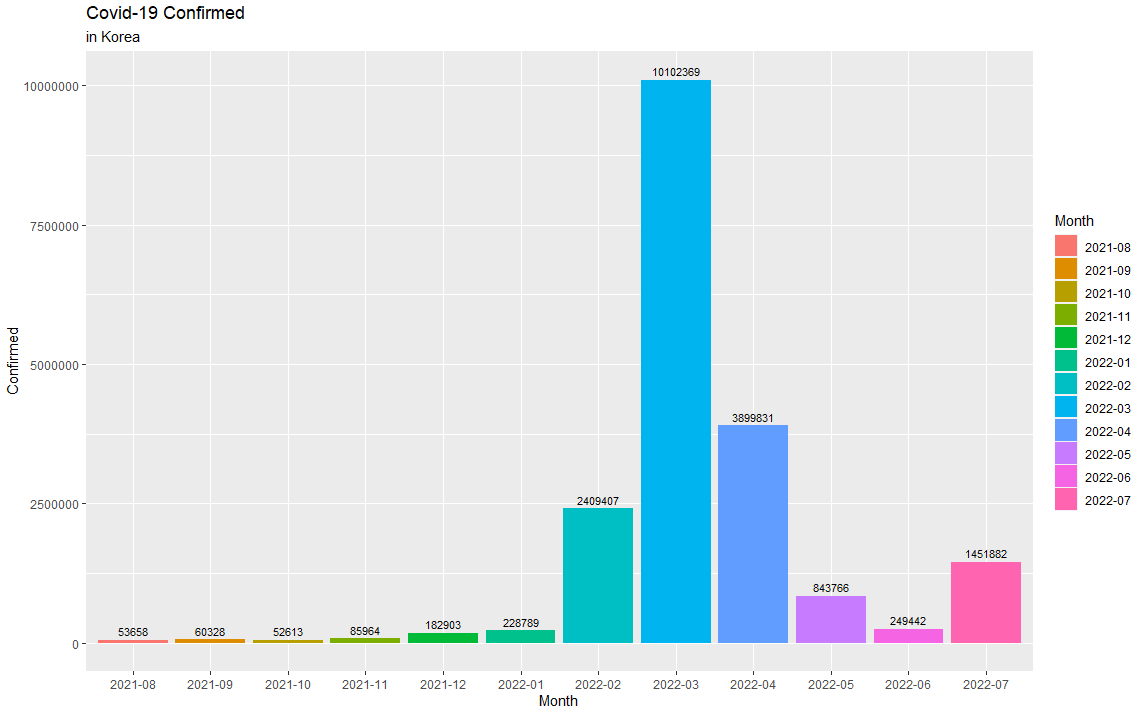
(3) 사망자
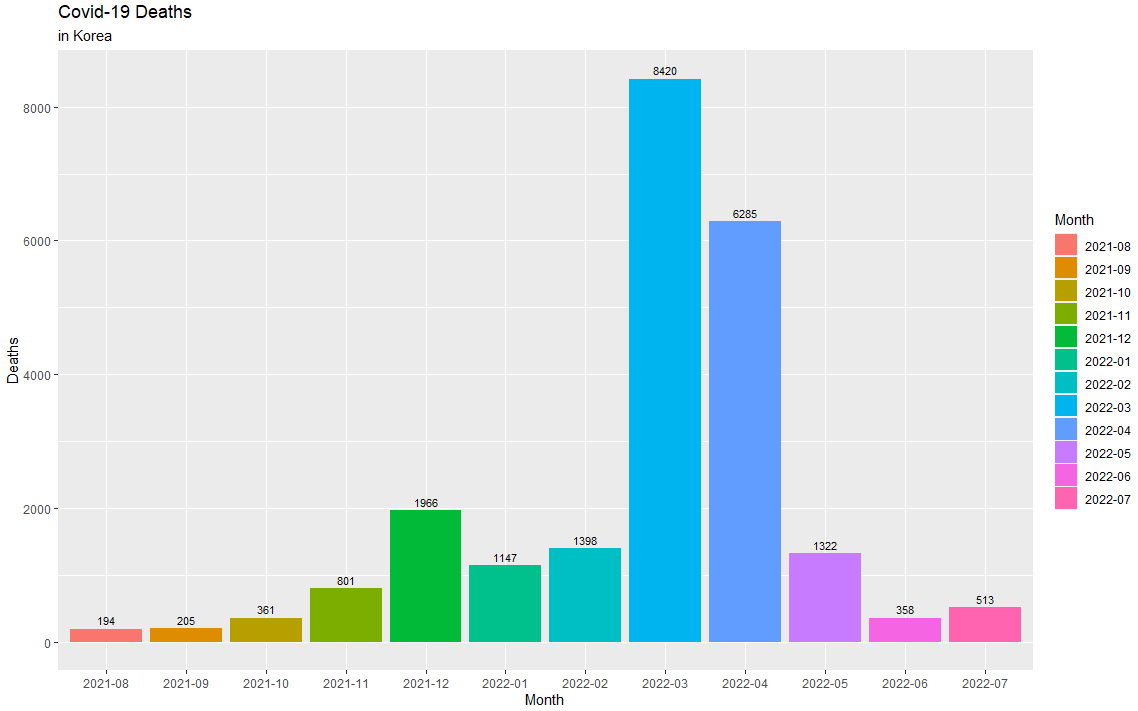
(4) 해석
막대 그래프를 통해 다른 달에 비해 특정 달의 확진자 및 사망자의 수가 확연하게 높은 것을 알 수 있다. 이는 앞에서 계속 품어왔던 의구심에 대한 답이 되므로 값에 대한 판단을 단순하게 하면 위험하다는 것을 다시 한번 깨닫게 된다.
따라서, 기술 통계량의 결과와 이에 대한 시각화를 통해 값의 흐름을 파악 및 추정하고 충분히 고민한 후 각 값의 상대적 크기를 알 수 있는 방법을 사용하며 데이터 분석을 진행해야 한다.
