개요
1. 스케일링(Scaling)이란?
1) 서로 다른 독립변수(feature)의 값 범위를 선형변환을 통하여 일정한 수준으로 맞추는 작업
2) 스케일링의 필요성
- 독립변수(feature)별로 값의 변위가 다르면 종속변수(target)에 대한
영향이 독립변수의 변위에 따라 크게 달라지고, 이는 머신러닝 시 학습효과
의 저하를 유발함 - 다차원의 값들을 동일한 수준에서 비교 분석하기 용이하게 만들어 줌
- 최적화 과정에서의 안정성 및 수렴 속도를 향상시킴
- 특히 k-means 등 거리 기반의 모델에서는 스케일링이 매우 중요함
3) 스케일링 종류(표준화 / 정규화 / 변환)
- 표준화 : 표준분포화(평균을 0, 분산을 1 로 스케일링)
- StandardScaler() : 기본 스케일러, 평균과 표준편차 사용
- RobustScaler() : 중앙값과 IQR을 사용 / 이상치의 영향을 최소화 - 정규화(규격화) : 특정 범위(주로 0~1)로 스케일링
- MinMaxScaler(): 범위가 0~1이 되도록 스케일링
- MaxAbsScaler(): 부호에 따라-1~1사이의 범위를 갖도록 스케일링 - 변환 : 특정한 분포나 모양을 따르도록 스케일링
- PowerTransformer(): 정규분포화
- QuantileTransformer() : 균일분포 또는 정규분포화
- Normalizer(): 한 행의 모든 피처들 사이의 유클리드 거리가 1이
되도록 변환
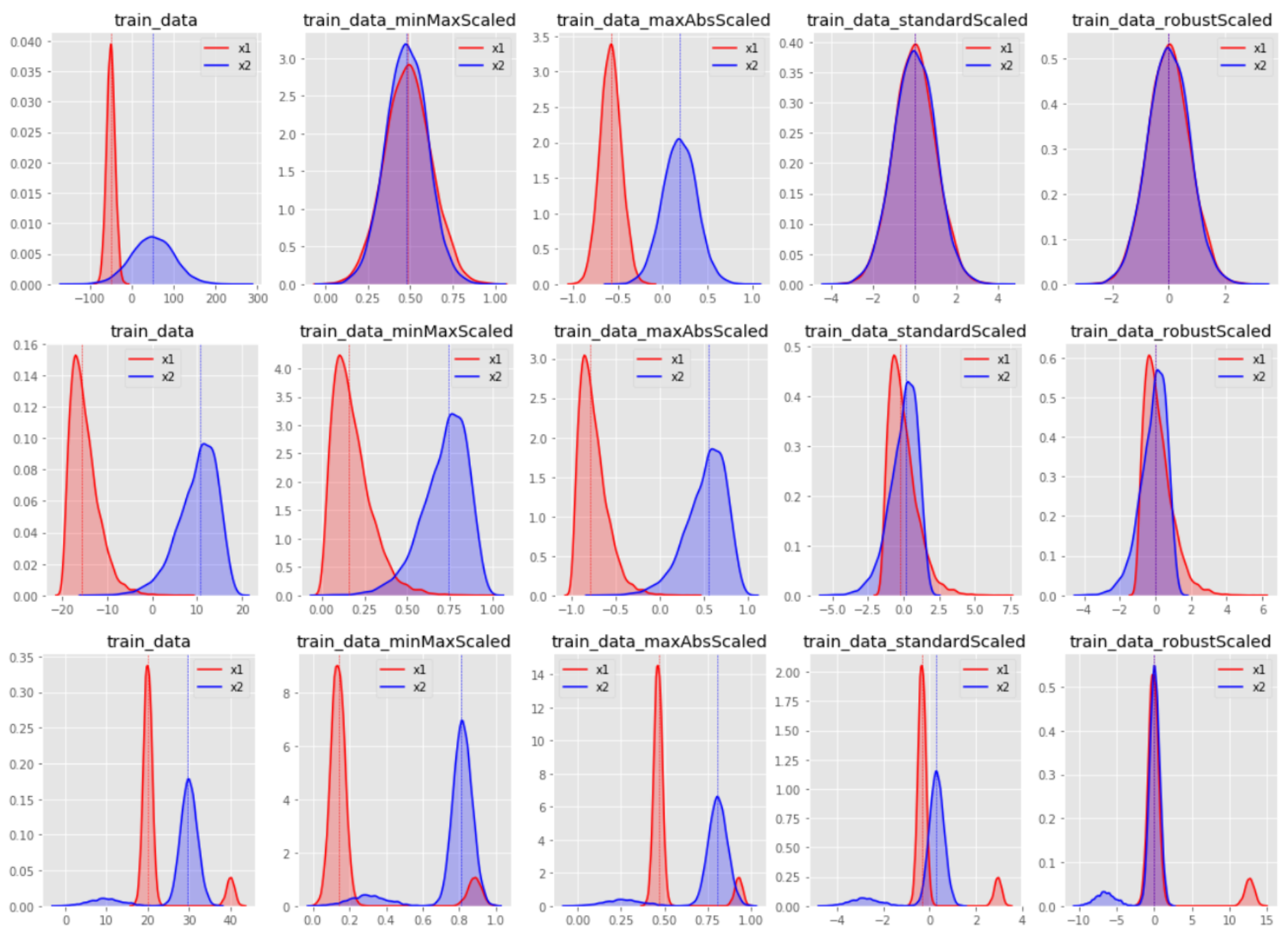
스케일링
1. 스케일링 절차 설명
- fit() -> transform() or fit_transform()
1) fit() : 주어진 데이터에 맞추어 학습
- 학습을 위한 기준 값을 설정 (ex.최소값,최대값 등)
2) transform(): 스케일러 적용, fit()된 정보를 이용해 데이터를 변환
3) fit_transform() : fit()과 transform()을 한 번에 실행
2. 스케일링(표준화(Standardization))
1) 표준화의 특징
- 서로 다른 통계 데이터들을 비교(독립변수간 비교)에 용이함
- 평균은 0, 분산과 표준편차는 1이 되므로 데이터의 분포가 단순화
되기 때문 - 이상치에 민감하며, 분류보다는 회귀에 유용
2) 코드
- 표준화 할 데이터 가져오기
import pandas as pd
import seaborn as sns
# Pandas 소수점 4째자리 이하에서 반올림
pd.set_option('display.float_format', lambda x: f'{x:.4f}')
# iris 데이터 로드
iris = sns.load_dataset('iris')
# iris의 수치형 변수만 선택
iris = iris.select_dtypes(exclude='object')
# sepal_lengh와 petal_length의 jointplot을 그림(데이터 확인차)
sns.jointplot(data=iris, x='petal_length', y='petal_width', kind='reg')- 표준화 하기
# 필요 패키지(클래스 / 함수) 가져오기
from sklearn.preprocessing import StandardScaler, RobustScaler
# Scaler 객체 생성
standard_scaler = StandardScaler()
robust_scaler = RobustScaler()
# 데이터 변환
iris_standard = pd.DataFrame(standard_scaler.fit_transform(iris), columns=iris.columns)
iris_robust = pd.DataFrame(robust_scaler.fit_transform(iris), columns=iris.columns)
# 결과 출력
# 평균 = 0, 표준편차 = 1
print('Standard Scaled: \n', iris_standard.describe())
print()
# 중위값 = 0, IQR = 1
print('Robust Scaled: \n', iris_robust.describe())- 표준화 된 결과를 시각화 해서 확인
# 그래프로 확인
import seaborn as sns
import patchworklib as pw
pw.overwrite_axisgrid()
# 첫번째 그래프
g1 = sns.jointplot(data=iris_standard, x='petal_length', y='petal_width', kind='reg')
g1 = pw.load_seaborngrid(g1)
g1.set_suptitle("Standard Scaled")
# 두번째 그래프
g2 = sns.jointplot(data=iris_robust, x='petal_length', y='petal_width', kind='reg')
g2 = pw.load_seaborngrid(g2)
g2.set_suptitle("Robust Scaled")
# 그래프 합쳐서 보기
g12 = (g1|g2)
g123. 스케일링(정규화(Normalization))
1) 사용할 함수별 특징
- MinMaxScaler(): 범위가 0~1이 되도록 스케일링
- MaxAbsScaler()
/- 모든 값이 양수이면, 범위가 0~1이 되도록 스케일링
/- 모든 값이 음수이면, 범위가 -1~0이 되도록 스케일링
/- 양수와 음수가 혼재하면, 범위가 -1~1이 되도록 스케일링
2) 코드
- 정규화 하기
# 필요 패키지(클래스 / 함수) 가져오기
from sklearn.preprocessing import MinMaxScaler, MaxAbsScaler
# Scaler 객체 생성
minmax_scaler = MinMaxScaler()
maxabs_scaler = MaxAbsScaler()
# 데이터 변환
iris_minmax = pd.DataFrame(minmax_scaler.fit_transform(iris), columns=iris.columns)
iris_maxabs = pd.DataFrame(maxabs_scaler.fit_transform(iris), columns=iris.columns)
# 결과 출력
# min = 0, max = 1
print('MinMax Scaled: \n', iris_minmax.describe())
print()
# min = -1 ~ 0, max = 1
print('MaxAbs Scaled: \n', iris_maxabs.describe())- 정규화 된 결과를 시각화 해서 확인
# 세번째 그래프(위에서 g1,g2저장했으므로)
g3 = sns.jointplot(data=iris_minmax, x='petal_length', y='petal_width', kind='reg')
g3 = pw.load_seaborngrid(g3)
g3.set_suptitle("MinMax Scaled")
# 네번째 그래프
g4 = sns.jointplot(data=iris_maxabs, x='petal_length', y='petal_width', kind='reg')
g4 = pw.load_seaborngrid(g4)
g4.set_suptitle("MaxAbs Scaled")
# 그래프 합치기
g34 = (g3|g4)
g34스케일링(변환(Transformation))
1) 사용할 함수 별 특징
- PowerTransformer(): 정규성 변환
- QuantileTransformer(): 균일분포 또는 정규분포로 변환
- Normalizer(): 한 행의 모든 피처들 사이의 유클리드 거리가 1이 되도
록 변환
2) 코드
- 변환 하기
# 필요 패키지(클래스 / 함수) 가져오기
import numpy as np
from sklearn.preprocessing import PowerTransformer, Normalizer
from sklearn.preprocessing import QuantileTransformer
# Scaler 객체 생성
power_scaler = PowerTransformer()
normal_scaler = Normalizer()
gaussian_scaler = QuantileTransformer(output_distribution='normal')
uniform_scaler = QuantileTransformer(output_distribution='uniform')
# 데이터 변환
iris_power = pd.DataFrame(power_scaler.fit_transform(iris), columns=iris.columns)
iris_normal = pd.DataFrame(normal_scaler.fit_transform(iris), columns=iris.columns)
iris_gaussian = pd.DataFrame(gaussian_scaler.fit_transform(iris), columns=iris.columns)
iris_uniform = pd.DataFrame(uniform_scaler.fit_transform(iris), columns=iris.columns)
# 결과 출력
# 평균 = 0, 표준편차 = 1
print('PowerTranformer Scaled: \n', iris_power.describe())
print()
print('Normalizer Scaled: \n', iris_normal.describe())
print('Euclidian Distance from 0: \n',
print('QuantileTranformer_Gaussian Scaled: \n', iris_gaussian.describe())
print()
print('QuantileTranformer_Uniform Scaled: \n', iris_uniform.describe())
# +) 각 행의 벡터 크기가 1이 되는지 확인
np.linalg.norm(iris_normal, axis=1))- 변환된 결과를 시각화 해서 확인
# 다섯번째 그래프(위에서 g3,g4만들어 놓았으므로)
g5 = sns.jointplot(data=iris_power, x='petal_length', y='petal_width', kind='reg')
g5 = pw.load_seaborngrid(g5)
g5.set_suptitle("PowerTransformer Scaled")
# 여섯번째 그래프
g6 = sns.jointplot(data=iris_normal, x='petal_length', y='petal_width', kind='reg')
g6 = pw.load_seaborngrid(g6)
g6.set_suptitle("Normalizer Scaled")
# 일곱번째 그래프
g7 = sns.jointplot(data=iris_gaussian, x='petal_length', y='petal_width', kind='reg')
g7 = pw.load_seaborngrid(g7)
g7.set_suptitle("QuantileTranformer_Gaussian Scaled")
# 여덟번째 그래프
g8 = sns.jointplot(data=iris_uniform, x='petal_length', y='petal_width', kind='reg')
g8 = pw.load_seaborngrid(g8)
g8.set_suptitle("QuantileTranformer_Uniform Scaled")
# 그래프 합치기(g56 / g78)
g56 = (g5|g6)
g56
g78 = (g7|g8)
g78
# 모든그래프(g1~g8) 한번에 보기
(g1|g2|g3|g4)/(g5|g6|g7|g8)