1. 모델 아키텍처
ProteinGPT는 다중 모달 데이터를 처리하기 위해 설계된 모델로, 두 개의 고정된 사전 학습된 인코더와 선형 투영 레이어 및 대형 언어 모델(LLM)로 구성됩니다. 이 아키텍처는 다음과 같은 주요 구성 요소로 이루어져 있습니다:
-
단백질 서열 인코더:
단백질 서열을 인코딩하는 모델로, ESM-2(Evolutionary Scale Modeling 2)에서 파생된 esm2_t36_3B_UR50D 모델이 사용됩니다. 이 모델은 36개의 트랜스포머 레이어와 30억 개의 파라미터를 특징으로 하며, UniRef50/D 데이터베이스에서 학습되어 서열의 다양성을 높이는 데 최적화되었습니다. 이를 통해 단백질 서열의 진화 정보, 기능적 위치, 구조-서열 관계 등을 포착할 수 있습니다. -
단백질 구조 인코더:
단백질의 구조 정보를 인코딩하는 역접힘 모델로, esm_if1_gvp4_t16_142M_UR50 모델을 사용합니다. 이 모델은 AlphaFold2가 예측한 1,200만 개의 구조에서 학습된 역접힘 모델로, 단백질의 3D 구조와 아미노산 잔기 간의 상호작용을 포착하는 데 최적화되었습니다. -
투영 레이어:
서열 인코더와 구조 인코더에서 생성된 임베딩을 LLM 임베딩과 정렬하기 위해 선형 투영 레이어가 사용됩니다. 이 투영 레이어는 구조적 및 서열적 정보를 통합하여 LLM에 맞춰 단백질 표현을 조정합니다. -
대형 언어 모델(LLM):
LLM은 사용자가 단백질 서열과 구조 데이터를 입력하면, 자연어 질문에 대해 응답할 수 있는 텍스트 기반 응답을 생성합니다. 이 과정에서 LLM은 서열 및 구조 임베딩과 결합된 입력을 바탕으로 질문에 맞는 문맥적이고 정확한 답변을 생성합니다.
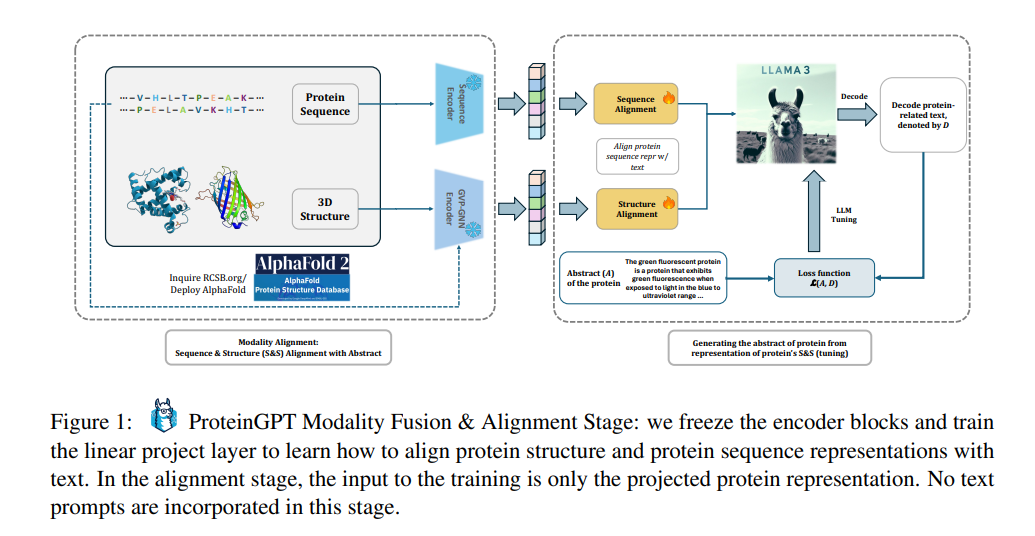
2. 훈련 방법
ProteinGPT는 두 단계로 나누어 훈련됩니다.
다음으로, 논문의 Methodology (방법론) 부분을 번역하였습니다.
2. 방법론
2.1 모델 아키텍처
ProteinGPT는 두 개의 고정된 사전 훈련된 인코더로 구성됩니다(그림 1 및 2 참조). 하나는 구조 인코딩을 위한 역접힘 모델(esm_if1_gvp4_t16_142M_UR50)이고, 다른 하나는 서열 인코딩을 위한 단백질 언어 모델(esm2_t36_3B_UR50D)입니다. 이 모델들이 생성한 임베딩은 선형 투영 레이어에 전달되어 LLM을 위한 소프트 프롬프트를 생성합니다. 모델 학습은 두 단계로 이루어집니다. 1) 서열 및 구조 정렬과 2) 지시 조정(Instruction Tuning)입니다.
2.1.1 서열 및 구조 정렬
정렬 단계에서는 단백질 구조가 먼저 사전 훈련된 구조 인코더에 전달되어 아미노산 잔기들 간의 공간 상호작용을 모델링하고 세밀한 3D 구조를 포착합니다. 우리는 구조 인코더로 esm_if1_gvp4_t16_142M_UR50를 사용합니다. 그 후, 서열은 36개의 트랜스포머 레이어와 30억 개의 매개변수를 특징으로 하는 esm2_t36_3B_UR50D 서열 인코더로 인코딩되며, 이 모델은 Protein UniRef50/D 데이터베이스에서 학습되어 서열의 다양성을 향상시킵니다. 이 모듈은 구조적 정보와 함께 구조적 접촉, 진화적 및 생화학적 정보를 통합합니다. 이러한 두 모듈은 효율적인 학습을 위해 고정됩니다. 우리는 단백질-텍스트 모달리티 정렬을 위해 특별한 토큰 프롬프트를 사용합니다:
질문(Q): \\\
\\
응답(A): \<Description>
구조적 및 서열적 정보는 소프트 프롬프트로 인코딩되어 질문 프롬프트 앞에 추가됩니다. 1단계 훈련에서는 추상적 설명을 단백질 표현에서 학습하는 데 우선순위를 두기 위해 질문 프롬프트 Q는 비어 있습니다. 설명 태그는 RCSB-PDB의 전체 주석으로 대체되어, 단백질과 해당 주석 설명을 정렬하는 투영 레이어를 학습시킵니다.
2.1.2 지시 조정(Instruction Tuning)
2단계에서는 우리가 마련한 QA 데이터셋을 사용하여 모델이 지시 조정 단계로 들어갑니다. 이전 연구들이 전체 주석을 사용했던 것과 달리, 우리는 구체적인 QA 예시를 사용하여 지시 조정을 촉진합니다. 우리는 1단계에서 얻은 추상적 데이터셋을 GPT-4o를 사용해 명시적인 QA 쌍으로 생성하여 이 단계에서 사용합니다. 1단계 프롬프트는 LLaMA 스타일로 적응되며("### Human : ..." 및 "### Assistant : ...") QA 데이터셋에서 명시적인 질문으로 Q가 대체됩니다. 예를 들어 "이 단백질은 몇 개의 어셈블리를 가지고 있나요?"와 같은 질문이 주어지면, 모델은 해당 단백질에 대한 설명적인 답변을 A로 생성합니다.
2.2 데이터셋
모달리티 정렬을 위해, 우리는 132,092개의 단백질 구조, 서열 및 추상적 설명으로 이루어진 RCSB-PDB 데이터베이스에서 대규모 데이터셋을 구축하였습니다. 원시 데이터셋에는 204,826개의 단백질이 포함되었지만, 추상적 설명, A 체인, 및 비코딩 문자가 없는 서열을 가진 단백질만을 유지하여 필터링하였습니다. 최종 데이터셋의 각 항목은 백본 원자 좌표로 표현된 3D 단백질 구조, 서열 문자열, 그리고 풍부한 단백질 주석을 포함하고 있습니다.
다음으로 Experiments (실험) 섹션으로 넘어갈까요?
3. 주요 실험 결과
3. 실험
3.1 훈련
훈련 과정은 두 단계로 나뉩니다: 모달리티 정렬(MA)과 지시 조정(IT)입니다. 이러한 접근 방식은 모델이 이전에 습득한 지식을 보존하면서 단백질 관련 질문에 대한 특정 지시를 효과적으로 처리할 수 있게 합니다.
단계 1: 모달리티 융합/정렬(MA)
이 단계에서는 서열 및 구조 인코더를 모두 고정하고 투영 어댑터의 훈련에 집중합니다. RCSB-PDB 데이터셋 내의 주석 길이를 고려하여, 추상 텍스트의 최대 길이를 384자로 설정합니다. 투영 레이어는 10 에포크 동안 배치 크기 1로 훈련되며, 가중치 감소는 0.05로 설정되고, 2,048개의 워밍업 스텝을 거칩니다. 데이터셋은 훈련 세트(70%, 105,673개의 단백질)와 테스트 세트(30%, 26,419개의 단백질)로 나뉩니다. 우리는 β1=0.9, β2=0.98로 설정된 AdamW 옵티마이저를 사용하고, 선형 워밍업 후 코사인 감소 스케줄러를 사용합니다. 초기 학습률은 1×10⁻⁴로 설정하고, 최소 학습률은 8×10⁻⁵이며, 워밍업 학습률은 1×10⁻⁶으로 설정합니다. 자동 혼합 정밀도(AMP)를 사용하여 훈련 효율성을 높였습니다.
단계 2: 지시 조정(IT)
이 단계에서는 단백질 질문-응답 작업에 대해 모델을 미세 조정합니다. 10 에포크 동안 배치 크기 1로 훈련하며, 가중치 감소는 0.05, 워밍업 스텝은 200개로 설정합니다. 이 단계에서 사용된 QA 데이터셋에는 약 370만 개의 샘플이 포함되며, 단백질당 약 35개의 질문이 주어집니다. 우리는 AdamW 옵티마이저와 AMP를 유사한 설정으로 사용하지만, 초기 학습률을 1×10⁻⁵로 낮추고, 최소 학습률은 1×10⁻⁶, 워밍업 학습률은 1×10⁻⁶으로 설정합니다.
3.2 계산 비용
훈련은 80GB의 NVIDIA H100 PCIe GPU 2대와 40GB의 NVIDIA A100 PCIe GPU 2대를 사용하여 진행되었습니다. 1단계 훈련에는 약 일주일이 소요되었고, 2단계는 약 60시간이 필요했습니다.
4. 결과
우리는 ProteinGPT를 4개의 기본 LLM 아키텍처(Vicuna, LLaMA-2, LLaMA-3, Mistral)에서 훈련하였습니다. 다양한 시나리오에서 ProteinGPT의 효과를 정량적, 정성적으로 평가하기 위한 일련의 실험을 진행하였습니다. 또한, 주요 모듈의 중요성을 파악하기 위해 제거 실험(ablations)을 수행하였습니다. 이는 원본 모델(단백질 서열을 텍스트 입력으로 사용한 LLM), 모달리티 정렬 모델, 지시 조정 모델(ProteinGPT)로 나뉩니다.
또한, OpenAI의 API를 기준으로 최첨단 대형 언어 모델의 단백질 관련 작업 능력을 벤치마킹하였습니다. 빈 모델 출력은 데이터 일관성을 위해 "N/A"로 대체하였습니다.
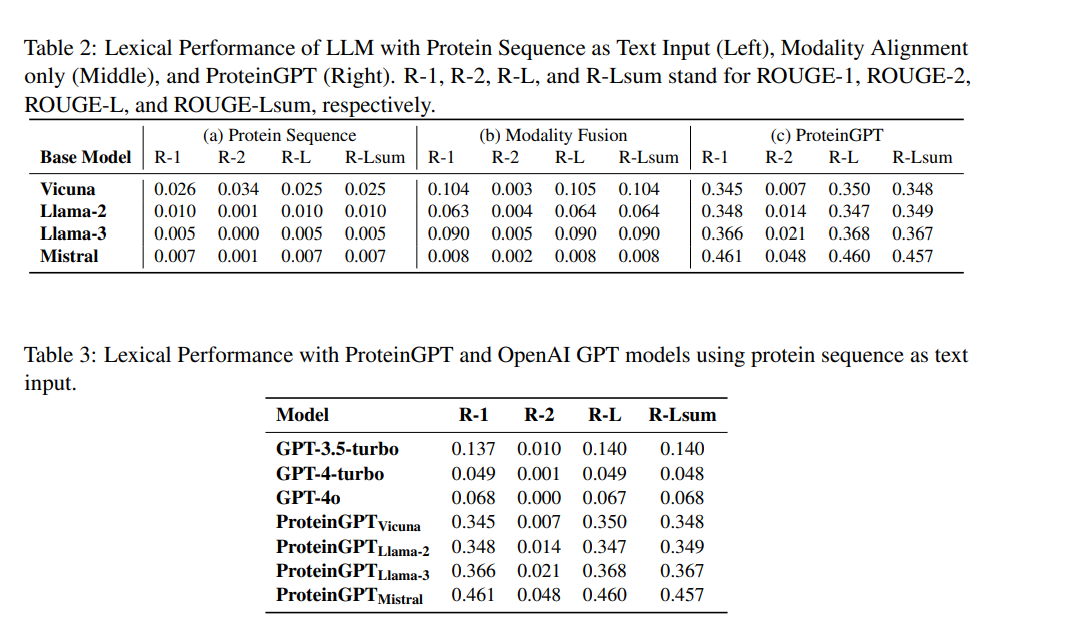
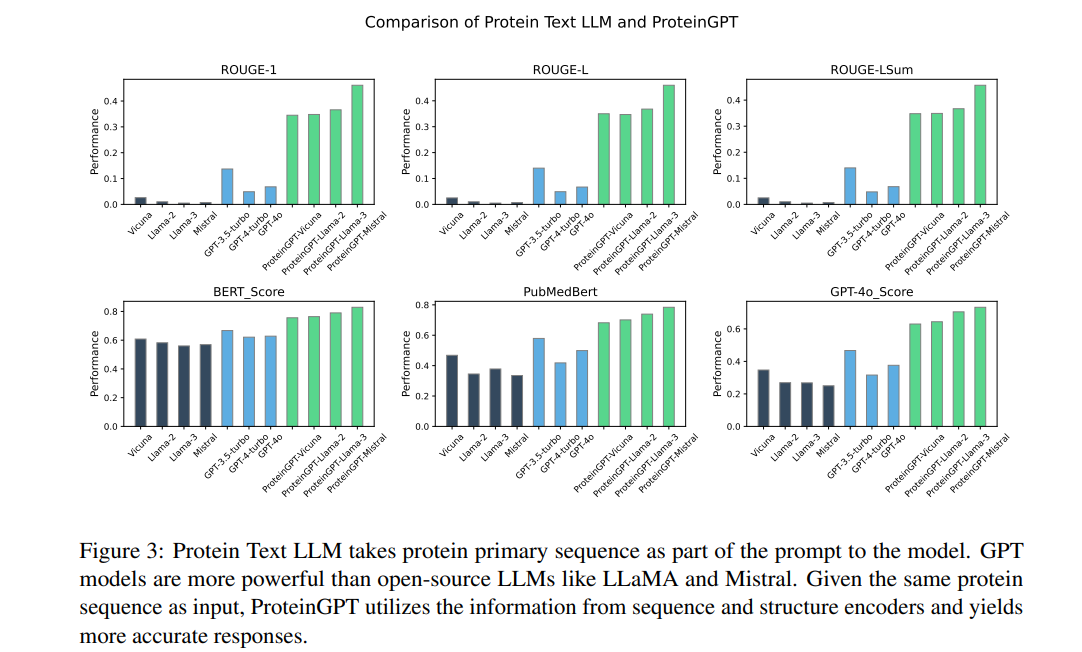
ProteinGPT의 성능을 평가하기 위해 다양한 실험이 수행되었습니다. 실험 결과는 정성적 및 정량적으로 평가되었으며, 기본 LLM과 다른 대형 언어 모델과의 비교도 진행되었습니다.
정성적 평가 (Qualitative Evaluation):
ProteinGPT는 인간 사용자의 질문에 대해 의미를 잘 해석하고, 논리적이고 정확한 답변을 제공하였습니다. 특히, 단백질의 기능(예: 질소를 암모니아로 환원하는 촉매 역할)과 구조적 세부 사항(예: 결합 부위의 구조적 의존성)에 대한 깊이 있는 응답을 제공하여, 단백질 구조와 서열의 이해 능력을 입증했습니다.
정량적 평가 (Quantitative Evaluation):
ProteinGPT는 다양한 정량적 지표를 사용하여 평가되었습니다. BERTScore, PubMedBERT-Score, GPT Score와 같은 의미 유사성 지표에서 매우 높은 점수를 기록하였으며, 특히 모달리티 정렬과 지시 조정 단계를 거친 ProteinGPT는 기존의 대형 언어 모델(GPT-4, GPT-3.5)과 비교하여 더 높은 성능을 발휘했습니다.
기본 LLM과 비교: ProteinGPT는 기존 LLM보다 단백질 서열 및 구조 데이터를 더 잘 이해하고 처리하여, 더 정확하고 의미 있는 답변을 제공하였습니다.
GPT-4와 비교: GPT-4 및 GPT-3.5-turbo와의 비교에서도 ProteinGPT는 일관되게 우수한 성능을 발휘했으며, 특히 다중 모달 정보를 활용하는 능력에서 더 뛰어난 성과를 보였습니다.
폐쇄형 질문 정확도:
폐쇄형 질문(예: "이 단백질에는 몇 개의 중합체가 있습니까?")에 대한 실험에서는 ProteinGPT가 80% 이상의 높은 정확도를 기록했습니다. 이는 단백질 구조 및 서열 정보에 대한 사실적 응답을 제공하는 능력을 입증하였으며, 특히 LLaMA-3와 Mistral 모델에서 가장 높은 성능을 보였습니다.
알파폴드랑 비교한게 없는데 그냥 단순한 질문-응답 방식의 Fine tuning이네요.
