요약
며칠전 구글 딥마인드에서 나온 따끈따끈한 단백질 설계 논문입니다.
일단 논문 자체에 인공지능 관련 지식은 많지 않고(사실상 전무하다고 봐도 됩니다.) AlphaProteo가 해당 분야에서 높은 성능을 낼 수 있음을 증명하는 논문이였습니다. 논문 내용도 실험 및 검증이 대부분이였고요. 그래서 그런지 모르겠는데 논문을 gpt로 쓴거 같다는 느낌도 드네요. 문장 구조라던가 번역본을 읽으면 gpt로 쓴게 바로 느껴집니다.
AlphaProteo는 자연에서 발견된 단백질을 기반으로 하지 않고, de novo(처음부터) 설계된 새로운 단백질을 생성하는 시스템입니다. 이는 특히 특정 표적 단백질과 결합하는 최적화된 단백질을 설계하는 데 초점을 맞추고 있습니다. 다시 말해, AlphaProteo는 미리 존재하는 자연 단백질을 사용하는 것이 아니라, 원하는 표적 단백질과 결합할 수 있도록 맞춤형으로 설계된 새로운 단백질을 생성합니다.
AlphaProteo의 주요 장점 중 하나는 표적 단백질의 특정 에피톱(결합 부위)에 맞춰 결합할 수 있는 단백질을 생성할 수 있다는 점입니다. 이를 통해 다음과 같은 것들이 가능합니다:
- 높은 결합 친화도: AlphaProteo는 특정 표적에 대해 나노몰(nM) 또는 서브 나노몰 수준의 매우 강력한 결합 친화도를 가진 단백질을 설계할 수 있습니다.
- 빠른 설계 및 검증: 몇 개의 설계 후보만으로도 실험적으로 유의미한 결합체를 얻을 수 있어, 고속 스크리닝이 필요 없습니다.
- 다양한 응용 가능성: AlphaProteo는 다양한 구조적 및 생화학적 특성을 가진 표적에 대해 적용할 수 있으며, 특정 질병 표적이나 생물학적 기능을 억제하거나 촉진하는 맞춤형 단백질 설계가 가능합니다.
즉, AlphaProteo는 원하는 단백질과 결합할 수 있는 최적화된 결합체를 설계하는 것이 가능하며, 이러한 결합체는 기존의 자연적 단백질이 아닌, AI 모델을 이용해 새롭게 만들어진 단백질입니다. 이는 신약 개발이나 단백질-단백질 상호작용 연구에서 매우 유용하게 사용될 수 있습니다.
Abstract
AlphaProteo는 de novo 단백질 결합체 설계 시스템으로, 특정 단백질 표적에 대한 맞춤형 결합 단백질을 생성하는 데 중점을 둡니다. AlphaProteo의 주요 기능은 다음과 같습니다:
1. 고성능 결합체 설계
AlphaProteo는 단백질-단백질 상호작용을 예측하고 설계하는 데 사용됩니다. 기존 자연 단백질을 이용하지 않고 컴퓨터 기반으로 새로운 결합체를 설계하여 특정 표적 단백질에 결합할 수 있는 단백질을 만듭니다. 이 시스템은 핫스팟이라고 불리는 표적 단백질의 특정 결합 부위를 선택적으로 겨냥하여 결합체를 설계할 수 있습니다.
2. 높은 성공률
AlphaProteo는 설계된 결합체의 성공률을 크게 높입니다. 실험적으로 테스트된 설계의 9%에서 88%의 성공률을 기록하며, 수십 개의 후보 중에서 가장 안정적이고 발현이 용이한 결합체를 선택할 수 있습니다.
3. 강력한 결합 친화도
AlphaProteo로 설계된 결합체는 나노몰(nM) 또는 서브 나노몰(sub-nM) 수준의 결합 친화도를 보여줍니다. 이는 매우 강력한 결합력을 의미하며, 일부 결합체는 임상적으로 중요한 단백질들과의 상호작용에서 높은 친화도를 나타냅니다.
4. 광범위한 응용 가능성
AlphaProteo는 다양한 구조적, 생화학적 특성을 가진 복잡한 표적 단백질에 대해 효과적으로 결합체를 설계할 수 있습니다. 이를 통해 암 치료, 감염병 대응 등 여러 분야에서 활용될 수 있는 새로운 결합체를 개발할 수 있습니다.
5. 심층 학습 기반 설계
AlphaProteo는 심층 학습 모델을 기반으로 결합체의 구조와 서열을 예측합니다. 이러한 모델은 단백질 구조 예측에 사용되는 최신 AI 기술인 AlphaFold와 통합되어 높은 정확도의 설계를 가능하게 합니다.
6. 자동화된 필터링 및 최적화
생성된 수많은 설계 후보 중에서 자동 필터링 과정을 통해 높은 가능성을 가진 결합체만을 선택할 수 있습니다. 이러한 자동화 과정은 실험적 비용을 절감하고 설계 효율을 극대화합니다.
1. 서론
서론
실험적 선택은 표적 에피톱을 제어할 수 없으며, 이는 일상적인 연구 응용에는 너무 많은 노력이 필요합니다. 컴퓨터 기반 de novo 결합체 설계는 자연 단백질을 출발점으로 사용하지 않고도 미리 지정된 에피톱을 표적으로 하여 항체보다 더 작고, 열 안정성이 높으며 발현이 더 쉬운 결합체를 생성할 수 있습니다.
이 기술 보고서는 AlphaProteo 단백질 설계 시스템을 소개하고 다음과 같은 장점으로 de novo 단백질 결합 단백질을 설계할 수 있음을 보여줍니다.
- 높은 성공률: 수십 개의 설계 후보를 스크리닝하여 안정적이고, 고도로 발현되며 특정한 결합체를 얻을 수 있어 고속 스크리닝 방법이 필요하지 않음.
- 높은 친화력: 테스트한 모든 표적 중 하나를 제외하고는 최상의 결합체가 서브 나노몰 또는 저 나노몰 결합 친화도를 가짐.
- 범용성: 복잡한 수동 개입 없이 단일 설계 방법을 사용하여 다양한 구조적, 생화학적 특성을 가진 표적에 대해 성공적으로 결합체를 얻음.
즉 기존 단백질 구조결합을 예측하는 방법은 쌩 노가다였기 때문에 결합체 그 자체를 생성하는 새로운 방법을 컴퓨터로 예측해서 하는것이 논문 내용의 핵심이라 볼 수 있을 것 같습니다.
2. 결과 (Results)
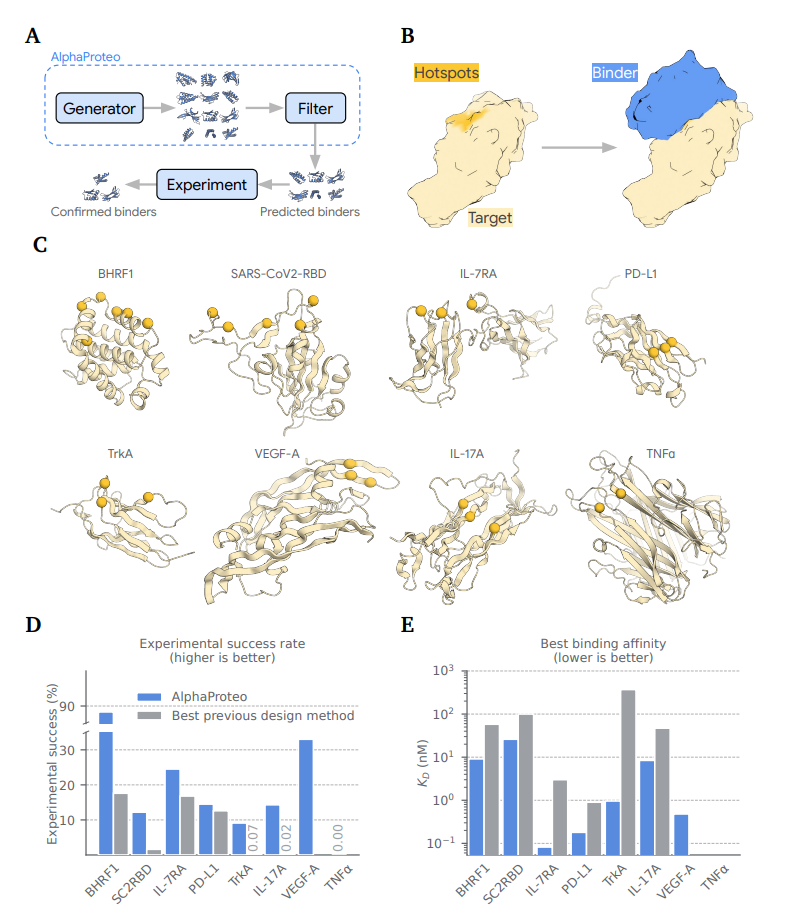
AlphaProteo는 두 가지 구성 요소로 이루어져 있습니다(Figure 1A). 첫 번째는 Protein Data Bank (PDB)의 구조 및 서열 데이터를 기반으로 훈련된 생성 모델이며, 두 번째는 AlphaFold 예측에서 추출한 디스틸레이션 집합입니다. 결합체를 설계하기 위해, '표적' 단백질의 구조를 입력하고 선택적으로 표적 에피톱을 나타내는 '핫스팟' 잔기를 지정할 수 있습니다. 생성 모델은 해당 표적에 대한 후보 결합체의 구조와 서열을 출력합니다(Figure 1B). AlphaProteo는 많은 수의 설계 후보를 생성한 후 실험적으로 테스트하기 전에 필터링을 통해 소수의 설계로 압축합니다. 생성 모델은 기존 최고의 방법들과 비교하여 in silico 벤치마크에서 우수한 성능을 보였습니다(Figure S1, Section S2).
2.1 중간 처리 스크리닝을 통한 서브 나노몰 친화력 결합체
AlphaProteo를 실험적으로 검증하기 위해, 연구진은 구조적 특성이 다양한 8개의 표적 단백질에 대해 결합체를 설계했습니다. 이 중 두 가지는 감염에 관련된 바이러스 단백질이고, 여섯 가지는 치료적으로 중요한 인간 단백질입니다(Figure 1C, Table S1).
- BHRF1: Epstein-Barr 바이러스의 발암 단백질로, 결합체를 통해 억제하면 암세포를 사멸시키고 종양 성장을 늦출 수 있습니다. 이 단백질은 결합 파트너의 헬릭스를 수용할 수 있는 완벽한 소수성 홈을 가지고 있어 결합을 촉진합니다.
- SARS-CoV-2 스파이크 단백질 수용체 결합 도메인(SC2RBD): COVID-19 감염에 필요한 단백질 도메인으로, 이 도메인과 인간 ACE2 수용체 간의 상호작용을 방해하면 SARS-CoV-2가 인간 세포를 감염시키는 것을 차단할 수 있습니다.
- 인터루킨-7 수용체 알파(IL-7RA): 림프구 발달에 중요한 세포 표면 수용체로, 급성 림프성 백혈병과 HIV 치료의 표적입니다.
- 프로그램 세포사멸 리간드 1(PD-L1): 면역 세포 증식을 조절하는 세포 표면 수용체로, 암 치료에서 중요한 표적입니다.
- 트로포미오신 수용체 키나아제 A(TrkA): 자가면역 질환과 만성 통증 치료에서 중요한 신경 성장 인자 수용체입니다.
- 인터루킨-17A (IL-17A): 염증을 유발하는 분비 단백질로, 자가면역 질환에서 중요한 치료 표적입니다.
- 혈관내피세포 성장인자 A (VEGF-A): 암과 당뇨성 망막병증에서 중요한 치료 표적입니다.
- 종양 괴사 인자 알파(TNFα): 염증 질환에서 중요한 프로염증성 사이토카인입니다.
연구진은 이러한 표적들을 생물학적 중요성, 설계 난이도의 범위, 그리고 기존 설계 방법들과의 비교를 위해 선택했습니다. 각 표적에 대해 수많은 in silico 설계를 생성했으며, 자동화된 필터링 절차를 통해 47개에서 172개의 후보 결합체를 테스트했습니다. 7개의 표적에 대한 초기 실험에서, AlphaProteo는 각 표적에서 9%에서 88%의 실험 성공률을 기록했으며, 이는 기존 방법보다 더 높은 성공률을 나타냈습니다.
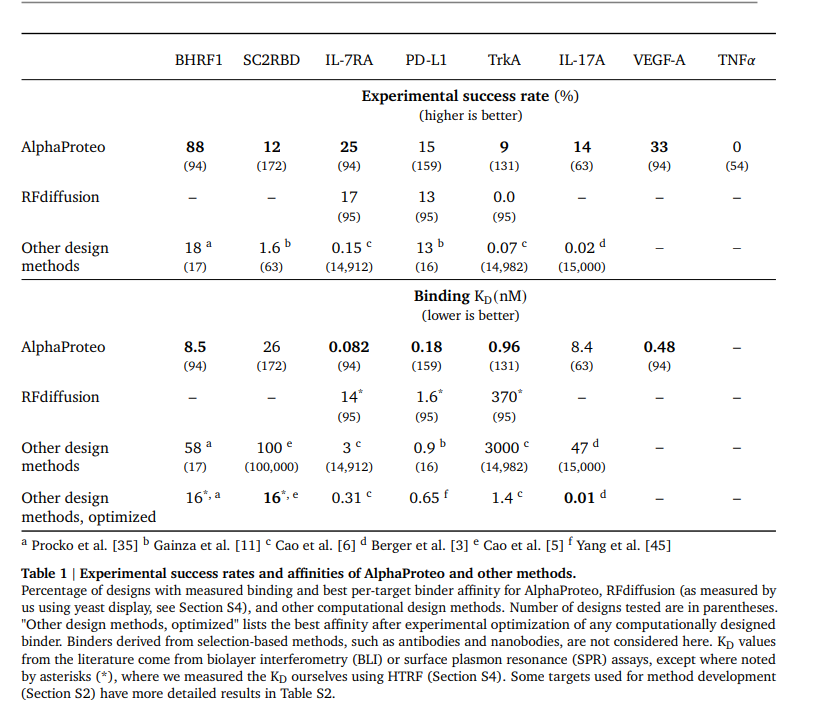
해당 성능 지표를 Table로 그린 것 입니다.
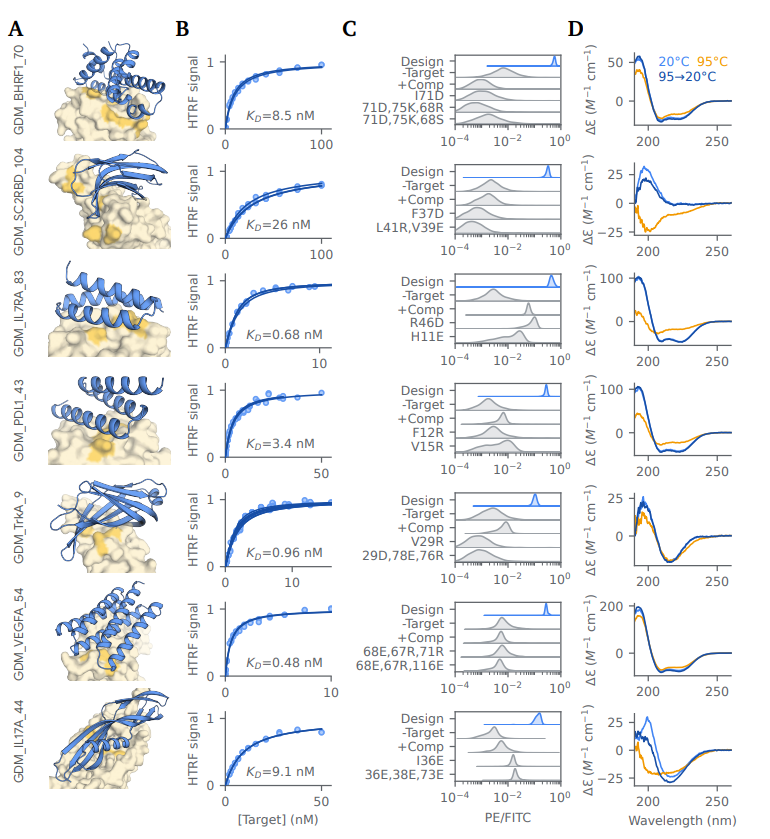
Figure S2 | In-silico 성공률의 분포
AlphaProteo의 결합체 설계를 위해 PDB에서 무작위로 선택된 200개의 표적 단백질에 대한 in-silico 성공률의 히스토그램(회색)과 누적 분포(주황색 선)입니다. 연구진이 실험적으로 결합체를 성공적으로 얻은 7개의 표적(파란색 점선)이 다양한 범위의 in-silico 성공률을 나타냅니다. TNFα는 매우 어려운 표적으로 평가되었고, IL-17A는 성공적으로 결합체를 얻은 표적 중 80%보다 어려운 표적입니다.

Figure S3 | 결합체 설계의 효모 발현 스크리닝
A: 결합 신호는 (Δ log(PE+) − Δ log(PE−))로 계산되며, 이는 결합 신호와 실험적 아티팩트를 제거한 신호의 차이를 나타냅니다.
B: FITC/PE 스캐터플롯에서 결합이 일어나지 않은 경우(왼쪽)와 결합이 약한 경우(오른쪽)의 예시입니다.
C: 각 표적에 대해 효모 표면 발현으로 테스트된 설계의 결합 신호 분포입니다. IL-17A의 경우 배경 결합 신호가 커서 임계값이 더 높게 설정되었습니다

Figure S4 | 결합체의 발현 수율 및 크기 배제 크로마토그래피
A: 효모 발현 스크리닝에서 결합 신호를 보인 결합체의 10 mL E. coli 배양에서 얻은 단백질 수율.
B: 결합체의 크기 배제 크로마토그래피(SEC) 결과. 대부분의 결합체가 단일 분산 형태로 나타났습니다.
